+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4jcj | ||||||
|---|---|---|---|---|---|---|---|
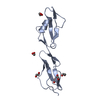
| タイトル | Crystal structure of Isl1 LIM domains with Ldb1 LIM-interaction domain | ||||||
 要素 要素 | Insulin gene enhancer protein ISL-1,LIM domain-binding protein 1 | ||||||
 キーワード キーワード |  PROTEIN BINDING (タンパク質) / PROTEIN BINDING (タンパク質) /  Zinc finger (ジンクフィンガー) / Zinc finger (ジンクフィンガー) /  Transcriptional complex (転写 (生物学)) / Transcriptional complex (転写 (生物学)) /  Zinc binding (亜鉛) / Zinc binding (亜鉛) /  Nucleus (Nucleus) Nucleus (Nucleus) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 sensory system development / positive regulation of macrophage colony-stimulating factor production / sinoatrial node cell development / trigeminal nerve development / sensory system development / positive regulation of macrophage colony-stimulating factor production / sinoatrial node cell development / trigeminal nerve development /  peripheral nervous system neuron axonogenesis / visceral motor neuron differentiation / positive regulation of granulocyte colony-stimulating factor production / spinal cord motor neuron cell fate specification / peripheral nervous system neuron axonogenesis / visceral motor neuron differentiation / positive regulation of granulocyte colony-stimulating factor production / spinal cord motor neuron cell fate specification /  regulation of kinase activity / regulation of kinase activity /  peripheral nervous system neuron development ... peripheral nervous system neuron development ... sensory system development / positive regulation of macrophage colony-stimulating factor production / sinoatrial node cell development / trigeminal nerve development / sensory system development / positive regulation of macrophage colony-stimulating factor production / sinoatrial node cell development / trigeminal nerve development /  peripheral nervous system neuron axonogenesis / visceral motor neuron differentiation / positive regulation of granulocyte colony-stimulating factor production / spinal cord motor neuron cell fate specification / peripheral nervous system neuron axonogenesis / visceral motor neuron differentiation / positive regulation of granulocyte colony-stimulating factor production / spinal cord motor neuron cell fate specification /  regulation of kinase activity / regulation of kinase activity /  peripheral nervous system neuron development / cellular component assembly / secondary heart field specification / negative regulation of erythrocyte differentiation / cardiac cell fate determination / negative regulation of mesenchymal cell proliferation / cardiac right ventricle morphogenesis / positive regulation of hemoglobin biosynthetic process / cerebellar Purkinje cell differentiation / regulation of secondary heart field cardioblast proliferation / epithelial structure maintenance / neuron fate specification / head development / atrial septum morphogenesis / primitive erythrocyte differentiation / positive regulation of type B pancreatic cell apoptotic process / transcription-dependent tethering of RNA polymerase II gene DNA at nuclear periphery / bHLH transcription factor binding / spinal cord motor neuron differentiation / positive regulation of interleukin-1 alpha production / beta-catenin-TCF complex / pituitary gland development / negative regulation of intracellular estrogen receptor signaling pathway / RUNX1 regulates transcription of genes involved in differentiation of HSCs / peripheral nervous system neuron development / cellular component assembly / secondary heart field specification / negative regulation of erythrocyte differentiation / cardiac cell fate determination / negative regulation of mesenchymal cell proliferation / cardiac right ventricle morphogenesis / positive regulation of hemoglobin biosynthetic process / cerebellar Purkinje cell differentiation / regulation of secondary heart field cardioblast proliferation / epithelial structure maintenance / neuron fate specification / head development / atrial septum morphogenesis / primitive erythrocyte differentiation / positive regulation of type B pancreatic cell apoptotic process / transcription-dependent tethering of RNA polymerase II gene DNA at nuclear periphery / bHLH transcription factor binding / spinal cord motor neuron differentiation / positive regulation of interleukin-1 alpha production / beta-catenin-TCF complex / pituitary gland development / negative regulation of intracellular estrogen receptor signaling pathway / RUNX1 regulates transcription of genes involved in differentiation of HSCs /  LIM domain binding / mesenchymal cell differentiation / cardiac muscle cell myoblast differentiation / neuron fate commitment / pharyngeal system development / LIM domain binding / mesenchymal cell differentiation / cardiac muscle cell myoblast differentiation / neuron fate commitment / pharyngeal system development /  神経 / outflow tract septum morphogenesis / positive regulation of smoothened signaling pathway / gastrulation with mouth forming second / 神経 / outflow tract septum morphogenesis / positive regulation of smoothened signaling pathway / gastrulation with mouth forming second /  膵臓 / retinal ganglion cell axon guidance / positive regulation of granulocyte macrophage colony-stimulating factor production / 膵臓 / retinal ganglion cell axon guidance / positive regulation of granulocyte macrophage colony-stimulating factor production /  神経堤 / anterior/posterior axis specification / endocardial cushion morphogenesis / ventricular cardiac muscle tissue morphogenesis / 神経堤 / anterior/posterior axis specification / endocardial cushion morphogenesis / ventricular cardiac muscle tissue morphogenesis /  regulation of focal adhesion assembly / positive regulation of calcium ion import / outflow tract morphogenesis / cell leading edge / regulation of heart rate by cardiac conduction / somatic stem cell population maintenance / negative regulation of neuron differentiation / hair follicle development / positive regulation of cell adhesion / canonical Wnt signaling pathway / positive regulation of vascular endothelial growth factor production / positive regulation of epithelial to mesenchymal transition / negative regulation of protein-containing complex assembly / cis-regulatory region sequence-specific DNA binding / heart morphogenesis / core promoter sequence-specific DNA binding / regulation of focal adhesion assembly / positive regulation of calcium ion import / outflow tract morphogenesis / cell leading edge / regulation of heart rate by cardiac conduction / somatic stem cell population maintenance / negative regulation of neuron differentiation / hair follicle development / positive regulation of cell adhesion / canonical Wnt signaling pathway / positive regulation of vascular endothelial growth factor production / positive regulation of epithelial to mesenchymal transition / negative regulation of protein-containing complex assembly / cis-regulatory region sequence-specific DNA binding / heart morphogenesis / core promoter sequence-specific DNA binding /  regulation of cell migration / positive regulation of tyrosine phosphorylation of STAT protein / positive regulation of interleukin-12 production / cerebellum development / regulation of cell migration / positive regulation of tyrosine phosphorylation of STAT protein / positive regulation of interleukin-12 production / cerebellum development /  軸索誘導 / positive regulation of interleukin-1 beta production / promoter-specific chromatin binding / 軸索誘導 / positive regulation of interleukin-1 beta production / promoter-specific chromatin binding /  細胞分化 / nuclear estrogen receptor binding / 細胞分化 / nuclear estrogen receptor binding /  nuclear receptor binding / positive regulation of cell differentiation / positive regulation of transcription elongation by RNA polymerase II / negative regulation of canonical Wnt signaling pathway / positive regulation of insulin secretion / neuron differentiation / nuclear receptor binding / positive regulation of cell differentiation / positive regulation of transcription elongation by RNA polymerase II / negative regulation of canonical Wnt signaling pathway / positive regulation of insulin secretion / neuron differentiation /  Wntシグナル経路 / negative regulation of inflammatory response / positive regulation of interleukin-6 production / RNA polymerase II transcription regulator complex / positive regulation of angiogenesis / Wntシグナル経路 / negative regulation of inflammatory response / positive regulation of interleukin-6 production / RNA polymerase II transcription regulator complex / positive regulation of angiogenesis /  : / negative regulation of epithelial cell proliferation / positive regulation of type II interferon production / sequence-specific double-stranded DNA binding / positive regulation of tumor necrosis factor production / positive regulation of peptidyl-serine phosphorylation / : / negative regulation of epithelial cell proliferation / positive regulation of type II interferon production / sequence-specific double-stranded DNA binding / positive regulation of tumor necrosis factor production / positive regulation of peptidyl-serine phosphorylation /  nervous system development / nervous system development /  heart development / heart development /  遺伝子発現の調節 / DNA-binding transcription activator activity, RNA polymerase II-specific / DNA-binding transcription factor binding / cell population proliferation / negative regulation of neuron apoptotic process / 遺伝子発現の調節 / DNA-binding transcription activator activity, RNA polymerase II-specific / DNA-binding transcription factor binding / cell population proliferation / negative regulation of neuron apoptotic process /  transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding transcription regulator complex / RNA polymerase II-specific DNA-binding transcription factor binding類似検索 - 分子機能 | ||||||
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 3 Å 単波長異常分散 / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Gadd, M.S. / Jacques, D.A. / Guss, J.M. / Matthews, J.M. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2013 ジャーナル: J.Biol.Chem. / 年: 2013タイトル: A structural basis for the regulation of the LIM-homeodomain protein islet 1 (Isl1) by intra- and intermolecular interactions. 著者: Gadd, M.S. / Jacques, D.A. / Nisevic, I. / Craig, V.J. / Kwan, A.H. / Guss, J.M. / Matthews, J.M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4jcj.cif.gz 4jcj.cif.gz | 172.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4jcj.ent.gz pdb4jcj.ent.gz | 142 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4jcj.json.gz 4jcj.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jc/4jcj https://data.pdbj.org/pub/pdb/validation_reports/jc/4jcj ftp://data.pdbj.org/pub/pdb/validation_reports/jc/4jcj ftp://data.pdbj.org/pub/pdb/validation_reports/jc/4jcj | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 19354.105 Da / 分子数: 3 / 断片: UNP RESIDUES 11-138 and unp residues 300-330 / 由来タイプ: 組換発現 / 由来: (組換発現)   Mus musculus (ハツカネズミ) / 遺伝子: Isl1, Ldb1, Nli / プラスミド: pGEX2T / 発現宿主: Mus musculus (ハツカネズミ) / 遺伝子: Isl1, Ldb1, Nli / プラスミド: pGEX2T / 発現宿主:   Escherichia coli (大腸菌) / 株 (発現宿主): BL21 (DE3) / 参照: UniProt: P61372, UniProt: P70662 Escherichia coli (大腸菌) / 株 (発現宿主): BL21 (DE3) / 参照: UniProt: P61372, UniProt: P70662#2: 化合物 | ChemComp-ZN / 配列の詳細 | THE CRYSTALLIZED SEQUENCE CORRESPONDS TO THE ISOFORM 3 OF LIM DOMAIN-BINDING PROTEIN 1.DATABASE ...THE CRYSTALLIZ | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.51 Å3/Da / 溶媒含有率: 50.93 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.9 詳細: 20% (w/v) PEG 8000, 0.2 M magnesium chloride, 0.1 M Tris, 0.3 mM zinc sulfate, pH 8.9, VAPOR DIFFUSION, HANGING DROP, temperature 298K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Australian Synchrotron Australian Synchrotron  / ビームライン: MX2 / 波長: 1.2816 Å / ビームライン: MX2 / 波長: 1.2816 Å |
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2011年12月6日 |
| 放射 | モノクロメーター: Zinc and platinum foils / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.2816 Å / 相対比: 1 : 1.2816 Å / 相対比: 1 |
| 反射 | 解像度: 3→50 Å / Num. all: 12362 / Num. obs: 12362 / % possible obs: 100 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 6.9 % / Biso Wilson estimate: 59.6 Å2 / Rmerge(I) obs: 0.16 / Net I/σ(I): 8.5 |
| 反射 シェル | 解像度: 3→3.05 Å / 冗長度: 7 % / Rmerge(I) obs: 0.66 / Mean I/σ(I) obs: 2.8 / Num. unique all: 584 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  単波長異常分散 / 解像度: 3→48.29 Å / Cor.coef. Fo:Fc: 0.908 / Cor.coef. Fo:Fc free: 0.835 / Occupancy max: 1 / Occupancy min: 1 / SU B: 41.993 / SU ML: 0.401 / Isotropic thermal model: Isotropic with TLS / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.471 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 単波長異常分散 / 解像度: 3→48.29 Å / Cor.coef. Fo:Fc: 0.908 / Cor.coef. Fo:Fc free: 0.835 / Occupancy max: 1 / Occupancy min: 1 / SU B: 41.993 / SU ML: 0.401 / Isotropic thermal model: Isotropic with TLS / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R Free: 0.471 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS U VALUES : WITH TLS ADDED
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 161.53 Å2 / Biso mean: 57.864 Å2 / Biso min: 20.62 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3→48.29 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.995→3.073 Å / Total num. of bins used: 20
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj




