+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4iq2 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | P21 crystal form of FKBP12.6 | ||||||
 要素 要素 | Peptidyl-prolyl cis-trans isomerase FKBP1B | ||||||
 キーワード キーワード |  ISOMERASE (異性化酵素) / ISOMERASE (異性化酵素) /  FKBP12.6 FKBP12.6 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of sequestering of calcium ion /  cyclic nucleotide binding / negative regulation of release of sequestered calcium ion into cytosol / negative regulation of insulin secretion involved in cellular response to glucose stimulus / neuronal action potential propagation / insulin secretion involved in cellular response to glucose stimulus / cell communication by electrical coupling involved in cardiac conduction / response to redox state / protein maturation by protein folding / 'de novo' protein folding ...positive regulation of sequestering of calcium ion / cyclic nucleotide binding / negative regulation of release of sequestered calcium ion into cytosol / negative regulation of insulin secretion involved in cellular response to glucose stimulus / neuronal action potential propagation / insulin secretion involved in cellular response to glucose stimulus / cell communication by electrical coupling involved in cardiac conduction / response to redox state / protein maturation by protein folding / 'de novo' protein folding ...positive regulation of sequestering of calcium ion /  cyclic nucleotide binding / negative regulation of release of sequestered calcium ion into cytosol / negative regulation of insulin secretion involved in cellular response to glucose stimulus / neuronal action potential propagation / insulin secretion involved in cellular response to glucose stimulus / cell communication by electrical coupling involved in cardiac conduction / response to redox state / protein maturation by protein folding / 'de novo' protein folding / negative regulation of heart rate / negative regulation of phosphoprotein phosphatase activity / cyclic nucleotide binding / negative regulation of release of sequestered calcium ion into cytosol / negative regulation of insulin secretion involved in cellular response to glucose stimulus / neuronal action potential propagation / insulin secretion involved in cellular response to glucose stimulus / cell communication by electrical coupling involved in cardiac conduction / response to redox state / protein maturation by protein folding / 'de novo' protein folding / negative regulation of heart rate / negative regulation of phosphoprotein phosphatase activity /  FK506 binding / positive regulation of axon regeneration / : / smooth muscle contraction / negative regulation of ryanodine-sensitive calcium-release channel activity / response to vitamin E / calcium channel inhibitor activity / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion / protein peptidyl-prolyl isomerization / T cell proliferation / regulation of release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / release of sequestered calcium ion into cytosol / regulation of ryanodine-sensitive calcium-release channel activity / Ion homeostasis / sarcoplasmic reticulum membrane / FK506 binding / positive regulation of axon regeneration / : / smooth muscle contraction / negative regulation of ryanodine-sensitive calcium-release channel activity / response to vitamin E / calcium channel inhibitor activity / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion / protein peptidyl-prolyl isomerization / T cell proliferation / regulation of release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / release of sequestered calcium ion into cytosol / regulation of ryanodine-sensitive calcium-release channel activity / Ion homeostasis / sarcoplasmic reticulum membrane /  calcium channel complex / regulation of cytosolic calcium ion concentration / calcium channel complex / regulation of cytosolic calcium ion concentration /  プロリルイソメラーゼ / プロリルイソメラーゼ /  peptidyl-prolyl cis-trans isomerase activity / response to hydrogen peroxide / Stimuli-sensing channels / Z disc / positive regulation of cytosolic calcium ion concentration / protein refolding / transmembrane transporter binding / peptidyl-prolyl cis-trans isomerase activity / response to hydrogen peroxide / Stimuli-sensing channels / Z disc / positive regulation of cytosolic calcium ion concentration / protein refolding / transmembrane transporter binding /  signaling receptor binding / signaling receptor binding /  生体膜 / 生体膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å | ||||||
 データ登録者 データ登録者 | Chen, H. / Mustafi, S.M. / Li, H.M. / LeMaster, D.M. / Hernandez, G. | ||||||
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2014 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2014タイトル: Crystal structure and conformational flexibility of the unligated FK506-binding protein FKBP12.6. 著者: Chen, H. / Mustafi, S.M. / LeMaster, D.M. / Li, Z. / Heroux, A. / Li, H. / Hernandez, G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
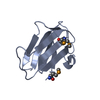
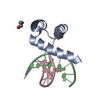
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4iq2.cif.gz 4iq2.cif.gz | 58.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4iq2.ent.gz pdb4iq2.ent.gz | 42.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4iq2.json.gz 4iq2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/iq/4iq2 https://data.pdbj.org/pub/pdb/validation_reports/iq/4iq2 ftp://data.pdbj.org/pub/pdb/validation_reports/iq/4iq2 ftp://data.pdbj.org/pub/pdb/validation_reports/iq/4iq2 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
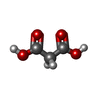
| #1: タンパク質 | 分子量: 11673.307 Da / 分子数: 2 / 変異: C22V, C76I / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: FKBP1B, FKBP12.6, FKBP1L, FKBP9, OTK4 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: FKBP1B, FKBP12.6, FKBP1L, FKBP9, OTK4 / 発現宿主:   Escherichia coli (大腸菌) / 参照: UniProt: P68106, Escherichia coli (大腸菌) / 参照: UniProt: P68106,  プロリルイソメラーゼ プロリルイソメラーゼ#2: 化合物 | ChemComp-MLA / |  マロン酸 マロン酸#3: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.06 Å3/Da / 溶媒含有率: 40.29 % |
|---|---|
結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7 詳細: 2.0 M malonic acid, pH 7.0, 4% isopropanol, VAPOR DIFFUSION, HANGING DROP, temperature 298K |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: Cu FINE FOCUS / 波長: 1.5418 Å 回転陽極 / タイプ: Cu FINE FOCUS / 波長: 1.5418 Å |
| 検出器 | タイプ: RIGAKU RAXIS IV++ / 検出器: IMAGE PLATE / 日付: 2012年6月2日 |
| 放射 | モノクロメーター: mirror / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.7→38.232 Å / Num. all: 20138 / Num. obs: 20138 / % possible obs: 95.8 % / Observed criterion σ(F): 1 / Observed criterion σ(I): 1 / 冗長度: 3.5 % / Rmerge(I) obs: 0.137 / Rsym value: 0.137 / Net I/σ(I): 4.6 |
| 反射 シェル | 解像度: 1.7→1.76 Å / 冗長度: 2.7 % / Rmerge(I) obs: 0.51 / Mean I/σ(I) obs: 1.4 / Num. unique all: 1549 / Rsym value: 0.51 / % possible all: 77.7 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1C9H 解像度: 1.7→38.232 Å / SU ML: 0.33 / 交差検証法: THROUGHOUT / σ(F): 1.34 / 位相誤差: 30.36 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.73 Å / VDWプローブ半径: 1 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL / Bsol: 46.081 Å2 / ksol: 0.419 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.7→38.232 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj