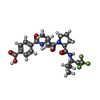
登録情報 データベース : PDB / ID : 3hgnタイトル Structure of porcine pancreatic elastase complexed with a potent peptidyl inhibitor FR130180 determined by neutron crystallography Elastase-1 キーワード / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Sus scrofa (ブタ)手法 / / / / 解像度 : 1.65 Å データ登録者 Tamada, T. / Kinoshita, T. / Kuroki, R. / Tada, T. 履歴 登録 2009年5月14日 登録サイト / 処理サイト 改定 1.0 2009年7月28日 Provider / タイプ 改定 1.1 2011年7月13日 Group / Non-polymer description / Version format compliance改定 1.2 2023年11月1日 Group Data collection / Database references ... Data collection / Database references / Derived calculations / Refinement description カテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_leaving_atom_flag / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 エラスターゼ
エラスターゼ  キーワード
キーワード HYDROLASE (加水分解酵素) /
HYDROLASE (加水分解酵素) /  Chymotrypsin Family (キモトリプシン) /
Chymotrypsin Family (キモトリプシン) /  Serine Protease (セリンプロテアーゼ) /
Serine Protease (セリンプロテアーゼ) /  Disulfide bond (ジスルフィド) / Metal-binding /
Disulfide bond (ジスルフィド) / Metal-binding /  Protease (プロテアーゼ) /
Protease (プロテアーゼ) /  Secreted (分泌) /
Secreted (分泌) /  Zymogen (酵素前駆体)
Zymogen (酵素前駆体) 機能・相同性情報
機能・相同性情報 pancreatic elastase / serine-type endopeptidase activity /
pancreatic elastase / serine-type endopeptidase activity /  タンパク質分解 /
タンパク質分解 /  extracellular space /
extracellular space /  metal ion binding
metal ion binding
 Sus scrofa (ブタ)
Sus scrofa (ブタ) 中性子回折 /
中性子回折 /  X線回折 / NUCLEAR REACTOR /
X線回折 / NUCLEAR REACTOR /  シンクロトロン / 解像度: 1.65 Å
シンクロトロン / 解像度: 1.65 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Am.Chem.Soc. / 年: 2009
ジャーナル: J.Am.Chem.Soc. / 年: 2009 ジャーナル: Acta Crystallogr.,Sect.F / 年: 2007
ジャーナル: Acta Crystallogr.,Sect.F / 年: 2007 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3hgn.cif.gz
3hgn.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3hgn.ent.gz
pdb3hgn.ent.gz PDB形式
PDB形式 3hgn.json.gz
3hgn.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/hg/3hgn
https://data.pdbj.org/pub/pdb/validation_reports/hg/3hgn ftp://data.pdbj.org/pub/pdb/validation_reports/hg/3hgn
ftp://data.pdbj.org/pub/pdb/validation_reports/hg/3hgn リンク
リンク 集合体
集合体
 要素
要素 エラスターゼ
エラスターゼ
 Sus scrofa (ブタ) / 参照: UniProt: P00772,
Sus scrofa (ブタ) / 参照: UniProt: P00772,  pancreatic elastase
pancreatic elastase 硫酸塩
硫酸塩 重水
重水 試料調製
試料調製
 解析
解析 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj