+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1n92 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Horse Liver Alcohol Dehydrogenase Complexed with NAD+ and 4-Iodopyrazole | ||||||
 要素 要素 | Alcohol Dehydrogenase E chain | ||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  dehydrogenase (脱水素酵素) / dehydrogenase (脱水素酵素) /  alcohol / nicotinamide coenzyme / 4-iodopyrazole alcohol / nicotinamide coenzyme / 4-iodopyrazole | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alcohol dehydrogenase (NAD+) activity, zinc-dependent / : / all-trans-retinol dehydrogenase (NAD+) activity /  アルコールデヒドロゲナーゼ / retinoic acid metabolic process / retinol metabolic process / zinc ion binding / アルコールデヒドロゲナーゼ / retinoic acid metabolic process / retinol metabolic process / zinc ion binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Equus caballus (ウマ) Equus caballus (ウマ) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.47 Å 分子置換 / 解像度: 1.47 Å | ||||||
 データ登録者 データ登録者 | Rubach, J.K. / Plapp, B.V. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 2003 ジャーナル: Biochemistry / 年: 2003タイトル: Amino Acid Residues in the Nicotinamide Binding Site Contribute to Catalysis by Horse Liver Alcohol Dehydrogenase 著者: Rubach, J.K. / Plapp, B.V. #1:  ジャーナル: Biochemistry / 年: 1982 ジャーナル: Biochemistry / 年: 1982タイトル: Pyrazole Binding in Crystalline Binary and Ternary Complexes with Liver Alcohol Dehydrogenase 著者: Eklund, H. / Samama, J.P. / Wallen, L. #2:  ジャーナル: J.Biol.Chem. / 年: 1996 ジャーナル: J.Biol.Chem. / 年: 1996タイトル: X-Ray Structure of Human beta3beta3 Alcohol Dehydrogenase. The contribution of ionic interactions to coenzyme binding. 著者: Davis, G.J. / Bosron, W.F. / Stone, C.L. / Owusu-Dekyi, K. / Hurley, T.D. #3:  ジャーナル: J.Mol.Biol. / 年: 1994 ジャーナル: J.Mol.Biol. / 年: 1994タイトル: Structures of Three Human Beta Alcohol Dehydrogenase Variants. Correlations with their Functional Differences. 著者: Hurley, T.D. / Bosron, W.F. / Stone, C.L. / Amzel, L.M. #4:  ジャーナル: J.Mol.Biol. / 年: 1981 ジャーナル: J.Mol.Biol. / 年: 1981タイトル: Structure of A Triclinic Ternary Complex of Horse Liver Alcohol Dehydrogenase at 2.9 A Resolution 著者: Eklund, H. / Samama, J.P. / Wallen, L. / Branden, C.I. / Akeson, A. / Jones, T.A. #5:  ジャーナル: J.Mol.Biol. / 年: 1976 ジャーナル: J.Mol.Biol. / 年: 1976タイトル: Three-Dimensional Structure of Horse Liver Alcohol Dehydrogenase at 2.4 A Resolution 著者: Eklund, H. / Nordstrom, B. / Zeppezauer, E. / Soderlund, G. / Ohlsson, I. / Boiwe, T. / Soderberg, B.O. / Tapia, O. / Branden, C.I. / Akeson, A. | ||||||
| 履歴 |
| ||||||
| Remark 600 | HETEROGEN THE STRUCTURE DETERMINED IN THIS ENTRY CONTAINS THE HETEROATOM MOLECULE NAJ, WHICH IS ...HETEROGEN THE STRUCTURE DETERMINED IN THIS ENTRY CONTAINS THE HETEROATOM MOLECULE NAJ, WHICH IS OXIDIZED NICOTINAMIDE ADENINE DINUCLEOTIDE, BUT THE RESTRAINTS ON THE PLANARITY OF THE NICOTINAMIDE RING WERE REMOVED DURING REFINEMENT WITH THE RESULT THAT THE RING IS PUCKERED. THE STRUCTURE ALSO INCLUDES 4-IODOPYRAZOLE, WHICH FORMS A PARTIAL COVALENT BOND BETWEEN N2 AND NC4 OF THE NICOTINAMIDE RING. NOTE THAT THE ABBREVIATION "PYZ" MEANS 4-IODOPYRAZOLE. | ||||||
| Remark 285 | CRYST1 TO GENERATE THE STANDARD UNIT CELL FOR P1 FROM THE CELL DESCRIBED IN THE CRYST CARD, APPLY ...CRYST1 TO GENERATE THE STANDARD UNIT CELL FOR P1 FROM THE CELL DESCRIBED IN THE CRYST CARD, APPLY THE FOLLOWING: 0.000000 1.000000 0.000000 0.00000 1.000000 0.000000 -1.000000 0.00000 -1.000000 0.000000 0.000000 0.00000 WHICH YIELDS THE FOLLOWING UNIT CELL PARAMETERS: 51.0 92.5 44.2 102.9 109.8 91.7 P 1 |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1n92.cif.gz 1n92.cif.gz | 300 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1n92.ent.gz pdb1n92.ent.gz | 240.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1n92.json.gz 1n92.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/n9/1n92 https://data.pdbj.org/pub/pdb/validation_reports/n9/1n92 ftp://data.pdbj.org/pub/pdb/validation_reports/n9/1n92 ftp://data.pdbj.org/pub/pdb/validation_reports/n9/1n92 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 2分子 AB
| #1: タンパク質 | 分子量: 39853.273 Da / 分子数: 2 / 由来タイプ: 天然 / 由来: (天然)   Equus caballus (ウマ) / 器官: liver Equus caballus (ウマ) / 器官: liver 肝臓 肝臓参照: UniProt: P00327,  アルコールデヒドロゲナーゼ アルコールデヒドロゲナーゼ |
|---|
-非ポリマー , 5種, 590分子 
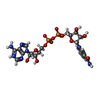
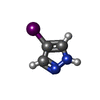






| #2: 化合物 | ChemComp-ZN / #3: 化合物 |  ニコチンアミドアデニンジヌクレオチド ニコチンアミドアデニンジヌクレオチド#4: 化合物 | #5: 化合物 | ChemComp-MPD / ( |  2-Methyl-2,4-pentanediol 2-Methyl-2,4-pentanediol#6: 水 | ChemComp-HOH / |  水 水 |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.17 Å3/Da / 溶媒含有率: 42.89 % | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
結晶化 | 温度: 277 K / 手法: dialysis / pH: 7 / 詳細: MPD, pH 7.0, Dialysis, temperature 277K | ||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / pH: 6.7 / 手法: microdialysis | ||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU / 波長: 1.5418 Å 回転陽極 / タイプ: RIGAKU / 波長: 1.5418 Å |
| 検出器 | タイプ: RIGAKU RAXIS IV / 検出器: IMAGE PLATE / 日付: 2001年3月28日 / 詳細: confocal osmic |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.47→20 Å / Num. all: 156075 / Num. obs: 109109 / % possible obs: 88.8 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / 冗長度: 2.1 % / Rmerge(I) obs: 0.054 / Rsym value: 0.054 / Net I/σ(I): 5.9 |
| 反射 シェル | 解像度: 1.47→1.508 Å / Rmerge(I) obs: 0.127 / Mean I/σ(I) obs: 4.1 / Num. unique all: 5097 / Rsym value: 0.127 / % possible all: 69 |
| 反射 | *PLUS Num. measured all: 227408 |
| 反射 シェル | *PLUS % possible obs: 69 % |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: PDB ENTRY 1HLD 解像度: 1.47→20 Å / Cor.coef. Fo:Fc: 0.972 / Cor.coef. Fo:Fc free: 0.961 / SU B: 1.121 / SU ML: 0.042 / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / ESU R: 0.078 / ESU R Free: 0.068 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: BABINET MODEL WITH MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 17.152 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.47→20 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.47→1.508 Å / Total num. of bins used: 20 /
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最低解像度: 20 Å / % reflection Rfree: 2.5 % / Rfactor Rfree : 0.183 : 0.183 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj





