+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20749 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of dimeric sIgA complex | |||||||||
 マップデータ マップデータ | Main map of sIgA Dimer. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 polymeric immunoglobulin receptor activity / immunoglobulin transcytosis in epithelial cells mediated by polymeric immunoglobulin receptor / dimeric IgA immunoglobulin complex / polymeric immunoglobulin binding / secretory dimeric IgA immunoglobulin complex / pentameric IgM immunoglobulin complex / monomeric IgA immunoglobulin complex / secretory IgA immunoglobulin complex / Fc receptor signaling pathway / IgA binding ... polymeric immunoglobulin receptor activity / immunoglobulin transcytosis in epithelial cells mediated by polymeric immunoglobulin receptor / dimeric IgA immunoglobulin complex / polymeric immunoglobulin binding / secretory dimeric IgA immunoglobulin complex / pentameric IgM immunoglobulin complex / monomeric IgA immunoglobulin complex / secretory IgA immunoglobulin complex / Fc receptor signaling pathway / IgA binding ... polymeric immunoglobulin receptor activity / immunoglobulin transcytosis in epithelial cells mediated by polymeric immunoglobulin receptor / dimeric IgA immunoglobulin complex / polymeric immunoglobulin binding / secretory dimeric IgA immunoglobulin complex / pentameric IgM immunoglobulin complex / monomeric IgA immunoglobulin complex / secretory IgA immunoglobulin complex / Fc receptor signaling pathway / IgA binding / detection of chemical stimulus involved in sensory perception of bitter taste / polymeric immunoglobulin receptor activity / immunoglobulin transcytosis in epithelial cells mediated by polymeric immunoglobulin receptor / dimeric IgA immunoglobulin complex / polymeric immunoglobulin binding / secretory dimeric IgA immunoglobulin complex / pentameric IgM immunoglobulin complex / monomeric IgA immunoglobulin complex / secretory IgA immunoglobulin complex / Fc receptor signaling pathway / IgA binding / detection of chemical stimulus involved in sensory perception of bitter taste /  クレアチニンクリアランス / IgA immunoglobulin complex / azurophil granule membrane / receptor clustering / immunoglobulin complex, circulating / IgG immunoglobulin complex / immunoglobulin receptor binding / positive regulation of respiratory burst / クレアチニンクリアランス / IgA immunoglobulin complex / azurophil granule membrane / receptor clustering / immunoglobulin complex, circulating / IgG immunoglobulin complex / immunoglobulin receptor binding / positive regulation of respiratory burst /  液性免疫 / Scavenging of heme from plasma / 液性免疫 / Scavenging of heme from plasma /  complement activation, classical pathway / complement activation, classical pathway /  antigen binding / Cell surface interactions at the vascular wall / B cell receptor signaling pathway / epidermal growth factor receptor signaling pathway / protein-macromolecule adaptor activity / antibacterial humoral response / protein-containing complex assembly / blood microparticle / antigen binding / Cell surface interactions at the vascular wall / B cell receptor signaling pathway / epidermal growth factor receptor signaling pathway / protein-macromolecule adaptor activity / antibacterial humoral response / protein-containing complex assembly / blood microparticle /  獲得免疫系 / 獲得免疫系 /  receptor complex / receptor complex /  免疫応答 / 免疫応答 /  自然免疫系 / Neutrophil degranulation / protein homodimerization activity / 自然免疫系 / Neutrophil degranulation / protein homodimerization activity /  extracellular space / extracellular exosome / extracellular region / extracellular space / extracellular exosome / extracellular region /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Kumar N / Arthur CP / Ciferri C / Matsumoto ML | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2020 ジャーナル: Science / 年: 2020タイトル: Structure of the secretory immunoglobulin A core. 著者: Nikit Kumar / Christopher P Arthur / Claudio Ciferri / Marissa L Matsumoto /  要旨: Secretory immunoglobulin A (sIgA) represents the immune system's first line of defense against mucosal pathogens. IgAs are transported across the epithelium, as dimers and higher-order polymers, by ...Secretory immunoglobulin A (sIgA) represents the immune system's first line of defense against mucosal pathogens. IgAs are transported across the epithelium, as dimers and higher-order polymers, by the polymeric immunoglobulin receptor (pIgR). Upon reaching the luminal side, sIgAs mediate host protection and pathogen neutralization. In recent years, an increasing amount of attention has been given to IgA as a novel therapeutic antibody. However, despite extensive studies, sIgA structures have remained elusive. Here, we determine the atomic resolution structures of dimeric, tetrameric, and pentameric IgA-Fc linked by the joining chain (JC) and in complex with the secretory component of the pIgR. We suggest a mechanism in which the JC templates IgA oligomerization and imparts asymmetry for pIgR binding and transcytosis. This framework will inform the design of future IgA-based therapeutics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20749.map.gz emd_20749.map.gz | 200.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20749-v30.xml emd-20749-v30.xml emd-20749.xml emd-20749.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20749.png emd_20749.png | 87.5 KB | ||
| その他 |  emd_20749_additional.map.gz emd_20749_additional.map.gz | 199.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20749 http://ftp.pdbj.org/pub/emdb/structures/EMD-20749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20749 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20749 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20749.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20749.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main map of sIgA Dimer. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.0188 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
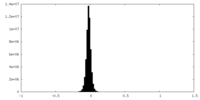
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: -150 sharpened map of sIgA Dimer complex.
| ファイル | emd_20749_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | -150 sharpened map of sIgA Dimer complex. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
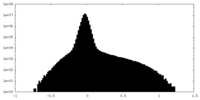
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : dimeric sIgA complex
| 全体 | 名称: dimeric sIgA complex |
|---|---|
| 要素 |
|
-超分子 #1: dimeric sIgA complex
| 超分子 | 名称: dimeric sIgA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 分子量 | 理論値: 188 KDa |
-分子 #1: Immunoglobulin heavy constant alpha 1
| 分子 | 名称: Immunoglobulin heavy constant alpha 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 26.667172 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: DYKDDDDKLV PRGSCHPRLS LHRPALEDLL LGSEANLTCT LTGLRDASGV TFTWTPSSGK SAVQGPPERD LCGCYSVSSV LPGCAEPWN HGKTFTCTAA YPESKTPLTA TLSKSGNTFR PEVHLLPPPS EELALNELVT LTCLARGFSP KDVLVRWLQG S QELPREKY ...文字列: DYKDDDDKLV PRGSCHPRLS LHRPALEDLL LGSEANLTCT LTGLRDASGV TFTWTPSSGK SAVQGPPERD LCGCYSVSSV LPGCAEPWN HGKTFTCTAA YPESKTPLTA TLSKSGNTFR PEVHLLPPPS EELALNELVT LTCLARGFSP KDVLVRWLQG S QELPREKY LTWASRQEPS QGTTTFAVTS ILRVAAEDWK KGDTFSCMVG HEALPLAFTQ KTIDRLAGKP THVNVSVVMA EV DGTCY |
-分子 #2: Polymeric immunoglobulin receptor
| 分子 | 名称: Polymeric immunoglobulin receptor / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 65.154094 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: KSPIFGPEEV NSVEGNSVSI TCYYPPTSVN RHTRKYWCRQ GARGGCITLI SSEGYVSSKY AGRANLTNFP ENGTFVVNIA QLSQDDSGR YKCGLGINSR GLSFDVSLEV SQGPGLLNDT KVYTVDLGRT VTINCPFKTE NAQKRKSLYK QIGLYPVLVI D SSGYVNPN ...文字列: KSPIFGPEEV NSVEGNSVSI TCYYPPTSVN RHTRKYWCRQ GARGGCITLI SSEGYVSSKY AGRANLTNFP ENGTFVVNIA QLSQDDSGR YKCGLGINSR GLSFDVSLEV SQGPGLLNDT KVYTVDLGRT VTINCPFKTE NAQKRKSLYK QIGLYPVLVI D SSGYVNPN YTGRIRLDIQ GTGQLLFSVV INQLRLSDAG QYLCQAGDDS NSNKKNADLQ VLKPEPELVY EDLRGSVTFH CA LGPEVAN VAKFLCRQSS GENCDVVVNT LGKRAPAFEG RILLNPQDKD GSFSVVITGL RKEDAGRYLC GAHSDGQLQE GSP IQAWQL FVNEESTIPR SPTVVKGVAG GSVAVLCPYN RKESKSIKYW CLWEGAQNGR CPLLVDSEGW VKAQYEGRLS LLEE PGNGT FTVILNQLTS RDAGFYWCLT NGDTLWRTTV EIKIIEGEPN LKVPGNVTAV LGETLKVPCH FPCKFSSYEK YWCKW NNTG CQALPSQDEG PSKAFVNCDE NSRLVSLTLN LVTRADEGWY WCGVKQGHFY GETAAVYVAV EERKAAGSRD VSLAKA DAA PDEKVLDSGF REIENKAIQD PRHHHHHH |
-分子 #3: Immunoglobulin J chain
| 分子 | 名称: Immunoglobulin J chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 15.611458 KDa |
| 組換発現 | 生物種:   Cricetulus griseus (モンゴルキヌゲネズミ) Cricetulus griseus (モンゴルキヌゲネズミ) |
| 配列 | 文字列: QEDERIVLVD NKCKCARITS RIIRSSEDPN EDIVERNIRI IVPLNNRENI SDPTSPLRTR FVYHLSDLCK KCDPTEVELD NQIVTATQS NICDEDSATE TCYTYDRNKC YTAVVPLVYG GETKMVETAL TPDACYPD |
-分子 #6: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 6 / コピー数: 7 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.18 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | モデル: UltrAuFoil / 材質: GOLD / メッシュ: 400 / 前処理 - タイプ: PLASMA CLEANING / 詳細: unspecified |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 倍率(公称値): 165000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 10.0 sec. / 平均電子線量: 51.91 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期 角度割当 | タイプ: RANDOM ASSIGNMENT |
|---|---|
| 最終 角度割当 | タイプ: OTHER / ソフトウェア - 名称: cisTEM |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cisTEM / 使用した粒子像数: 233402 |
 ムービー
ムービー コントローラー
コントローラー


































 Z
Z Y
Y X
X