登録情報 データベース : EMDB / ID : EMD-12041タイトル Focused map-catalytic side. Ubiquitin ligation to F-box protein substrates by SCF-RBR E3-E3 super-assembly: NEDD8-CUL1-RBX1-UBE2L3~Ub~ARIH1. 複合体 : NEDD8-CUL1-RBX1-SKP1-SKP2-CKSHS1-Cyclin A-CDK2-p27-UBE2L3~Ub~ARIH1. Transition State 1 composite map.複合体 : NEDD8-SKP1-SKP2-CKSHS1-Cyclin A-CDK2-p27-UBE2L3~Ub~ARIH1. Transition State 1 composite map.タンパク質・ペプチド : E3 ubiquitin-protein ligase ARIH1タンパク質・ペプチド : Polyubiquitin-Cタンパク質・ペプチド : NEDD8タンパク質・ペプチド : Ubiquitin-conjugating enzyme E2 L3複合体 : CUL1, RBX1タンパク質・ペプチド : Cullin-1タンパク質・ペプチド : E3 ubiquitin-protein ligase RBX1リガンド : ZINC IONリガンド : 5-azanylpentan-2-one機能・相同性 分子機能 ドメイン・相同性 構成要素
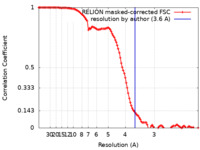
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / / 解像度 : 3.6 Å Horn-Ghetko D / Prabu JR / Schulman BA 資金援助 Organization Grant number 国 European Research Council (ERC) H2020 789016-NEDD8Activate German Research Foundation (DFG) SCHU 3196/1-1
ジャーナル : Nature / 年 : 2021タイトル : Ubiquitin ligation to F-box protein targets by SCF-RBR E3-E3 super-assembly.著者 : Daniel Horn-Ghetko / David T Krist / J Rajan Prabu / Kheewoong Baek / Monique P C Mulder / Maren Klügel / Daniel C Scott / Huib Ovaa / Gary Kleiger / Brenda A Schulman / 要旨 : E3 ligases are typically classified by hallmark domains such as RING and RBR, which are thought to specify unique catalytic mechanisms of ubiquitin transfer to recruited substrates. However, rather ... E3 ligases are typically classified by hallmark domains such as RING and RBR, which are thought to specify unique catalytic mechanisms of ubiquitin transfer to recruited substrates. However, rather than functioning individually, many neddylated cullin-RING E3 ligases (CRLs) and RBR-type E3 ligases in the ARIH family-which together account for nearly half of all ubiquitin ligases in humans-form E3-E3 super-assemblies. Here, by studying CRLs in the SKP1-CUL1-F-box (SCF) family, we show how neddylated SCF ligases and ARIH1 (an RBR-type E3 ligase) co-evolved to ubiquitylate diverse substrates presented on various F-box proteins. We developed activity-based chemical probes that enabled cryo-electron microscopy visualization of steps in E3-E3 ubiquitylation, initiating with ubiquitin linked to the E2 enzyme UBE2L3, then transferred to the catalytic cysteine of ARIH1, and culminating in ubiquitin linkage to a substrate bound to the SCF E3 ligase. The E3-E3 mechanism places the ubiquitin-linked active site of ARIH1 adjacent to substrates bound to F-box proteins (for example, substrates with folded structures or limited length) that are incompatible with previously described conventional RING E3-only mechanisms. The versatile E3-E3 super-assembly may therefore underlie widespread ubiquitylation. 履歴 登録 2020年12月5日 - ヘッダ(付随情報) 公開 2021年2月10日 - マップ公開 2021年2月10日 - 更新 2021年3月10日 - 現状 2021年3月10日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 レビー小体 / Parkin-FBXW7-Cul1 ubiquitin ligase complex / RBR-type E3 ubiquitin transferase / cell cycle phase transition / ubiquitin-protein transferase activator activity / cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity ...PKR/eIFalpha signaling / ubiquitin-like protein transferase activity /
レビー小体 / Parkin-FBXW7-Cul1 ubiquitin ligase complex / RBR-type E3 ubiquitin transferase / cell cycle phase transition / ubiquitin-protein transferase activator activity / cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity ...PKR/eIFalpha signaling / ubiquitin-like protein transferase activity /  レビー小体 / Parkin-FBXW7-Cul1 ubiquitin ligase complex / RBR-type E3 ubiquitin transferase / cell cycle phase transition / ubiquitin-protein transferase activator activity / cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway /
レビー小体 / Parkin-FBXW7-Cul1 ubiquitin ligase complex / RBR-type E3 ubiquitin transferase / cell cycle phase transition / ubiquitin-protein transferase activator activity / cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / cullin-RING ubiquitin ligase complex / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway /  regulation of proteolysis / protein K11-linked ubiquitination / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / protein neddylation / ubiquitin conjugating enzyme binding / cellular response to glucocorticoid stimulus / positive regulation of ubiquitin-protein transferase activity / NEDD8 ligase activity / Cul5-RING ubiquitin ligase complex / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / ubiquitin-ubiquitin ligase activity / positive regulation of protein targeting to mitochondrion / Cul4A-RING E3 ubiquitin ligase complex /
regulation of proteolysis / protein K11-linked ubiquitination / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / positive regulation of protein autoubiquitination / protein neddylation / ubiquitin conjugating enzyme binding / cellular response to glucocorticoid stimulus / positive regulation of ubiquitin-protein transferase activity / NEDD8 ligase activity / Cul5-RING ubiquitin ligase complex / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / ubiquitin-ubiquitin ligase activity / positive regulation of protein targeting to mitochondrion / Cul4A-RING E3 ubiquitin ligase complex /  ユビキチン結合酵素 / Cul2-RING ubiquitin ligase complex /
ユビキチン結合酵素 / Cul2-RING ubiquitin ligase complex /  SCF複合体 / negative regulation of type I interferon production / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / Cul3-RING ubiquitin ligase complex / cellular response to steroid hormone stimulus / Prolactin receptor signaling / protein monoubiquitination / ubiquitin conjugating enzyme activity / TGF-beta receptor signaling activates SMADs / cullin family protein binding / protein K48-linked ubiquitination / anatomical structure morphogenesis /
SCF複合体 / negative regulation of type I interferon production / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / Cul3-RING ubiquitin ligase complex / cellular response to steroid hormone stimulus / Prolactin receptor signaling / protein monoubiquitination / ubiquitin conjugating enzyme activity / TGF-beta receptor signaling activates SMADs / cullin family protein binding / protein K48-linked ubiquitination / anatomical structure morphogenesis /  カハール体 / Nuclear events stimulated by ALK signaling in cancer /
カハール体 / Nuclear events stimulated by ALK signaling in cancer /  ubiquitin ligase complex / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway /
ubiquitin ligase complex / Maturation of protein E / Maturation of protein E / ER Quality Control Compartment (ERQC) / Myoclonic epilepsy of Lafora / FLT3 signaling by CBL mutants / Prevention of phagosomal-lysosomal fusion / IRAK2 mediated activation of TAK1 complex / Alpha-protein kinase 1 signaling pathway /  グリコーゲン合成 / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Constitutive Signaling by NOTCH1 HD Domain Mutants / Endosomal Sorting Complex Required For Transport (ESCRT) / NOTCH2 Activation and Transmission of Signal to the Nucleus / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / positive regulation of TORC1 signaling / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / p75NTR recruits signalling complexes / Downregulation of ERBB4 signaling / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Pexophagy / InlA-mediated entry of Listeria monocytogenes into host cells / Regulation of innate immune responses to cytosolic DNA / VLDLR internalisation and degradation / Downregulation of ERBB2:ERBB3 signaling / NRIF signals cell death from the nucleus / Activated NOTCH1 Transmits Signal to the Nucleus / NF-kB is activated and signals survival / Regulation of PTEN localization /
グリコーゲン合成 / IRAK1 recruits IKK complex / IRAK1 recruits IKK complex upon TLR7/8 or 9 stimulation / Membrane binding and targetting of GAG proteins / Constitutive Signaling by NOTCH1 HD Domain Mutants / Endosomal Sorting Complex Required For Transport (ESCRT) / NOTCH2 Activation and Transmission of Signal to the Nucleus / IRAK2 mediated activation of TAK1 complex upon TLR7/8 or 9 stimulation / positive regulation of TORC1 signaling / PTK6 Regulates RTKs and Their Effectors AKT1 and DOK1 / Negative regulation of FLT3 / Regulation of FZD by ubiquitination / TICAM1,TRAF6-dependent induction of TAK1 complex / TICAM1-dependent activation of IRF3/IRF7 / APC/C:Cdc20 mediated degradation of Cyclin B / p75NTR recruits signalling complexes / Downregulation of ERBB4 signaling / TRAF6 mediated IRF7 activation in TLR7/8 or 9 signaling / APC-Cdc20 mediated degradation of Nek2A / PINK1-PRKN Mediated Mitophagy / TRAF6-mediated induction of TAK1 complex within TLR4 complex / Pexophagy / InlA-mediated entry of Listeria monocytogenes into host cells / Regulation of innate immune responses to cytosolic DNA / VLDLR internalisation and degradation / Downregulation of ERBB2:ERBB3 signaling / NRIF signals cell death from the nucleus / Activated NOTCH1 Transmits Signal to the Nucleus / NF-kB is activated and signals survival / Regulation of PTEN localization /  Regulation of BACH1 activity / Translesion synthesis by REV1 /
Regulation of BACH1 activity / Translesion synthesis by REV1 /  T細胞 / Synthesis of active ubiquitin: roles of E1 and E2 enzymes /
T細胞 / Synthesis of active ubiquitin: roles of E1 and E2 enzymes /  post-translational protein modification / Translesion synthesis by POLK / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / intrinsic apoptotic signaling pathway / Gap-filling DNA repair synthesis and ligation in GG-NER / Downregulation of TGF-beta receptor signaling / Translesion synthesis by POLI / Josephin domain DUBs / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Regulation of activated PAK-2p34 by proteasome mediated degradation / InlB-mediated entry of Listeria monocytogenes into host cell / IKK complex recruitment mediated by RIP1
post-translational protein modification / Translesion synthesis by POLK / MAP3K8 (TPL2)-dependent MAPK1/3 activation / TICAM1, RIP1-mediated IKK complex recruitment / intrinsic apoptotic signaling pathway / Gap-filling DNA repair synthesis and ligation in GG-NER / Downregulation of TGF-beta receptor signaling / Translesion synthesis by POLI / Josephin domain DUBs / Activation of IRF3, IRF7 mediated by TBK1, IKKε (IKBKE) / Regulation of activated PAK-2p34 by proteasome mediated degradation / InlB-mediated entry of Listeria monocytogenes into host cell / IKK complex recruitment mediated by RIP1
 Homo sapiens (ヒト)
Homo sapiens (ヒト) 単粒子再構成法 /
単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å
クライオ電子顕微鏡法 / 解像度: 3.6 Å  データ登録者
データ登録者 ドイツ, 2件
ドイツ, 2件  引用
引用 ジャーナル: Nature / 年: 2021
ジャーナル: Nature / 年: 2021


 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_12041.map.gz
emd_12041.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-12041-v30.xml
emd-12041-v30.xml emd-12041.xml
emd-12041.xml EMDBヘッダ
EMDBヘッダ emd_12041_fsc.xml
emd_12041_fsc.xml FSCデータファイル
FSCデータファイル emd_12041.png
emd_12041.png emd_12041_msk_1.map
emd_12041_msk_1.map マスクマップ
マスクマップ emd_12041_additional_1.map.gz
emd_12041_additional_1.map.gz emd_12041_half_map_1.map.gz
emd_12041_half_map_1.map.gz emd_12041_half_map_2.map.gz
emd_12041_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-12041
http://ftp.pdbj.org/pub/emdb/structures/EMD-12041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12041
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-12041 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_12041.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_12041.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_12041_msk_1.map
emd_12041_msk_1.map 試料の構成要素
試料の構成要素 クライオ電子顕微鏡法
クライオ電子顕微鏡法 解析
解析 単粒子再構成法
単粒子再構成法 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN Bright-field microscopy
Bright-field microscopy
 ムービー
ムービー コントローラー
コントローラー














































 Z
Z Y
Y X
X