+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-11729 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of MIB-MIP in complex with a polyclonal goat Fab | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 | Uncharacterised protein family MG067 / Mycoplasma peptidase DUF31 / Mycoplasma peptidase (DUF31) / Mycoplasma virulence, signal domain / Putative immunoglobulin-blocking virulence protein / Mycoplasma virulence signal region (Myco_arth_vir_N) / IgG-blocking virulence domain / Putative immunoglobulin-blocking virulence protein /  リポタンパク質 リポタンパク質 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Mycoplasma mycoides subsp. capri str. GM12 (牛肺疫菌) / Mycoplasma mycoides subsp. capri str. GM12 (牛肺疫菌) /   Mycoplasma mycoides subsp. capri (牛肺疫菌) Mycoplasma mycoides subsp. capri (牛肺疫菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.8 Å クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Nottelet P / Bataille L / Gourgues G / Anger R / Lartigue C / Sirand-Pugnet P / Marza E / Fronzes R / Arfi Y | |||||||||
| 資金援助 |  フランス, 1件 フランス, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: The mycoplasma surface proteins MIB and MIP promote the dissociation of the antibody-antigen interaction. 著者: Pierre Nottelet / Laure Bataille / Geraldine Gourgues / Robin Anger / Carole Lartigue / Pascal Sirand-Pugnet / Esther Marza / Remi Fronzes / Yonathan Arfi /  要旨: Mycoplasma immunoglobulin binding (MIB) and mycoplasma immunoglobulin protease (MIP) are surface proteins found in the majority of mycoplasma species, acting sequentially to capture antibodies and ...Mycoplasma immunoglobulin binding (MIB) and mycoplasma immunoglobulin protease (MIP) are surface proteins found in the majority of mycoplasma species, acting sequentially to capture antibodies and cleave off their V domains. Cryo-electron microscopy structures show how MIB and MIP bind to a Fab fragment in a "hug of death" mechanism. As a result, the orientation of the V and V domains is twisted out of alignment, disrupting the antigen binding site. We also show that MIB-MIP has the ability to promote the dissociation of the antibody-antigen complex. This system is functional in cells and protects mycoplasmas from antibody-mediated agglutination. These results highlight the key role of the MIB-MIP system in immunity evasion by mycoplasmas through an unprecedented mechanism, and open exciting perspectives to use these proteins as potential tools in the antibody field. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_11729.map.gz emd_11729.map.gz | 59.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-11729-v30.xml emd-11729-v30.xml emd-11729.xml emd-11729.xml | 19 KB 19 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_11729.png emd_11729.png | 47.8 KB | ||
| その他 |  emd_11729_additional_1.map.gz emd_11729_additional_1.map.gz emd_11729_half_map_1.map.gz emd_11729_half_map_1.map.gz emd_11729_half_map_2.map.gz emd_11729_half_map_2.map.gz | 49.4 MB 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-11729 http://ftp.pdbj.org/pub/emdb/structures/EMD-11729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11729 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-11729 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_11729.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_11729.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_11729_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
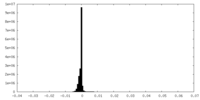
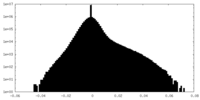
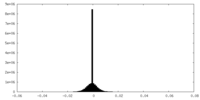
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_11729_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
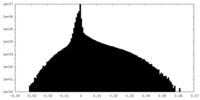
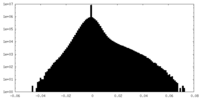
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_11729_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
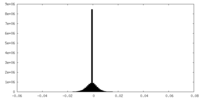
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mycoplasma MIB-MIP proteins in complex with a goat Fab
| 全体 | 名称: Mycoplasma MIB-MIP proteins in complex with a goat Fab |
|---|---|
| 要素 |
|
-超分子 #1: Mycoplasma MIB-MIP proteins in complex with a goat Fab
| 超分子 | 名称: Mycoplasma MIB-MIP proteins in complex with a goat Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Mycoplasma mycoides subsp. capri str. GM12 (牛肺疫菌) Mycoplasma mycoides subsp. capri str. GM12 (牛肺疫菌) |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) / 組換株: Rosetta Escherichia coli BL21(DE3) (大腸菌) / 組換株: Rosetta |
-分子 #1: Putative immunoglobulin-blocking virulence protein
| 分子 | 名称: Putative immunoglobulin-blocking virulence protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mycoplasma mycoides subsp. capri (牛肺疫菌) Mycoplasma mycoides subsp. capri (牛肺疫菌) |
| 分子量 | 理論値: 84.962719 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: VYFLKKKKNK ILTIALVASL AASVSFGSVL YYSFSDNHIS FDTSSNGITD AELAPINNAI NDAIVSNRDN KLKPSEEKII KETEKKIEE KIIIPPAKKE EKIEAAKPIP KPVVRKPETK ITSPKITRRK QTITIAGIEV EAEIEGPPGF VTHQRDKDRK I SNPTKPYQ ...文字列: VYFLKKKKNK ILTIALVASL AASVSFGSVL YYSFSDNHIS FDTSSNGITD AELAPINNAI NDAIVSNRDN KLKPSEEKII KETEKKIEE KIIIPPAKKE EKIEAAKPIP KPVVRKPETK ITSPKITRRK QTITIAGIEV EAEIEGPPGF VTHQRDKDRK I SNPTKPYQ NHTVNKILSV KVTDKLKEQV AKDALSGGNG YDEGVGLFNN SIFNVFKEEF NSGKELNDIL SSLESVARQN SG AFQNTLE RYKKMLDSNN VINFLKSEAQ KEYPKLKSKF QTKNQEYIWL IANLDQSKFT KIASTSEKYL EKGLTISPRS AFI NEAGEI DSNGWGPPDE YNTVTSRLRR DNSEYRVFDY DEYYSRSSDR IANGTYPGWV KEDVSEPYSK KYNFKASDGI RFSK LERIN PNPAKGKLNS GLVLDLDVSN DEAYRRSKEL IEKLQKDGEQ ITSYRIKNMG EKNSDQAFKD ILGALPKDIQ QLELF FSDK ATNTASLIAL ENKNIKELSL YTSGNSLKKA WSYNPLALRN TTWINTIDYN VSAEYSSHDK ITTRITFNTL AFDQED FSN GSYERINDGL RMVYYARNNE PFFQGGHGPG LEPDKKLGQN SYPTGLDFSR VTGIKSLKGL RFDDDLDTSN EPRKITE LT LYNNESYFEI SSDELNEANL QHLSTGEGNP EKPKIHFSNG NNTTSIRISG KTLLSDEGRR NLDKYFEYNE SLRNSGKQ I QIPNGSDELK KQLEGWGYKV STASDRSFT |
-分子 #2: Lipoprotein
| 分子 | 名称: Lipoprotein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mycoplasma mycoides subsp. capri (牛肺疫菌) Mycoplasma mycoides subsp. capri (牛肺疫菌) |
| 分子量 | 理論値: 98.559016 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: MKRLNKLLMY ISSSTLLLPI TLLVACTPSK VVAKPINDDE FNKLIDSIKT ENDLLKYADI RFKNPNGADT KKEDIIPSQL KSENISITF KGKYQGQISA VVTNVDVDRT NPFAVQNEAT IFVQFKNLKT NTSKPISITI KGLNQKGNFD ASGNIVVDDF A YFGGTSGY ...文字列: MKRLNKLLMY ISSSTLLLPI TLLVACTPSK VVAKPINDDE FNKLIDSIKT ENDLLKYADI RFKNPNGADT KKEDIIPSQL KSENISITF KGKYQGQISA VVTNVDVDRT NPFAVQNEAT IFVQFKNLKT NTSKPISITI KGLNQKGNFD ASGNIVVDDF A YFGGTSGY DEYTKKDQKS RFDYDNERYM TRLKSQFGNS SNSINLKEYR GLETKQENIK KFDDQAAISN FDTYYNAALK GF TLPVYGS DGKVSGLKIY EGAEIGKGPS VVDSLGRNEK AKTVGLARTL PNEEYKTSAI QTFQTNFTIY KDYEKEIEEA EDN IKLFDS WNEQQIQSYI SAQLTQLRLN YEDEVSQIDR EISQTQPDKT TILSNLNQKK SKIESEYQKE LSTISKLNKD SLKE WQRKE IEKYNEKKKE KTFQISESGT MWIMDYLDEN AGKNPTKFYF GTNSHVAKGI KDGMVSFSLT RLNSEVKVGQ TFKLN GHDS NFTKFTFSPI NGNKLEDAVT AIFHATDFIN ENSSPLKLLD SEQKSKYNGA GIFADFAIVE VDFAKLLDKG KYSYSV WSA SNDITNQYET EQNKLISKIT NNYSESDKKV KFFSDSLLNE QTYAKFDRPL DFDPKKEDEL KKYNDLDSLY IVGYPTA YK DFYLDQYEDE KQLKNKKYDF SLWINSEYKF YNKLINKEGS TNSFKEYETG KGNFFSYQIG YRSFIDKPGL TDAFITVN K VGKKLYSLKD KNKNEVKKYF NYGLEILPRF YAPAGGASGS SVRTKDNKLL AVYHASNETA RTGLAVAFRS DGYDYKNLF GDYKLGQYDL IYGGGKDQQK EKSYREVMNK MYSGKKSALF QNGFTDDKIP SEFKFNNGTQ N |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm Bright-field microscopy / Cs: 2.7 mm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 1.45 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | ソフトウェア - 名称: Gctf |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION / 使用した粒子像数: 255911 |
 ムービー
ムービー コントローラー
コントローラー


















 Z
Z Y
Y X
X