+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-0406 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Single-particle cryo-EM reconstruction of horse liver alcohol dehydrogenase using 200 keV | |||||||||
 マップデータ マップデータ | Single-particle cryo-EM reconstruction of horse liver alcohol dehydrogenase (sharpened) | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報alcohol dehydrogenase (NAD+) activity, zinc-dependent / : / all-trans-retinol dehydrogenase (NAD+) activity /  アルコールデヒドロゲナーゼ / retinoic acid metabolic process / retinol metabolic process / zinc ion binding / アルコールデヒドロゲナーゼ / retinoic acid metabolic process / retinol metabolic process / zinc ion binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Equus caballus (ウマ) Equus caballus (ウマ) | |||||||||
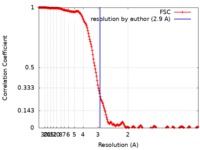
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.9 Å クライオ電子顕微鏡法 / 解像度: 2.9 Å | |||||||||
 データ登録者 データ登録者 | Herzik Jr MA / Wu M / Lander GC | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2019 ジャーナル: Nat Commun / 年: 2019タイトル: High-resolution structure determination of sub-100 kDa complexes using conventional cryo-EM. 著者: Mark A Herzik / Mengyu Wu / Gabriel C Lander /  要旨: Determining high-resolution structures of biological macromolecules amassing less than 100 kilodaltons (kDa) has been a longstanding goal of the cryo-electron microscopy (cryo-EM) community. While ...Determining high-resolution structures of biological macromolecules amassing less than 100 kilodaltons (kDa) has been a longstanding goal of the cryo-electron microscopy (cryo-EM) community. While the Volta phase plate has enabled visualization of specimens in this size range, this instrumentation is not yet fully automated and can present technical challenges. Here, we show that conventional defocus-based cryo-EM methodologies can be used to determine high-resolution structures of specimens amassing less than 100 kDa using a transmission electron microscope operating at 200 keV coupled with a direct electron detector. Our ~2.7 Å structure of alcohol dehydrogenase (82 kDa) proves that bound ligands can be resolved with high fidelity to enable investigation of drug-target interactions. Our ~2.8 Å and ~3.2 Å structures of methemoglobin demonstrate that distinct conformational states can be identified within a dataset for proteins as small as 64 kDa. Furthermore, we provide the sub-nanometer cryo-EM structure of a sub-50 kDa protein. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_0406.map.gz emd_0406.map.gz | 176.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-0406-v30.xml emd-0406-v30.xml emd-0406.xml emd-0406.xml | 22.1 KB 22.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_0406_fsc.xml emd_0406_fsc.xml | 21.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_0406.png emd_0406.png | 155.6 KB | ||
| マスクデータ |  emd_0406_msk_1.map emd_0406_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| その他 |  emd_0406_additional.map.gz emd_0406_additional.map.gz emd_0406_half_map_1.map.gz emd_0406_half_map_1.map.gz emd_0406_half_map_2.map.gz emd_0406_half_map_2.map.gz | 172.4 MB 408.2 MB 408.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-0406 http://ftp.pdbj.org/pub/emdb/structures/EMD-0406 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0406 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-0406 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6nbbMC  0407C  0408C  0409C  6nbcC  6nbdC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10249 (タイトル: Horse liver alcohol dehydrogenase movies obtained using Talos Arctica operating at 200 kV equipped with a K2 EMPIAR-10249 (タイトル: Horse liver alcohol dehydrogenase movies obtained using Talos Arctica operating at 200 kV equipped with a K2Data size: 1.3 TB Data #1: Raw, unaligned movies of horse liver alcohol dehydrogenase acquired on a Talos Arctica using a K2 direct electron detector [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_0406.map.gz / 形式: CCP4 / 大きさ: 190.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_0406.map.gz / 形式: CCP4 / 大きさ: 190.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle cryo-EM reconstruction of horse liver alcohol dehydrogenase (sharpened) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.5585 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_0406_msk_1.map emd_0406_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
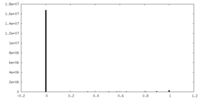
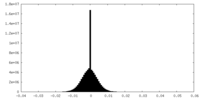
| 密度ヒストグラム |
-追加マップ: Single-particle cryo-EM reconstruction of horse liver alcohol dehydrogenase...
| ファイル | emd_0406_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Single-particle cryo-EM reconstruction of horse liver alcohol dehydrogenase (unsharpened) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
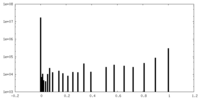
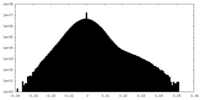
| 密度ヒストグラム |
-ハーフマップ: Even half map
| ファイル | emd_0406_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Even half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
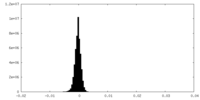
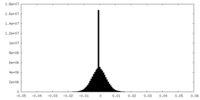
| 密度ヒストグラム |
-ハーフマップ: Odd half map
| ファイル | emd_0406_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Odd half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
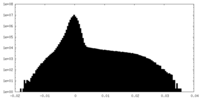
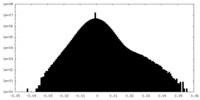
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Alcohol dehydrogenase from equine liver
| 全体 | 名称: Alcohol dehydrogenase from equine liver アルコールデヒドロゲナーゼ アルコールデヒドロゲナーゼ |
|---|---|
| 要素 |
|
-超分子 #1: Alcohol dehydrogenase from equine liver
| 超分子 | 名称: Alcohol dehydrogenase from equine liver / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Lyophilized horse liver ADH purchased from Sigma Aldrich was further purified to homogeneity. |
|---|---|
| 由来(天然) | 生物種:   Equus caballus (ウマ) Equus caballus (ウマ) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 分子量 | 理論値: 81 KDa |
-分子 #1: Alcohol dehydrogenase E chain
| 分子 | 名称: Alcohol dehydrogenase E chain / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  アルコールデヒドロゲナーゼ アルコールデヒドロゲナーゼ |
|---|---|
| 由来(天然) | 生物種:   Equus caballus (ウマ) Equus caballus (ウマ) |
| 分子量 | 理論値: 39.853273 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: STAGKVIKCK AAVLWEEKKP FSIEEVEVAP PKAHEVRIKM VATGICRSDD HVVSGTLVTP LPVIAGHEAA GIVESIGEGV TTVRPGDKV IPLFTPQCGK CRVCKHPEGN FCLKNDLSMP RGTMQDGTSR FTCRGKPIHH FLGTSTFSQY TVVDEISVAK I DAASPLEK ...文字列: STAGKVIKCK AAVLWEEKKP FSIEEVEVAP PKAHEVRIKM VATGICRSDD HVVSGTLVTP LPVIAGHEAA GIVESIGEGV TTVRPGDKV IPLFTPQCGK CRVCKHPEGN FCLKNDLSMP RGTMQDGTSR FTCRGKPIHH FLGTSTFSQY TVVDEISVAK I DAASPLEK VCLIGCGFST GYGSAVKVAK VTQGSTCAVF GLGGVGLSVI MGCKAAGAAR IIGVDINKDK FAKAKEVGAT EC VNPQDYK KPIQEVLTEM SNGGVDFSFE VIGRLDTMVT ALSCCQEAYG VSVIVGVPPD SQNLSMNPML LLSGRTWKGA IFG GFKSKD SVPKLVADFM AKKFALDPLI THVLPFEKIN EGFDLLRSGE SIRTILTF |
-分子 #2: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 2 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2.5 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8.5 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil, UltrAuFoil, R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER 詳細: Grids were plasma cleaned using a Solarus plasma cleaner (Gatan, Inc.). | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277.15 K / 装置: HOMEMADE PLUNGER 詳細: Sample was manually blotted for 4-5 seconds using Whatman No. 1 filter paper immediately prior to plunge-freezing.. | |||||||||||||||
| 詳細 | Lyophilized horse liver ADH (Sigma Aldrich) was solubilized and further purified to homogeneity. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 16.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 73000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 16.0 µm / 最小 デフォーカス(公称値): 5.0 µm / 倍率(公称値): 73000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - サンプリング間隔: 5.0 µm / デジタル化 - 画像ごとのフレーム数: 1-44 / 撮影したグリッド数: 2 / 実像数: 1151 / 平均露光時間: 11.0 sec. / 平均電子線量: 69.0 e/Å2 |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z
Z Y
Y X
X