+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: SASBDB / ID: SASDAK4 |
|---|---|
 試料 試料 | CRM1 RanGTP
|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Estrogen-dependent nuclear events downstream of ESR-membrane signaling / Cyclin A/B1/B2 associated events during G2/M transition / HuR (ELAVL1) binds and stabilizes mRNA / Heme signaling / cellular response to triglyceride / cellular response to salt / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / Deactivation of the beta-catenin transactivating complex / Transcriptional and post-translational regulation of MITF-M expression and activity / Mitotic Prometaphase ...Estrogen-dependent nuclear events downstream of ESR-membrane signaling / Cyclin A/B1/B2 associated events during G2/M transition / HuR (ELAVL1) binds and stabilizes mRNA / Heme signaling / cellular response to triglyceride / cellular response to salt / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / Deactivation of the beta-catenin transactivating complex / Transcriptional and post-translational regulation of MITF-M expression and activity / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / Resolution of Sister Chromatid Cohesion / annulate lamellae / RHO GTPases Activate Formins / regulation of proteasomal ubiquitin-dependent protein catabolic process / Separation of Sister Chromatids / MAPK6/MAPK4 signaling / pre-miRNA export from nucleus / RNA nuclear export complex / snRNA import into nucleus / regulation of centrosome duplication / nuclear export signal receptor activity / manchette / cellular response to mineralocorticoid stimulus / Regulation of cholesterol biosynthesis by SREBP (SREBF) / importin-alpha family protein binding / regulation of protein export from nucleus / Rev-mediated nuclear export of HIV RNA / Nuclear import of Rev protein / protein localization to nucleolus / NEP/NS2 Interacts with the Cellular Export Machinery / GTP metabolic process / tRNA processing in the nucleus / Postmitotic nuclear pore complex (NPC) reformation / MicroRNA (miRNA) biogenesis / DNA metabolic process / regulation of protein catabolic process / dynein intermediate chain binding / mitotic sister chromatid segregation / ribosomal large subunit export from nucleus / spermatid development / protein localization to nucleus / viral process / positive regulation of protein binding / sperm flagellum / nuclear pore / mRNA export from nucleus / ribosomal subunit export from nucleus / Cajal body / ribosomal small subunit export from nucleus / centriole / protein export from nucleus / mitotic spindle organization / male germ cell nucleus / hippocampus development / Transcriptional regulation by small RNAs / recycling endosome / kinetochore / positive regulation of protein import into nucleus / small GTPase binding / protein import into nucleus / GDP binding / melanosome / nuclear envelope / mitotic cell cycle / G protein activity / actin cytoskeleton organization / midbody / nuclear membrane / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / DNA-binding transcription factor binding / cadherin binding / response to xenobiotic stimulus / ribonucleoprotein complex / protein heterodimerization activity / protein domain specific binding / cell division / GTPase activity / chromatin binding / chromatin / GTP binding / protein-containing complex binding / nucleolus / magnesium ion binding / negative regulation of transcription by RNA polymerase II / protein-containing complex / RNA binding / extracellular exosome / nucleoplasm / nucleus / membrane / cytoplasm / cytosol 類似検索 - 分子機能 |
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
 引用 引用 |  ジャーナル: Structure / 年: 2013 ジャーナル: Structure / 年: 2013タイトル: Structural determinants and mechanism of mammalian CRM1 allostery. 著者: Nicole Dölker / Clement E Blanchet / Béla Voß / David Haselbach / Christian Kappel / Thomas Monecke / Dmitri I Svergun / Holger Stark / Ralf Ficner / Ulrich Zachariae / Helmut Grubmüller / Achim Dickmanns /  要旨: Proteins carrying nuclear export signals cooperatively assemble with the export factor CRM1 and the effector protein RanGTP. In lower eukaryotes, this cooperativity is coupled to CRM1 conformational ...Proteins carrying nuclear export signals cooperatively assemble with the export factor CRM1 and the effector protein RanGTP. In lower eukaryotes, this cooperativity is coupled to CRM1 conformational changes; however, it is unknown if mammalian CRM1 maintains its compact conformation or shows similar structural flexibility. Here, combinations of small-angle X-ray solution scattering and electron microscopy experiments with molecular dynamics simulations reveal pronounced conformational flexibility in mammalian CRM1 and demonstrate that RanGTP binding induces association of its N- and C-terminal regions to form a toroid structure. The CRM1 toroid is stabilized mainly by local interactions between the terminal regions, rather than by global strain. The CRM1 acidic loop is key in transmitting the effect of this RanGTP-induced global conformational change to the NES-binding cleft by shifting its population to the open state, which displays enhanced cargo affinity. Cooperative CRM1 export complex assembly thus constitutes a highly dynamic process, encompassing an intricate interplay of global and local structural changes. |
 登録者 登録者 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-モデル
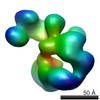
| モデル #69 |  タイプ: dummy / ソフトウェア: Dammif / ダミー原子の半径: 1.90 A  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
|---|---|
| モデル #72 |  タイプ: dummy / ソフトウェア: Monsa / ダミー原子の半径: 1.90 A / カイ2乗値: 1.515361  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
| モデル #73 |  タイプ: dummy / ソフトウェア: Monsa / ダミー原子の半径: 1.90 A / カイ2乗値: 1.560001  Omokage検索でこの集合体の類似形状データを探す (詳細) Omokage検索でこの集合体の類似形状データを探す (詳細) |
- 試料
試料
 試料 試料 | 名称: CRM1 RanGTP / Sample MW: 147.51 kDa / 試料濃度: 1.00-10.00 / Entity id: 62 / 63 |
|---|---|
| バッファ | 名称: 50 mM Tris-HCL / pH: 7.5 / 組成: 150 mM NaCl, 1.0 mM DTT |
| 要素 #62 | タイプ: protein / 記述: Exportin-1 / 分子量: 123.09 / 分子数: 1 / 由来: Mus musculus / 参照: UniProt: Q6P5F9 配列: MPAIMTMLAD HAARQLLDFS QKLDINLLDN VVNCLYHGEG AQQRMAQEVL THLKEHPDAW TRVDTILEFS QNMNTKYYGL QILENVIKTR WKILPRNQCE GIKKYVVGLI IKTSSDPTCV EKEKVYIGKL NMILVQILKQ EWPKHWPTFI SDIVGASRTS ESLCQNNMVI ...配列: MPAIMTMLAD HAARQLLDFS QKLDINLLDN VVNCLYHGEG AQQRMAQEVL THLKEHPDAW TRVDTILEFS QNMNTKYYGL QILENVIKTR WKILPRNQCE GIKKYVVGLI IKTSSDPTCV EKEKVYIGKL NMILVQILKQ EWPKHWPTFI SDIVGASRTS ESLCQNNMVI LKLLSEEVFD FSSGQITQVK AKHLKDSMCN EFSQIFQLCQ FVMENSQNAP LVHATLETLL RFLNWIPLGY IFETKLISTL IYKFLNVPMF RNVSLKCLTE IAGVSVSQYE EQFETLFTLT MMQLKQMLPL NTNIRLAYSN GKDDEQNFIQ NLSLFLCTFL KEHGQLLEKR LNLREALMEA LHYMLLVSEV EETEIFKICL EYWNHLAAEL YRESPFSTSA SPLLSGSQHF DIPPRRQLYL TVLSKVRLLM VSRMAKPEEV LVVENDQGEV VREFMKDTDS INLYKNMRET LVYLTHLDYV DTEIIMTKKL QNQVNGTEWS WKNLNTLCWA IGSISGAMHE EDEKRFLVTV IKDLLGLCEQ KRGKDNKAII ASNIMYIVGQ YPRFLRAHWK FLKTVVNKLF EFMHETHDGV QDMACDTFIK IAQKCRRHFV QVQVGEVMPF IDEILNNINT IICDLQPQQV HTFYEAVGYM IGAQTDQTVQ EHLIEKYMLL PNQVWDSIIQ QATKNVDILK DPETVKQLGS ILKTNVRACK AVGHPFVIQL GRIYLDMLNV YKCLSENISA AIQANGEMVT KQPLIRSMRT VKRETLKLIS GWVSRSNDPQ MVAENFVPPL LDAVLIDYQR NVPAAREPEV LSTMAIIVNK LGGHITAEIP QIFDAVFECT LNMINKDFEE YPEHRTNFFL LLQAVNSHCF PAFLAIPPAQ FKLVLDSIIW AFKHTMRNVA DTGLQILFTL LQNVAQEEAA AQSFYQTYFC DILQHIFSVV TDTSHTAGLT MHASILAYMF NLVEEGKIST PLNPGNPVNN QMFIQDYVAN LLKSAFPHLQ DAQVKLFVTG LFSLNQDIPA FKEHLRDFLV QIKEFAGEDT SDLFLEERET ALRQAQEEKH KLQMSVPGIL NPHEIPEEMC D |
| 要素 #63 | タイプ: protein / 記述: GTP-binding nuclear protein Ran / 分子量: 24.42 / 分子数: 1 / 由来: Homo sapiens / 参照: UniProt: P62826 配列: MAAQGEPQVQ FKLVLVGDGG TGKTTFVKRH LTGEFEKKYV ATLGVEVHPL VFHTNRGPIK FNVWDTAGQE KFGGLRDGYY IQAQCAIIMF DVTSRVTYKN VPNWHRDLVR VCENIPIVLC GNKVDIKDRK VKAKSIVFHR KKNLQYYDIS AKSNYNFEKP FLWLARKLIG ...配列: MAAQGEPQVQ FKLVLVGDGG TGKTTFVKRH LTGEFEKKYV ATLGVEVHPL VFHTNRGPIK FNVWDTAGQE KFGGLRDGYY IQAQCAIIMF DVTSRVTYKN VPNWHRDLVR VCENIPIVLC GNKVDIKDRK VKAKSIVFHR KKNLQYYDIS AKSNYNFEKP FLWLARKLIG DPNLEFVAMP ALAPPEVVMD PALAAQYEHD LEVAQTTALP DEDDDL |
-実験情報
| ビーム | 設備名称:  DORIS III X33 DORIS III X33  / 地域: Hamburg / 国: Germany / 地域: Hamburg / 国: Germany  / 線源: X-ray synchrotron / 線源: X-ray synchrotron | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 検出器 | 名称: Pilatus 2M | ||||||||||||||||||||||||
| スキャン |
| ||||||||||||||||||||||||
| 結果 | カーブのタイプ: single_conc /
|
 ムービー
ムービー コントローラー
コントローラー


 SASDAK4
SASDAK4