+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7mzz | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | AUGbeta - FAM150A - ALKL1 60-128 | ||||||
 要素 要素 | ALK and LTK ligand 1 | ||||||
 キーワード キーワード | CYTOKINE / Anaplastic lymphoma kinase (ALK) activating ligand / FAM150B / ALKL2 77-152 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of ERK5 cascade / receptor signaling protein tyrosine kinase activator activity / transmembrane receptor protein tyrosine kinase activator activity / Signaling by LTK / Signaling by ALK / cell surface receptor protein tyrosine kinase signaling pathway / cytokine activity / positive regulation of neuron projection development / receptor tyrosine kinase binding / positive regulation of ERK1 and ERK2 cascade ...positive regulation of ERK5 cascade / receptor signaling protein tyrosine kinase activator activity / transmembrane receptor protein tyrosine kinase activator activity / Signaling by LTK / Signaling by ALK / cell surface receptor protein tyrosine kinase signaling pathway / cytokine activity / positive regulation of neuron projection development / receptor tyrosine kinase binding / positive regulation of ERK1 and ERK2 cascade / extracellular space / extracellular region / plasma membrane 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / molecular dynamics | ||||||
 データ登録者 データ登録者 | Rossi, P. / Sowaileh, M. / Reshetnyak, A.V. / Kalodimos, C.G. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Mechanism for the activation of the anaplastic lymphoma kinase receptor. 著者: Andrey V Reshetnyak / Paolo Rossi / Alexander G Myasnikov / Munia Sowaileh / Jyotidarsini Mohanty / Amanda Nourse / Darcie J Miller / Irit Lax / Joseph Schlessinger / Charalampos G Kalodimos /  要旨: Anaplastic lymphoma kinase (ALK) is a receptor tyrosine kinase (RTK) that regulates important functions in the central nervous system. The ALK gene is a hotspot for chromosomal translocation events ...Anaplastic lymphoma kinase (ALK) is a receptor tyrosine kinase (RTK) that regulates important functions in the central nervous system. The ALK gene is a hotspot for chromosomal translocation events that result in several fusion proteins that cause a variety of human malignancies. Somatic and germline gain-of-function mutations in ALK were identified in paediatric neuroblastoma. ALK is composed of an extracellular region (ECR), a single transmembrane helix and an intracellular tyrosine kinase domain. ALK is activated by the binding of ALKAL1 and ALKAL2 ligands to its ECR, but the lack of structural information for the ALK-ECR or for ALKAL ligands has limited our understanding of ALK activation. Here we used cryo-electron microscopy, nuclear magnetic resonance and X-ray crystallography to determine the atomic details of human ALK dimerization and activation by ALKAL1 and ALKAL2. Our data reveal a mechanism of RTK activation that allows dimerization by either dimeric (ALKAL2) or monomeric (ALKAL1) ligands. This mechanism is underpinned by an unusual architecture of the receptor-ligand complex. The ALK-ECR undergoes a pronounced ligand-induced rearrangement and adopts an orientation parallel to the membrane surface. This orientation is further stabilized by an interaction between the ligand and the membrane. Our findings highlight the diversity in RTK oligomerization and activation mechanisms. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7mzz.cif.gz 7mzz.cif.gz | 512.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7mzz.ent.gz pdb7mzz.ent.gz | 435.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7mzz.json.gz 7mzz.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  7mzz_validation.pdf.gz 7mzz_validation.pdf.gz | 536.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  7mzz_full_validation.pdf.gz 7mzz_full_validation.pdf.gz | 795.8 KB | 表示 | |
| XML形式データ |  7mzz_validation.xml.gz 7mzz_validation.xml.gz | 40.8 KB | 表示 | |
| CIF形式データ |  7mzz_validation.cif.gz 7mzz_validation.cif.gz | 56.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mz/7mzz https://data.pdbj.org/pub/pdb/validation_reports/mz/7mzz ftp://data.pdbj.org/pub/pdb/validation_reports/mz/7mzz ftp://data.pdbj.org/pub/pdb/validation_reports/mz/7mzz | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7mzwC  7mzxC  7mzyC  7n00C C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
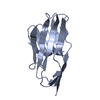
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 8187.402 Da / 分子数: 1 / 断片: residues 60-129 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: ALKAL1, FAM150A, UNQ9433/PRO34745 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: ALKAL1, FAM150A, UNQ9433/PRO34745 / 発現宿主:  |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||
| 試料状態 | イオン強度: 170 mM / Label: conditions_1 / pH: 7 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: molecular dynamics / ソフトェア番号: 1 / 詳細: restrained MD in explicit H2O bath | ||||||||||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: target function / 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

 light scattering
light scattering