+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7k6h | ||||||
|---|---|---|---|---|---|---|---|
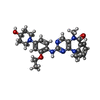
| タイトル | Crystal structure of the first bromodomain (BD1) of human BRD4 bound to XMD8-92 | ||||||
 要素 要素 | Bromodomain-containing protein 4 | ||||||
 キーワード キーワード | GENE REGULATION / BET / ERK5 / dual BRD-kinase inhibitor | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RNA polymerase II C-terminal domain binding / P-TEFb complex binding / negative regulation of DNA damage checkpoint / histone H4 reader activity / host-mediated suppression of viral transcription / positive regulation of G2/M transition of mitotic cell cycle / positive regulation of T-helper 17 cell lineage commitment / : / RNA polymerase II CTD heptapeptide repeat kinase activity / condensed nuclear chromosome ...RNA polymerase II C-terminal domain binding / P-TEFb complex binding / negative regulation of DNA damage checkpoint / histone H4 reader activity / host-mediated suppression of viral transcription / positive regulation of G2/M transition of mitotic cell cycle / positive regulation of T-helper 17 cell lineage commitment / : / RNA polymerase II CTD heptapeptide repeat kinase activity / condensed nuclear chromosome / transcription coregulator activity / positive regulation of transcription elongation by RNA polymerase II / p53 binding / chromosome / regulation of inflammatory response / histone binding / Potential therapeutics for SARS / transcription coactivator activity / positive regulation of canonical NF-kappaB signal transduction / transcription cis-regulatory region binding / chromatin remodeling / protein serine/threonine kinase activity / DNA damage response / chromatin binding / regulation of transcription by RNA polymerase II / chromatin / positive regulation of DNA-templated transcription / enzyme binding / positive regulation of transcription by RNA polymerase II / nucleoplasm / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Karim, M.R. / Zhu, J.Y. / Schonbrunn, E. | ||||||
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2021 ジャーナル: J.Med.Chem. / 年: 2021タイトル: Differential BET Bromodomain Inhibition by Dihydropteridinone and Pyrimidodiazepinone Kinase Inhibitors. 著者: Karim, R.M. / Bikowitz, M.J. / Chan, A. / Zhu, J.Y. / Grassie, D. / Becker, A. / Berndt, N. / Gunawan, S. / Lawrence, N.J. / Schonbrunn, E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7k6h.cif.gz 7k6h.cif.gz | 117 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7k6h.ent.gz pdb7k6h.ent.gz | 87 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7k6h.json.gz 7k6h.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/k6/7k6h https://data.pdbj.org/pub/pdb/validation_reports/k6/7k6h ftp://data.pdbj.org/pub/pdb/validation_reports/k6/7k6h ftp://data.pdbj.org/pub/pdb/validation_reports/k6/7k6h | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  5v67C  5vboC  5vbpC  5vbqC  5vbrC  7bjyC  7k6gSC  7ko0C  7l6dC  7l72C  7l73C  7l9gC  7l9jC  7l9kC  7l9lC  7lahC  7laiC  7lajC  7lakC  7lauC  7layC  7lazC  7lb4C  7lbtC  7lejC  7lekC  7lelC  7lemC S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 15099.380 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: BRD4, HUNK1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: BRD4, HUNK1 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-4WG / |
| #3: 水 | ChemComp-HOH / |
| 研究の焦点であるリガンドがあるか | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.06 Å3/Da / 溶媒含有率: 40.38 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 詳細: 0.2 M Ammonium acetate, 0.1 M Sodium citrate tribasic dihydrate pH 5.6, 30% w/v Polyethylene glycol 4,000 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 22-ID / 波長: 1 Å / ビームライン: 22-ID / 波長: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: MARMOSAIC 300 mm CCD / 検出器: CCD / 日付: 2017年4月5日 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 1 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 1.5→28.635 Å / Num. obs: 20618 / % possible obs: 99.9 % / 冗長度: 6.999 % / Biso Wilson estimate: 16.68 Å2 / CC1/2: 0.991 / Rmerge(I) obs: 0.107 / Rrim(I) all: 0.116 / Χ2: 0.985 / Net I/σ(I): 18.6 / Num. measured all: 144304 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1
|
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 7K6G 解像度: 1.5→28.635 Å / SU ML: 0.11 / 交差検証法: THROUGHOUT / σ(F): 1.4 / 位相誤差: 18.8 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 145.86 Å2 / Biso mean: 29.6633 Å2 / Biso min: 10.2 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.5→28.635 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / % reflection obs: 100 %
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj