+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6z8m | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of [NiFeSe] hydrogenase G491S variant from Desulfovibrio vulgaris Hildenborough pressurized with Oxygen gas - structure G491S-O2 | |||||||||
 要素 要素 | (Periplasmic [NiFeSe] hydrogenase, ...) x 2 | |||||||||
 キーワード キーワード | OXIDOREDUCTASE / Hydrogenase / Selenium / gas channels / high-pressure derivatization | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cytochrome-c3 hydrogenase / cytochrome-c3 hydrogenase activity / ferredoxin hydrogenase complex / [Ni-Fe] hydrogenase complex / ferredoxin hydrogenase activity / anaerobic respiration / cell envelope / 3 iron, 4 sulfur cluster binding / nickel cation binding / 4 iron, 4 sulfur cluster binding ...cytochrome-c3 hydrogenase / cytochrome-c3 hydrogenase activity / ferredoxin hydrogenase complex / [Ni-Fe] hydrogenase complex / ferredoxin hydrogenase activity / anaerobic respiration / cell envelope / 3 iron, 4 sulfur cluster binding / nickel cation binding / 4 iron, 4 sulfur cluster binding / periplasmic space / electron transfer activity / metal ion binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Desulfovibrio vulgaris (バクテリア) Desulfovibrio vulgaris (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.02 Å 分子置換 / 解像度: 1.02 Å | |||||||||
 データ登録者 データ登録者 | Zacarias, S. / Temporao, A. / Carpentier, P. / van der Linden, P. / Pereira, I.A.C. / Matias, P.M. | |||||||||
| 資金援助 |  ポルトガル, 2件 ポルトガル, 2件
| |||||||||
 引用 引用 |  ジャーナル: J.Biol.Inorg.Chem. / 年: 2020 ジャーナル: J.Biol.Inorg.Chem. / 年: 2020タイトル: Exploring the gas access routes in a [NiFeSe] hydrogenase using crystals pressurized with krypton and oxygen. 著者: Zacarias, S. / Temporao, A. / Carpentier, P. / van der Linden, P. / Pereira, I.A.C. / Matias, P.M. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6z8m.cif.gz 6z8m.cif.gz | 548.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6z8m.ent.gz pdb6z8m.ent.gz | 373.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6z8m.json.gz 6z8m.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  6z8m_validation.pdf.gz 6z8m_validation.pdf.gz | 502.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  6z8m_full_validation.pdf.gz 6z8m_full_validation.pdf.gz | 503.9 KB | 表示 | |
| XML形式データ |  6z8m_validation.xml.gz 6z8m_validation.xml.gz | 34.3 KB | 表示 | |
| CIF形式データ |  6z8m_validation.cif.gz 6z8m_validation.cif.gz | 53.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/z8/6z8m https://data.pdbj.org/pub/pdb/validation_reports/z8/6z8m ftp://data.pdbj.org/pub/pdb/validation_reports/z8/6z8m ftp://data.pdbj.org/pub/pdb/validation_reports/z8/6z8m | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6z7rC  6z8jC  6z8oC  6z9gC  6z9oC  6za1C  5jskS S: 精密化の開始モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 実験データセット #1 | データ参照:  10.5281/zenodo.3873088 / データの種類: diffraction image data 10.5281/zenodo.3873088 / データの種類: diffraction image data |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||
| 単位格子 |
|
- 要素
要素
-Periplasmic [NiFeSe] hydrogenase, ... , 2種, 2分子 AB
| #1: タンパク質 | 分子量: 30261.568 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 / 遺伝子: hysB, DVU_1917 発現宿主:  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア)参照: UniProt: Q72AS4, ferredoxin hydrogenase |
|---|---|
| #2: タンパク質 | 分子量: 53413.086 Da / 分子数: 1 / Mutation: G491S / 由来タイプ: 組換発現 由来: (組換発現)  Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア) Desulfovibrio vulgaris (strain Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303) (バクテリア)株: Hildenborough / ATCC 29579 / DSM 644 / NCIMB 8303 / 遺伝子: hysA, DVU_1918 発現宿主:  Desulfovibrio vulgaris str. Hildenborough (バクテリア) Desulfovibrio vulgaris str. Hildenborough (バクテリア)参照: UniProt: Q72AS3, ferredoxin hydrogenase |
-非ポリマー , 10種, 698分子 
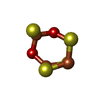

















| #3: 化合物 | | #4: 化合物 | ChemComp-6ML / | #5: 化合物 | #6: 化合物 | ChemComp-FCO / | #7: 化合物 | ChemComp-NI / | #8: 化合物 | ChemComp-FE2 / | #9: 化合物 | ChemComp-H2S / | #10: 化合物 | ChemComp-CL / | #11: 化合物 | #12: 水 | ChemComp-HOH / | |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.79 Å3/Da / 溶媒含有率: 31.3 % / 解説: thin plate |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.6 / 詳細: 20% PEG 1500 (w/v) and 0.1 mM Tris-HCl pH 7.6 |
-データ収集
| 回折 | 平均測定温度: 100 K / Serial crystal experiment: N |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID29 / 波長: 0.9184 Å / ビームライン: ID29 / 波長: 0.9184 Å |
| 検出器 | タイプ: DECTRIS PILATUS3 6M / 検出器: PIXEL / 日付: 2017年10月2日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9184 Å / 相対比: 1 |
| 反射 | 解像度: 1.02→53.33 Å / Num. obs: 331872 / % possible obs: 94.6 % / 冗長度: 2.7 % / Biso Wilson estimate: 8.3 Å2 / CC1/2: 0.998 / Rmerge(I) obs: 0.058 / Rpim(I) all: 0.04 / Net I/σ(I): 7.6 |
| 反射 シェル | 解像度: 1.02→1.04 Å / 冗長度: 2.7 % / Rmerge(I) obs: 1.098 / Mean I/σ(I) obs: 0.7 / Num. unique obs: 16250 / CC1/2: 0.349 / Rpim(I) all: 0.781 / % possible all: 93 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 5JSK 解像度: 1.02→53.33 Å / SU ML: 0.1179 / 交差検証法: FREE R-VALUE / σ(F): 0.92 / 位相誤差: 16.3404 立体化学のターゲット値: GeoStd + Monomer Library + CDL v1.2 詳細: UNIQUE REFLECTIONS (NON-ANOMALOUS) USED IN REFINEMENT: 331545. Hydrogen atoms were included in calculated positions, solvent molecules were added manually in COOT using 2|Fo|-|Fc| and |Fo|- ...詳細: UNIQUE REFLECTIONS (NON-ANOMALOUS) USED IN REFINEMENT: 331545. Hydrogen atoms were included in calculated positions, solvent molecules were added manually in COOT using 2|Fo|-|Fc| and |Fo|-|Fc| maps and occupancy factors were refined for disordered residues and the O2 molecules; anomalous dispersion parameters were refined for the selenium atom in Sec 489; anisotropic atomic displacement parameters were refined for all non-hydrogen and non-solvent atoms.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 13.78 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.02→53.33 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj





















