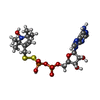
| 登録情報 | データベース: PDB / ID: 6mi6
|
|---|
| タイトル | STRUCTURE OF CHEA DOMAIN P4 IN COMPLEX WITH AN ADP ANALOG |
|---|
 要素 要素 | Chemotaxis protein CheA |
|---|
 キーワード キーワード | SIGNALING PROTEIN / bacterial chemotaxis / histidine kinase / Thermotoga maritima |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
histidine kinase / phosphorelay sensor kinase activity / chemotaxis / protein domain specific binding / ATP binding / cytoplasm類似検索 - 分子機能 Chemotaxis protein CheA, P2 response regulator-binding / Chemotaxis protein CheA, P2 response regulator-binding domain superfamily / P2 response regulator binding domain / Histidine kinase CheA-like, homodimeric domain / CheY-binding domain of CheA / Histidine kinase CheA-like, homodimeric domain superfamily / Signal transducing histidine kinase, homodimeric domain / Signal transducing histidine kinase, homodimeric domain / : / CheW-like domain profile. ...Chemotaxis protein CheA, P2 response regulator-binding / Chemotaxis protein CheA, P2 response regulator-binding domain superfamily / P2 response regulator binding domain / Histidine kinase CheA-like, homodimeric domain / CheY-binding domain of CheA / Histidine kinase CheA-like, homodimeric domain superfamily / Signal transducing histidine kinase, homodimeric domain / Signal transducing histidine kinase, homodimeric domain / : / CheW-like domain profile. / CheW-like domain / CheW-like domain superfamily / CheW-like domain / Two component signalling adaptor domain / Histidine Phosphotransfer domain / Hpt domain / Histidine-containing phosphotransfer (HPt) domain profile. / Signal transduction histidine kinase, phosphotransfer (Hpt) domain / HPT domain superfamily / Signal transduction histidine kinase-related protein, C-terminal / Signal transduction histidine kinase, dimerisation/phosphoacceptor domain superfamily / Histidine kinase domain / Histidine kinase domain profile. / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase superfamily類似検索 - ドメイン・相同性 ADENOSINE-5'-DIPHOSPHATE / Chem-JSJ / Chemotaxis protein CheA類似検索 - 構成要素 |
|---|
| 生物種 |   Thermotoga maritima (バクテリア) Thermotoga maritima (バクテリア) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.95 Å シンクロトロン / 解像度: 2.95 Å |
|---|
 データ登録者 データ登録者 | Crane, B.R. / Muok, A.R. / Chua, T.K. / Le, H. |
|---|
 引用 引用 |  ジャーナル: Appl.Magn.Reson. / 年: 2018 ジャーナル: Appl.Magn.Reson. / 年: 2018
タイトル: Nucleotide Spin Labeling for ESR Spectroscopy of ATP-Binding Proteins.
著者: Muok, A.R. / Chua, T.K. / Le, H. / Crane, B.R. |
|---|
| 履歴 | | 登録 | 2018年9月19日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2018年12月5日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2019年11月13日 | Group: Database references / カテゴリ: citation / citation_author
Item: _citation.pdbx_database_id_PubMed / _citation.title / _citation_author.identifier_ORCID |
|---|
| 改定 1.2 | 2024年12月25日 | Group: Advisory / Data collection ...Advisory / Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_validate_close_contact / struct_conn
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Thermotoga maritima (バクテリア)
Thermotoga maritima (バクテリア) X線回折 /
X線回折 /  シンクロトロン / 解像度: 2.95 Å
シンクロトロン / 解像度: 2.95 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Appl.Magn.Reson. / 年: 2018
ジャーナル: Appl.Magn.Reson. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6mi6.cif.gz
6mi6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6mi6.ent.gz
pdb6mi6.ent.gz PDB形式
PDB形式 6mi6.json.gz
6mi6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6mi6_validation.pdf.gz
6mi6_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6mi6_full_validation.pdf.gz
6mi6_full_validation.pdf.gz 6mi6_validation.xml.gz
6mi6_validation.xml.gz 6mi6_validation.cif.gz
6mi6_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/mi/6mi6
https://data.pdbj.org/pub/pdb/validation_reports/mi/6mi6 ftp://data.pdbj.org/pub/pdb/validation_reports/mi/6mi6
ftp://data.pdbj.org/pub/pdb/validation_reports/mi/6mi6 リンク
リンク 集合体
集合体




 要素
要素
 Thermotoga maritima (strain ATCC 43589 / MSB8 / DSM 3109 / JCM 10099) (バクテリア)
Thermotoga maritima (strain ATCC 43589 / MSB8 / DSM 3109 / JCM 10099) (バクテリア)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 24-ID-E / 波長: 0.9722 Å
/ ビームライン: 24-ID-E / 波長: 0.9722 Å 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj