| 登録情報 | データベース: PDB / ID: 6avj
|
|---|
| タイトル | Crystal structure of human Mitochondrial inner NEET protein (MiNT)/CISD3 |
|---|
 要素 要素 | CDGSH iron-sulfur domain-containing protein 3, mitochondrial |
|---|
 キーワード キーワード | METAL BINDING PROTEIN / NEET-fold / Iron-Sulfur cluster / Mitochondria |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
: / 2 iron, 2 sulfur cluster binding / mitochondrion / metal ion binding類似検索 - 分子機能 : / Iron-binding zinc finger CDGSH type / Iron-binding zinc finger, CDGSH type / MitoNEET, CDGSH iron-sulfur domain / CDGSH-type zinc finger. Function unknown.類似検索 - ドメイン・相同性 FE2/S2 (INORGANIC) CLUSTER / CDGSH iron-sulfur domain-containing protein 3, mitochondrial類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å |
|---|
 データ登録者 データ登録者 | Lipper, C.H. / Karmi, O. / Sohn, Y.S. / Darash-Yahana, M. / Lammert, H. / Song, L. / Liu, A. / Mittler, R. / Nechushtai, R. / Onuchic, J.N. / Jennings, P.A. |
|---|
| 資金援助 |  米国, 米国,  イスラエル, 6件 イスラエル, 6件 | 組織 | 認可番号 | 国 |
|---|
| National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | GM101467 |  米国 米国 | | National Science Foundation (NSF, United States) | PHY-1427654 |  米国 米国 | | National Science Foundation (NSF, United States) | CHE-1614101 |  米国 米国 | | Israel Science Foundation | ISF-865/13 |  イスラエル イスラエル | | National Science Foundation (NSF, United States) | MCB-1613462 |  米国 米国 | | United States - Israel Binational Science Foundation (BSF) | 2015831 |  イスラエル イスラエル |
|
|---|
 引用 引用 |  ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2018 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2018
タイトル: Structure of the human monomeric NEET protein MiNT and its role in regulating iron and reactive oxygen species in cancer cells.
著者: Lipper, C.H. / Karmi, O. / Sohn, Y.S. / Darash-Yahana, M. / Lammert, H. / Song, L. / Liu, A. / Mittler, R. / Nechushtai, R. / Onuchic, J.N. / Jennings, P.A. |
|---|
| 履歴 | | 登録 | 2017年9月2日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2017年12月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2018年1月3日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_abbrev / _citation.pdbx_database_id_PubMed / _citation.title |
|---|
| 改定 1.2 | 2018年1月17日 | Group: Database references / カテゴリ: citation
Item: _citation.journal_volume / _citation.page_first ..._citation.journal_volume / _citation.page_first / _citation.page_last / _citation.year |
|---|
| 改定 1.3 | 2019年11月27日 | Group: Author supporting evidence / カテゴリ: pdbx_audit_support / Item: _pdbx_audit_support.funding_organization |
|---|
| 改定 1.4 | 2023年10月4日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 米国,
米国,  イスラエル, 6件
イスラエル, 6件  引用
引用 ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2018
ジャーナル: Proc. Natl. Acad. Sci. U.S.A. / 年: 2018 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6avj.cif.gz
6avj.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6avj.ent.gz
pdb6avj.ent.gz PDB形式
PDB形式 6avj.json.gz
6avj.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 6avj_validation.pdf.gz
6avj_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 6avj_full_validation.pdf.gz
6avj_full_validation.pdf.gz 6avj_validation.xml.gz
6avj_validation.xml.gz 6avj_validation.cif.gz
6avj_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/av/6avj
https://data.pdbj.org/pub/pdb/validation_reports/av/6avj ftp://data.pdbj.org/pub/pdb/validation_reports/av/6avj
ftp://data.pdbj.org/pub/pdb/validation_reports/av/6avj
 リンク
リンク 集合体
集合体


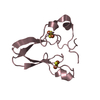
 要素
要素 Homo sapiens (ヒト) / 遺伝子: CISD3 / プラスミド: pET24a / 細胞株 (発現宿主): BL21-CodonPlus (DE3)-RIL / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CISD3 / プラスミド: pET24a / 細胞株 (発現宿主): BL21-CodonPlus (DE3)-RIL / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54178 Å
回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54178 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー









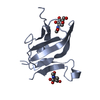
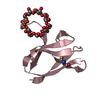
 PDBj
PDBj










