+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5x1x | ||||||
|---|---|---|---|---|---|---|---|
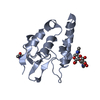
| タイトル | Solution NMR Structure of DNA Mismatch Repair Protein MutT (Family Nudix Hydrolase) from Methicillin Resistant Staphylococcus aureus 252 | ||||||
 要素 要素 | Mutator mutT protein | ||||||
 キーワード キーワード | HYDROLASE / divalent ion (Mg2+ / Ca2+) binding protein / adenosine monophosphate (AMP) binding protein | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報8-oxo-dGTP diphosphatase / 8-oxo-7,8-dihydrodeoxyguanosine triphosphate pyrophosphatase activity / DNA repair 類似検索 - 分子機能 | ||||||
| 生物種 |  Staphylococcus aureus subsp. aureus MN8 (黄色ブドウ球菌) Staphylococcus aureus subsp. aureus MN8 (黄色ブドウ球菌) | ||||||
| 手法 | 溶液NMR / distance geometry | ||||||
 データ登録者 データ登録者 | Wahab, A. / Durreshahwar, S. / Schwalbe, H. / Richter, C. / Choudhary, M.I. | ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Solution NMR Structure of DNA Mismatch Repair Protein MutT (Family Nudix Hydrolase) from Methicillin Resistant Staphylococcus aureus 252 著者: Wahab, A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5x1x.cif.gz 5x1x.cif.gz | 942.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5x1x.ent.gz pdb5x1x.ent.gz | 813.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5x1x.json.gz 5x1x.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5x1x_validation.pdf.gz 5x1x_validation.pdf.gz | 467.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5x1x_full_validation.pdf.gz 5x1x_full_validation.pdf.gz | 773.8 KB | 表示 | |
| XML形式データ |  5x1x_validation.xml.gz 5x1x_validation.xml.gz | 97.6 KB | 表示 | |
| CIF形式データ |  5x1x_validation.cif.gz 5x1x_validation.cif.gz | 89.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/x1/5x1x https://data.pdbj.org/pub/pdb/validation_reports/x1/5x1x ftp://data.pdbj.org/pub/pdb/validation_reports/x1/5x1x ftp://data.pdbj.org/pub/pdb/validation_reports/x1/5x1x | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 14914.342 Da / 分子数: 1 / 由来タイプ: 組換発現 / 詳細: methicillin resistant 由来: (組換発現)  Staphylococcus aureus subsp. aureus MN8 (黄色ブドウ球菌) Staphylococcus aureus subsp. aureus MN8 (黄色ブドウ球菌)Cell: Bacterial / 遺伝子: HMPREF0769_10658 / Variant: MRSA252 / プラスミド: pSpeedET 詳細 (発現宿主): Bacterial expression vector, arabinose or T7 induced expression, adds N-terminal MGSDKIHHHHHHENLYFQG tag; kanamycin resistance; PIPE cloning Cell (発現宿主): bacterial / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | タイプ: solution 内容: 1 mM [U-99% 13C; U-99% 15N] DNA Mismatch Repair Protein MutT from Nudix Hydrolase Family, 95% H2O/5% D2O 詳細: The protein sample was made using 25 mM sodium phosphate buffer, pH 6.9, 100 mM NaCl, 1 mM DTT, 0.15 mM DSS, 5% D2O Label: 15N, 13C labeled / 溶媒系: 95% H2O/5% D2O |
|---|---|
| 試料 | 濃度: 1 mM 構成要素: DNA Mismatch Repair Protein MutT from Nudix Hydrolase Family Isotopic labeling: [U-99% 13C; U-99% 15N] |
| 試料状態 | 詳細: The protein sample was made using 25 mM sodium phosphate buffer, pH 6.9, 100 mM NaCl, 1 mM DTT, 0.15 mM DSS, 5% D2O イオン強度: 25 mM sodium phosphate, 100 mM NaCl mM / Label: conditions-1 / pH: 6.9 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: distance geometry / ソフトェア番号: 8 | ||||||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: all calculated structures submitted 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

 HSQC
HSQC