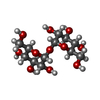
登録情報 データベース : PDB / ID : 5vf2タイトル scFv 2D10 re-refined as a complex with trehalose replacing the original alpha-1,6-mannobiose scFv 2D10 キーワード / 機能・相同性 / / / / 生物種 Mus musculus (ハツカネズミ)手法 / / / 解像度 : 1.55 Å データ登録者 Porebski, P.J. / Wlodawer, A. / Dauter, Z. / Minor, W. / Stanfield, R. / Jaskolski, M. / Pozharski, E. / Weichenberger, C.X. / Rupp, B. 資金援助 組織 認可番号 国 National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) U01HG008424, R01GM117080, R01GM117325 Austrian Science Fund P28395-B26 Polish National Science Centre 2013/10/M/NZ1/00251
ジャーナル : Chemistryselect / 年 : 2016タイトル : Antibodies Can Exploit Molecular Crowding to Bind New Antigens at Noncanonical Paratope Positions著者 : Vashisht, S. / Kumar, A. / Kaur, K.J. / Salunke, D.M. 履歴 登録 2017年4月6日 登録サイト / 処理サイト 改定 1.0 2017年12月6日 Provider / タイプ 改定 1.1 2018年2月14日 Group / カテゴリ Item _citation.journal_volume / _citation.page_first ... _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.title / _citation.year 改定 1.2 2018年3月21日 Group / カテゴリ / Item 改定 1.3 2020年1月1日 Group / カテゴリ / Item 改定 2.0 2020年7月29日 Group Atomic model / Data collection ... Atomic model / Data collection / Derived calculations / Non-polymer description / Structure summary カテゴリ atom_site / chem_comp ... atom_site / chem_comp / entity / entity_name_com / pdbx_branch_scheme / pdbx_chem_comp_identifier / pdbx_entity_branch / pdbx_entity_branch_descriptor / pdbx_entity_branch_link / pdbx_entity_branch_list / pdbx_entity_nonpoly / pdbx_molecule_features / pdbx_nonpoly_scheme / pdbx_struct_conn_angle / pdbx_struct_special_symmetry / struct_conn / struct_conn_type / struct_site / struct_site_gen Item _atom_site.B_iso_or_equiv / _atom_site.Cartn_x ... _atom_site.B_iso_or_equiv / _atom_site.Cartn_x / _atom_site.Cartn_y / _atom_site.Cartn_z / _atom_site.auth_asym_id / _atom_site.auth_atom_id / _atom_site.auth_comp_id / _atom_site.auth_seq_id / _atom_site.label_atom_id / _atom_site.label_comp_id / _atom_site.type_symbol / _chem_comp.formula / _chem_comp.formula_weight / _chem_comp.id / _chem_comp.mon_nstd_flag / _chem_comp.name / _chem_comp.pdbx_synonyms / _chem_comp.type / _entity.formula_weight / _entity.pdbx_description / _entity.src_method / _entity.type / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_symmetry / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_symmetry / _pdbx_struct_conn_angle.value / _pdbx_struct_special_symmetry.auth_asym_id / _pdbx_struct_special_symmetry.auth_comp_id / _pdbx_struct_special_symmetry.auth_seq_id / _pdbx_struct_special_symmetry.label_comp_id 解説 / Provider / タイプ 改定 2.1 2022年4月13日 Group / Structure summaryカテゴリ audit_author / chem_comp ... audit_author / chem_comp / citation_author / database_2 Item _audit_author.identifier_ORCID / _chem_comp.pdbx_synonyms ... _audit_author.identifier_ORCID / _chem_comp.pdbx_synonyms / _citation_author.identifier_ORCID / _database_2.pdbx_DOI / _database_2.pdbx_database_accession 改定 2.2 2023年10月4日 Group / Refinement descriptionカテゴリ / chem_comp_bond / pdbx_initial_refinement_model改定 2.3 2024年10月23日 Group カテゴリ / pdbx_modification_feature
すべて表示 表示を減らす Remark 0 THIS ENTRY 5VF2 REFLECTS AN ALTERNATIVE MODELING OF THE ORIGINAL DATA IN 5I4F, DETERMINED BY S. ... THIS ENTRY 5VF2 REFLECTS AN ALTERNATIVE MODELING OF THE ORIGINAL DATA IN 5I4F, DETERMINED BY S.VASHISHT,A.KUMAR,K.J.KAUR,D.M.SALUNKE
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 1.55 Å
フーリエ合成 / 解像度: 1.55 Å  データ登録者
データ登録者 米国,
米国,  オーストリア,
オーストリア,  ポーランド, 3件
ポーランド, 3件  引用
引用 ジャーナル: Chemistryselect / 年: 2016
ジャーナル: Chemistryselect / 年: 2016 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 5vf2.cif.gz
5vf2.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb5vf2.ent.gz
pdb5vf2.ent.gz PDB形式
PDB形式 5vf2.json.gz
5vf2.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 5vf2_validation.pdf.gz
5vf2_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 5vf2_full_validation.pdf.gz
5vf2_full_validation.pdf.gz 5vf2_validation.xml.gz
5vf2_validation.xml.gz 5vf2_validation.cif.gz
5vf2_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/vf/5vf2
https://data.pdbj.org/pub/pdb/validation_reports/vf/5vf2 ftp://data.pdbj.org/pub/pdb/validation_reports/vf/5vf2
ftp://data.pdbj.org/pub/pdb/validation_reports/vf/5vf2 リンク
リンク 集合体
集合体
 要素
要素








 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: BM14 / 波長: 0.97856 Å
/ ビームライン: BM14 / 波長: 0.97856 Å 解析
解析 フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー



















 PDBj
PDBj