+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5oe1 | |||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Im polyamide in complex with 5'CGATGTACATCG3'- hairpin polyamides studies | |||||||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / Minor groove binders / hairpin poyamides | 機能・相同性 | Chem-9T8 / 1-methylimidazole-2-carboxylic acid / GAMMA-AMINO-BUTANOIC ACID / 4-AMINO-(1-METHYLIMIDAZOLE)-2-CARBOXYLIC ACID / 4-AMINO-(1-METHYLPYRROLE)-2-CARBOXYLIC ACID / DNA / DNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト)手法 | 溶液NMR / molecular dynamics / matrix relaxation |  データ登録者 データ登録者Padroni, G. / Parkinson, J. / Burley, G.A. |  引用 引用 ジャーナル: Nucleic Acids Res. / 年: 2018 ジャーナル: Nucleic Acids Res. / 年: 2018タイトル: Structural basis of DNA duplex distortion induced by thiazole-containing hairpin polyamides. 著者: Padroni, G. / Parkinson, J.A. / Fox, K.R. / Burley, G.A. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5oe1.cif.gz 5oe1.cif.gz | 193 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5oe1.ent.gz pdb5oe1.ent.gz | 155.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5oe1.json.gz 5oe1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5oe1_validation.pdf.gz 5oe1_validation.pdf.gz | 493 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5oe1_full_validation.pdf.gz 5oe1_full_validation.pdf.gz | 689 KB | 表示 | |
| XML形式データ |  5oe1_validation.xml.gz 5oe1_validation.xml.gz | 27.8 KB | 表示 | |
| CIF形式データ |  5oe1_validation.cif.gz 5oe1_validation.cif.gz | 36.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/oe/5oe1 https://data.pdbj.org/pub/pdb/validation_reports/oe/5oe1 ftp://data.pdbj.org/pub/pdb/validation_reports/oe/5oe1 ftp://data.pdbj.org/pub/pdb/validation_reports/oe/5oe1 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5oczC 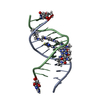 5odfC  5odmC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
-DNA鎖 , 1種, 2分子 AB
| #1: DNA鎖 | 分子量: 3582.424 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
-非ポリマー , 5種, 10分子 





| #2: 化合物 | ChemComp-9TK / | ||||||
|---|---|---|---|---|---|---|---|
| #3: 化合物 | ChemComp-PYB / #4: 化合物 | ChemComp-ABU / | #5: 化合物 | ChemComp-IMT / | #6: 化合物 | ChemComp-9T8 / | |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||
| 試料状態 | イオン強度: 100 phosphate mM / Label: Standard / pH: 7.4 / 圧: 1 atm / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker AVANCE / 製造業者: Bruker / モデル: AVANCE / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 |
| ||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: clustering / 計算したコンフォーマーの数: 2000 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj






































 scanning transmission electron microscopy
scanning transmission electron microscopy




