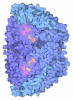
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5g0r | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | METHYL-COENZYME M REDUCTASE I FROM METHANOTHERMOBACTER MARBURGENSIS EXPOSED TO 3-NITROOXYPROPANOL | |||||||||
 要素 要素 | (METHYL-COENZYME M REDUCTASE I SUBUNIT ...) x 3 | |||||||||
 キーワード キーワード | TRANSFERASE / METHYL-COENZYMEM / METHYL-COENZYMEM REDUCTASE EXPOSED TO 3-NITROOXYPROPANOL / POSTTRANSLATIONAL MODIFICATION / BINDING SITES / CATALYSIS / COENZYMES / DISULFIDES / HYDROGEN / LIGANDS / MESNA / METALLOPORPHYRINS / METHANE / METHANOBACTERIUM / NICKEL / OXIDATION-REDUCTION / OXDOREDUCTASES / PHOSPHOTHREONINE / THIOGLYCINE / 3- NITROOXYPROPANOL / INHIBITOR / GREENHOUSE GAS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報coenzyme-B sulfoethylthiotransferase / coenzyme-B sulfoethylthiotransferase activity / methanogenesis / metal ion binding / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.25 Å 分子置換 / 解像度: 1.25 Å | |||||||||
 データ登録者 データ登録者 | Wagner, T. | |||||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2016タイトル: Mode of Action Uncovered for the Specific Reduction of Methane Emissions from Ruminants by the Small Molecule 3-Nitrooxypropanol. 著者: Duin, E.C. / Wagner, T. / Shima, S. / Prakash, D. / Cronin, B. / Yanez-Ruiz, D.R. / Duval, S. / Rumbeli, R. / Stemmler, R.T. / Thauer, R.K. / Kindermann, M. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5g0r.cif.gz 5g0r.cif.gz | 1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5g0r.ent.gz pdb5g0r.ent.gz | 846.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5g0r.json.gz 5g0r.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  5g0r_validation.pdf.gz 5g0r_validation.pdf.gz | 1.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  5g0r_full_validation.pdf.gz 5g0r_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  5g0r_validation.xml.gz 5g0r_validation.xml.gz | 111.8 KB | 表示 | |
| CIF形式データ |  5g0r_validation.cif.gz 5g0r_validation.cif.gz | 171.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/g0/5g0r https://data.pdbj.org/pub/pdb/validation_reports/g0/5g0r ftp://data.pdbj.org/pub/pdb/validation_reports/g0/5g0r ftp://data.pdbj.org/pub/pdb/validation_reports/g0/5g0r | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5a0yS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||
| 単位格子 |
| ||||||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-METHYL-COENZYME M REDUCTASE I SUBUNIT ... , 3種, 6分子 ADBECF
| #1: タンパク質 | 分子量: 60637.648 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM) 由来: (天然)   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌)株: MARBURG 参照: UniProt: P11558, coenzyme-B sulfoethylthiotransferase #2: タンパク質 | 分子量: 47279.676 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM) 由来: (天然)   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌)株: MARBURG 参照: UniProt: P11560, coenzyme-B sulfoethylthiotransferase #3: タンパク質 | 分子量: 28797.234 Da / 分子数: 2 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM) 由来: (天然)   METHANOTHERMOBACTER MARBURGENSIS (古細菌) METHANOTHERMOBACTER MARBURGENSIS (古細菌)株: MARBURG 参照: UniProt: P11562, coenzyme-B sulfoethylthiotransferase |
|---|
-非ポリマー , 7種, 2638分子 
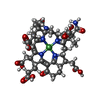











| #4: 化合物 | ChemComp-MG / #5: 化合物 | #6: 化合物 | #7: 化合物 | #8: 化合物 | ChemComp-K / | #9: 化合物 | #10: 水 | ChemComp-HOH / | |
|---|
-詳細
| 配列の詳細 | RESIDUE 450 IS A DIDEHYDROA |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.18 Å3/Da / 溶媒含有率: 43.6 % / 解説: NONE |
|---|---|
| 結晶化 | 温度: 281 K / 手法: 蒸気拡散法, シッティングドロップ法 詳細: 25-28 % V/V PEG 400, 300 MM MGCL2, 200 MM NACL AND 100 MM HEPES PH 7.5 CRYSTAL GROWTH AT 8 DEGREE CELSIUS UNDER AEROBIC CONDITION |
-データ収集
| 回折 | 平均測定温度: 77 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SLS SLS  / ビームライン: X10SA / 波長: 0.97128 / ビームライン: X10SA / 波長: 0.97128 |
| 検出器 | タイプ: DECTRIS PILATUS 6M / 検出器: PIXEL / 日付: 2014年3月9日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.97128 Å / 相対比: 1 |
| 反射 | 解像度: 1.25→38.79 Å / Num. obs: 633330 / % possible obs: 98.5 % / Observed criterion σ(I): 4 / 冗長度: 4.6 % / Biso Wilson estimate: 9.86 Å2 / Rmerge(I) obs: 0.05 / Net I/σ(I): 14.4 |
| 反射 シェル | 解像度: 1.25→1.32 Å / 冗長度: 4.4 % / Rmerge(I) obs: 0.27 / Mean I/σ(I) obs: 4.4 / % possible all: 98.1 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 5A0Y 解像度: 1.25→38.795 Å / SU ML: 0.07 / σ(F): 1.34 / 位相誤差: 10.8 / 立体化学のターゲット値: ML 詳細: CONSIDERING THE RESOLUTION HYDROGEN REFINEMENT MODEL HAS BEEN USED WITH THE RIDING MODE THE INHIBITOR 3-NITROOXYPROPANOL HAS POSSIBLY REDUCED THE NICKEL CENTER AND IS NOW HYDROLYSED IN THE ...詳細: CONSIDERING THE RESOLUTION HYDROGEN REFINEMENT MODEL HAS BEEN USED WITH THE RIDING MODE THE INHIBITOR 3-NITROOXYPROPANOL HAS POSSIBLY REDUCED THE NICKEL CENTER AND IS NOW HYDROLYSED IN THE ACTIVE SITE. SINCE THERE IS A LOW PARTIAL OCCUPANCY OF THE BROKEN COMPOUND (NITRITE AND 1-3-PROPANEDIOL) IN THE ACTIVE SITE, WATER MOLECULE HAVE BEEN PLACED INSTEAD.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 15.54 Å2 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.25→38.795 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj