+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4xk8 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of plant photosystem I-LHCI super-complex at 2.8 angstrom resolution | |||||||||
 要素 要素 |
| |||||||||
 キーワード キーワード | PHOTOSYNTHESIS / photosystem i / plant LHCI-PSI supercomplex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報response to low light intensity stimulus / plastoglobule / response to high light intensity / chloroplast thylakoid / photosynthesis, light harvesting in photosystem I / thylakoid / chloroplast thylakoid lumen / photosynthesis, light harvesting / chloroplast envelope / photosystem I reaction center ...response to low light intensity stimulus / plastoglobule / response to high light intensity / chloroplast thylakoid / photosynthesis, light harvesting in photosystem I / thylakoid / chloroplast thylakoid lumen / photosynthesis, light harvesting / chloroplast envelope / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / photosystem II / chlorophyll binding / chloroplast thylakoid membrane / photosynthesis / response to cold / chloroplast / phosphoprotein binding / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / protein domain specific binding / mRNA binding / magnesium ion binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pisum sativum (マメ科) Pisum sativum (マメ科) | |||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 2.8 Å シンクロトロン / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Suga, M. / Qin, X. / Kuang, T. / Shen, J.R. | |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2015 ジャーナル: Science / 年: 2015タイトル: Photosynthesis. Structural basis for energy transfer pathways in the plant PSI-LHCI supercomplex. 著者: Xiaochun Qin / Michihiro Suga / Tingyun Kuang / Jian-Ren Shen /   要旨: Photosynthesis converts solar energy to chemical energy by means of two large pigment-protein complexes: photosystem I (PSI) and photosystem II (PSII). In higher plants, the PSI core is surrounded by ...Photosynthesis converts solar energy to chemical energy by means of two large pigment-protein complexes: photosystem I (PSI) and photosystem II (PSII). In higher plants, the PSI core is surrounded by a large light-harvesting complex I (LHCI) that captures sunlight and transfers the excitation energy to the core with extremely high efficiency. We report the structure of PSI-LHCI, a 600-kilodalton membrane protein supercomplex, from Pisum sativum (pea) at a resolution of 2.8 angstroms. The structure reveals the detailed arrangement of pigments and other cofactors—especially within LHCI—as well as numerous specific interactions between the PSI core and LHCI. These results provide a firm structural basis for our understanding on the energy transfer and photoprotection mechanisms within the PSI-LHCI supercomplex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4xk8.cif.gz 4xk8.cif.gz | 3.4 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4xk8.ent.gz pdb4xk8.ent.gz | 3 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4xk8.json.gz 4xk8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4xk8_validation.pdf.gz 4xk8_validation.pdf.gz | 92.6 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4xk8_full_validation.pdf.gz 4xk8_full_validation.pdf.gz | 97 MB | 表示 | |
| XML形式データ |  4xk8_validation.xml.gz 4xk8_validation.xml.gz | 450.6 KB | 表示 | |
| CIF形式データ |  4xk8_validation.cif.gz 4xk8_validation.cif.gz | 513.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xk/4xk8 https://data.pdbj.org/pub/pdb/validation_reports/xk/4xk8 ftp://data.pdbj.org/pub/pdb/validation_reports/xk/4xk8 ftp://data.pdbj.org/pub/pdb/validation_reports/xk/4xk8 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-Photosystem I P700 chlorophyll a apoprotein ... , 2種, 4分子 AaBb
| #1: タンパク質 | 分子量: 82408.281 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: P05310*PLUS Pisum sativum (マメ科) / 参照: UniProt: P05310*PLUS#2: タンパク質 | 分子量: 82466.820 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: P05311*PLUS Pisum sativum (マメ科) / 参照: UniProt: P05311*PLUS |
|---|
-タンパク質 , 3種, 6分子 CcDd27
| #3: タンパク質 | 分子量: 8860.276 Da / 分子数: 2 / Fragment: UNP residues 2-81 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: P10793, photosystem I Pisum sativum (マメ科) / 参照: UniProt: P10793, photosystem I#4: タンパク質 | 分子量: 15853.142 Da / 分子数: 2 / Fragment: UNP residues 63-203 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: I1NGD2, UniProt: A5Z2K3*PLUS Pisum sativum (マメ科) / 参照: UniProt: I1NGD2, UniProt: A5Z2K3*PLUS#14: タンパク質 | 分子量: 22693.666 Da / 分子数: 2 / Fragment: UNP residues 62-267 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: Q41038 Pisum sativum (マメ科) / 参照: UniProt: Q41038 |
|---|
-Putative uncharacterized ... , 3種, 6分子 EeHhLl
| #5: タンパク質 | 分子量: 7299.219 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: E1C9K6*PLUS Pisum sativum (マメ科) / 参照: UniProt: E1C9K6*PLUS#8: タンパク質 | 分子量: 9639.886 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: C6T221*PLUS Pisum sativum (マメ科) / 参照: UniProt: C6T221*PLUS#12: タンパク質 | 分子量: 16130.484 Da / 分子数: 2 / Fragment: UNP residues 4-156 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: E1C9L1 Pisum sativum (マメ科) / 参照: UniProt: E1C9L1 |
|---|
-Photosystem I reaction center subunit ... , 5種, 10分子 FfGgIiJjKk
| #6: タンパク質 | 分子量: 17009.771 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: P12355*PLUS Pisum sativum (マメ科) / 参照: UniProt: P12355*PLUS#7: タンパク質 | 分子量: 10460.736 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: P12357*PLUS Pisum sativum (マメ科) / 参照: UniProt: P12357*PLUS#9: タンパク質・ペプチド | 分子量: 3296.041 Da / 分子数: 2 / Fragment: UNP residues 2-31 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: P17227 Pisum sativum (マメ科) / 参照: UniProt: P17227#10: タンパク質・ペプチド | 分子量: 4402.227 Da / 分子数: 2 / Fragment: UNP residues 1-39 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: A4GGC6 Pisum sativum (マメ科) / 参照: UniProt: A4GGC6#11: タンパク質 | 分子量: 8462.847 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: E1C9L3 Pisum sativum (マメ科) / 参照: UniProt: E1C9L3 |
|---|
-Chlorophyll a-b binding protein ... , 3種, 6分子 163849
| #13: タンパク質 | 分子量: 21354.418 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: Q01667*PLUS Pisum sativum (マメ科) / 参照: UniProt: Q01667*PLUS#15: タンパク質 | 分子量: 23725.020 Da / 分子数: 2 / Fragment: UNP residues 55-272 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: Q32904 Pisum sativum (マメ科) / 参照: UniProt: Q32904#16: タンパク質 | 分子量: 21719.670 Da / 分子数: 2 / 由来タイプ: 合成 / 由来: (合成)  Pisum sativum (マメ科) / 参照: UniProt: Q9SQL2*PLUS Pisum sativum (マメ科) / 参照: UniProt: Q9SQL2*PLUS |
|---|
-糖 , 3種, 9分子 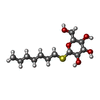




| #22: 糖 | ChemComp-HTG / #23: 糖 | ChemComp-LMT / | #24: 糖 | |
|---|
-非ポリマー , 10種, 590分子 


















| #17: 化合物 | ChemComp-CLA / #18: 化合物 | ChemComp-PQN / #19: 化合物 | ChemComp-LHG / #20: 化合物 | ChemComp-BCR / #21: 化合物 | ChemComp-SF4 / #25: 化合物 | ChemComp-LMG / #26: 化合物 | ChemComp-CHL / #27: 化合物 | ChemComp-LUT / ( #28: 化合物 | ChemComp-XAT / ( #29: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.92 Å3/Da / 溶媒含有率: 68.59 % |
|---|---|
| 結晶化 | 温度: 285 K / 手法: 蒸気拡散法 / 詳細: PEG |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SPring-8 SPring-8  / ビームライン: BL41XU / 波長: 0.9 Å / ビームライン: BL41XU / 波長: 0.9 Å |
| 検出器 | タイプ: RAYONIX MX225HE / 検出器: CCD / 日付: 2013年12月11日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.9 Å / 相対比: 1 |
| 反射 | 解像度: 2.8→50 Å / Num. obs: 268282 / % possible obs: 99.8 % / 冗長度: 7.5 % / Net I/σ(I): 7 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 2.8→49.147 Å / SU ML: 0.4 / 交差検証法: FREE R-VALUE / σ(F): 1.34 / 位相誤差: 27.53 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.8→49.147 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Origin x: -28.0049 Å / Origin y: -0.025 Å / Origin z: 145.821 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ | Selection details: all |
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





















