[English] 日本語
 Yorodumi
Yorodumi- PDB-4y28: The structure of plant photosystem I super-complex at 2.8 angstro... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4y28 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | The structure of plant photosystem I super-complex at 2.8 angstrom resolution. | |||||||||
 Components Components |
| |||||||||
 Keywords Keywords | PHOTOSYNTHESIS / light harvesting / antenna / reaction center | |||||||||
| Function / homology |  Function and homology information Function and homology informationphotosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting / chloroplast thylakoid lumen / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / photosystem II / chlorophyll binding / chloroplast thylakoid membrane ...photosynthesis, light harvesting in photosystem I / photosynthesis, light harvesting / chloroplast thylakoid lumen / photosystem I reaction center / photosystem I / photosynthetic electron transport in photosystem I / photosystem I / photosystem II / chlorophyll binding / chloroplast thylakoid membrane / photosynthesis / chloroplast / 4 iron, 4 sulfur cluster binding / electron transfer activity / oxidoreductase activity / protein domain specific binding / magnesium ion binding / metal ion binding Similarity search - Function | |||||||||
| Biological species |  Pisum sativum (garden pea) Pisum sativum (garden pea) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  SAD / Resolution: 2.8 Å SAD / Resolution: 2.8 Å | |||||||||
 Authors Authors | Mazor, Y. / Brovikov, A. / Nelson, N. | |||||||||
 Citation Citation |  Journal: Elife / Year: 2015 Journal: Elife / Year: 2015Title: The structure of plant photosystem I super-complex at 2.8 angstrom resolution. Authors: Mazor, Y. / Borovikova, A. / Nelson, N. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4y28.cif.gz 4y28.cif.gz | 1.8 MB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4y28.ent.gz pdb4y28.ent.gz | 1.5 MB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4y28.json.gz 4y28.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/y2/4y28 https://data.pdbj.org/pub/pdb/validation_reports/y2/4y28 ftp://data.pdbj.org/pub/pdb/validation_reports/y2/4y28 ftp://data.pdbj.org/pub/pdb/validation_reports/y2/4y28 | HTTPS FTP |
|---|
-Related structure data
| Similar structure data |
|---|
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Photosystem I P700 chlorophyll a apoprotein ... , 2 types, 2 molecules AB
| #1: Protein | Mass: 84328.562 Da / Num. of mol.: 1 / Fragment: UNP residues 17-758 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: P05310, photosystem I Pisum sativum (garden pea) / References: UniProt: P05310, photosystem I |
|---|---|
| #2: Protein | Mass: 82503.039 Da / Num. of mol.: 1 / Fragment: UNP residues 2-733 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: P05311, photosystem I Pisum sativum (garden pea) / References: UniProt: P05311, photosystem I |
-Photosystem I reaction center subunit ... , 7 types, 7 molecules IJFDEHK
| #3: Protein/peptide | Mass: 3296.041 Da / Num. of mol.: 1 / Fragment: UNP residues 3-32 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: P17227 Pisum sativum (garden pea) / References: UniProt: P17227 |
|---|---|
| #4: Protein/peptide | Mass: 4733.593 Da / Num. of mol.: 1 / Fragment: UNP residues 2-42 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: D5MAL3 Pisum sativum (garden pea) / References: UniProt: D5MAL3 |
| #5: Protein | Mass: 17256.016 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: A0A0M3KL12*PLUS Pisum sativum (garden pea) / References: UniProt: A0A0M3KL12*PLUS |
| #9: Protein | Mass: 16419.852 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: E1C9K8*PLUS Pisum sativum (garden pea) / References: UniProt: E1C9K8*PLUS |
| #10: Protein | Mass: 7509.449 Da / Num. of mol.: 1 / Fragment: UNP residues 1-64 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: E1C9K6*PLUS Pisum sativum (garden pea) / References: UniProt: E1C9K6*PLUS |
| #11: Protein | Mass: 9692.042 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: A0A0M3KL10*PLUS Pisum sativum (garden pea) / References: UniProt: A0A0M3KL10*PLUS |
| #12: Protein | Mass: 13320.767 Da / Num. of mol.: 1 / Fragment: UNP residues 2-80 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: A0A0M3KL11*PLUS Pisum sativum (garden pea) / References: UniProt: A0A0M3KL11*PLUS |
-Protein , 5 types, 5 molecules GLC21
| #6: Protein | Mass: 10439.700 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: A0A0M3KL13*PLUS Pisum sativum (garden pea) / References: UniProt: A0A0M3KL13*PLUS |
|---|---|
| #7: Protein | Mass: 17781.295 Da / Num. of mol.: 1 / Fragment: UNP residues 3-161 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: E1C9L1*PLUS Pisum sativum (garden pea) / References: UniProt: E1C9L1*PLUS |
| #8: Protein | Mass: 8991.474 Da / Num. of mol.: 1 / Fragment: UNP residues 2-81 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: P10793, photosystem I Pisum sativum (garden pea) / References: UniProt: P10793, photosystem I |
| #13: Protein | Mass: 28911.773 Da / Num. of mol.: 1 / Fragment: UNP residues 59-264 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: Q41038 Pisum sativum (garden pea) / References: UniProt: Q41038 |
| #15: Protein | Mass: 22383.611 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: E1C9L2*PLUS Pisum sativum (garden pea) / References: UniProt: E1C9L2*PLUS |
-Chlorophyll a-b binding protein ... , 2 types, 2 molecules 43
| #14: Protein | Mass: 27297.074 Da / Num. of mol.: 1 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: Q9SQL2 Pisum sativum (garden pea) / References: UniProt: Q9SQL2 |
|---|---|
| #16: Protein | Mass: 29634.801 Da / Num. of mol.: 1 / Fragment: UNP residues 58-272 / Source method: isolated from a natural source / Source: (natural)  Pisum sativum (garden pea) / References: UniProt: Q32904 Pisum sativum (garden pea) / References: UniProt: Q32904 |
-Sugars , 2 types, 3 molecules 
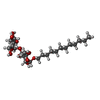

| #25: Sugar | ChemComp-DGD / |
|---|---|
| #26: Sugar |
-Non-polymers , 11 types, 206 molecules 




















| #17: Chemical | ChemComp-CL0 / | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #18: Chemical | ChemComp-CLA / #19: Chemical | #20: Chemical | #21: Chemical | ChemComp-LHG / #22: Chemical | ChemComp-BCR / #23: Chemical | ChemComp-LMG / #24: Chemical | ChemComp-CA / | #27: Chemical | ChemComp-LUT / ( #28: Chemical | ChemComp-CHL / #29: Chemical | ChemComp-ZEX / ( | |
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal grow | Temperature: 277 K / Method: vapor diffusion, sitting drop / pH: 8.75 Details: 30mM Tris-Hcl pH8.75, 50mM KH2PO4, 0.03% Octyl Glucose Neopentyl Glycol, 14% PEG400, 2mM glutathione PH range: 8-8.75 |
|---|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SLS SLS  / Beamline: X06DA / Wavelength: 1.74 Å / Beamline: X06DA / Wavelength: 1.74 Å |
| Detector | Type: DECTRIS PILATUS 6M / Detector: PIXEL / Date: Sep 20, 2014 |
| Radiation | Monochromator: Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.74 Å / Relative weight: 1 |
| Reflection | Resolution: 2.8→74.1 Å / Num. all: 200218 / Num. obs: 200218 / % possible obs: 99.99 % / Redundancy: 74.1 % / Rmerge(I) obs: 0.26 / Net I/σ(I): 13.4 |
| Reflection shell | Resolution: 2.8→2.85 Å / Redundancy: 33.4 % / Rmerge(I) obs: 1.035 / Mean I/σ(I) obs: 1.4 / % possible all: 98.4 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  SAD / Resolution: 2.8→48.646 Å / SU ML: 0.59 / Cross valid method: FREE R-VALUE / σ(F): 1.9 / Phase error: 32.33 / Stereochemistry target values: MLHL SAD / Resolution: 2.8→48.646 Å / SU ML: 0.59 / Cross valid method: FREE R-VALUE / σ(F): 1.9 / Phase error: 32.33 / Stereochemistry target values: MLHL
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.9 Å / VDW probe radii: 1.11 Å / Solvent model: FLAT BULK SOLVENT MODEL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 584.44 Å2 / Biso mean: 98.1935 Å2 / Biso min: 20 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: final / Resolution: 2.8→48.646 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 28 / % reflection obs: 100 %
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Origin x: 14.9211 Å / Origin y: 7.8237 Å / Origin z: 64.3676 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group | Selection details: ALL and not water |
 Movie
Movie Controller
Controller












 PDBj
PDBj






















