+ データを開く
データを開く
- 基本情報
基本情報
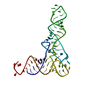
| 登録情報 | データベース: PDB / ID: 4v72 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli 70S-fMetVal-tRNAVal-tRNAfMet complex in hybrid pre-translocation state (pre4) | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | RIBOSOME / cryo-EM refinement / tRNA / translocation intermediate | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報stringent response / ornithine decarboxylase inhibitor activity / transcription antitermination factor activity, RNA binding / misfolded RNA binding / Group I intron splicing / RNA folding / transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis ...stringent response / ornithine decarboxylase inhibitor activity / transcription antitermination factor activity, RNA binding / misfolded RNA binding / Group I intron splicing / RNA folding / transcriptional attenuation / endoribonuclease inhibitor activity / RNA-binding transcription regulator activity / positive regulation of ribosome biogenesis / negative regulation of cytoplasmic translation / four-way junction DNA binding / translational termination / DnaA-L2 complex / translation repressor activity / negative regulation of DNA-templated DNA replication initiation / negative regulation of translational initiation / regulation of mRNA stability / mRNA regulatory element binding translation repressor activity / ribosome assembly / assembly of large subunit precursor of preribosome / positive regulation of RNA splicing / transcription elongation factor complex / cytosolic ribosome assembly / regulation of DNA-templated transcription elongation / DNA endonuclease activity / response to reactive oxygen species / transcription antitermination / regulation of cell growth / DNA-templated transcription termination / maintenance of translational fidelity / response to radiation / mRNA 5'-UTR binding / ribosomal small subunit biogenesis / small ribosomal subunit rRNA binding / large ribosomal subunit / ribosome biogenesis / ribosome binding / regulation of translation / ribosomal small subunit assembly / small ribosomal subunit / 5S rRNA binding / large ribosomal subunit rRNA binding / transferase activity / cytosolic small ribosomal subunit / ribosomal large subunit assembly / cytoplasmic translation / cytosolic large ribosomal subunit / tRNA binding / molecular adaptor activity / negative regulation of translation / rRNA binding / ribosome / structural constituent of ribosome / translation / response to antibiotic / negative regulation of DNA-templated transcription / mRNA binding / DNA binding / RNA binding / zinc ion binding / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  | ||||||||||||
| 手法 | 電子顕微鏡法 / 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 13 Å | ||||||||||||
 データ登録者 データ登録者 | Blau, C. / Bock, L.V. / Schroder, G.F. / Davydov, I. / Fischer, N. / Stark, H. / Rodnina, M.V. / Vaiana, A.C. / Grubmuller, H. | ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2013 ジャーナル: Nat Struct Mol Biol / 年: 2013タイトル: Energy barriers and driving forces in tRNA translocation through the ribosome. 著者: Lars V Bock / Christian Blau / Gunnar F Schröder / Iakov I Davydov / Niels Fischer / Holger Stark / Marina V Rodnina / Andrea C Vaiana / Helmut Grubmüller /  要旨: During protein synthesis, tRNAs move from the ribosome's aminoacyl to peptidyl to exit sites. Here we investigate conformational motions during spontaneous translocation, using molecular dynamics ...During protein synthesis, tRNAs move from the ribosome's aminoacyl to peptidyl to exit sites. Here we investigate conformational motions during spontaneous translocation, using molecular dynamics simulations of 13 intermediate-translocation-state models obtained by combining Escherichia coli ribosome crystal structures with cryo-EM data. Resolving fast transitions between states, we find that tRNA motions govern the transition rates within the pre- and post-translocation states. Intersubunit rotations and L1-stalk motion exhibit fast intrinsic submicrosecond dynamics. The L1 stalk drives the tRNA from the peptidyl site and links intersubunit rotation to translocation. Displacement of tRNAs is controlled by 'sliding' and 'stepping' mechanisms involving conserved L16, L5 and L1 residues, thus ensuring binding to the ribosome despite large-scale tRNA movement. Our results complement structural data with a time axis, intrinsic transition rates and molecular forces, revealing correlated functional motions inaccessible by other means. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4v72.cif.gz 4v72.cif.gz | 3.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4v72.ent.gz pdb4v72.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  4v72.json.gz 4v72.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4v72_validation.pdf.gz 4v72_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4v72_full_validation.pdf.gz 4v72_full_validation.pdf.gz | 2.2 MB | 表示 | |
| XML形式データ |  4v72_validation.xml.gz 4v72_validation.xml.gz | 263.3 KB | 表示 | |
| CIF形式データ |  4v72_validation.cif.gz 4v72_validation.cif.gz | 449.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/v7/4v72 https://data.pdbj.org/pub/pdb/validation_reports/v7/4v72 ftp://data.pdbj.org/pub/pdb/validation_reports/v7/4v72 ftp://data.pdbj.org/pub/pdb/validation_reports/v7/4v72 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1719M  2472C  2473C  2474C  2475C  4v6yC  4v6zC  4v70C  4v71C  4v73C  4v74C  4v75C  4v76C  4v77C  4v78C  4v79C  4v7aC C: 同じ文献を引用 ( M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-30S ribosomal protein ... , 20種, 20分子 ABACADAEAFAGAHAIAJAKALAMANAOAPAQARASATAU
| #1: タンパク質 | 分子量: 24277.992 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #2: タンパク質 | 分子量: 23207.990 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #3: タンパク質 | 分子量: 23514.199 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #4: タンパク質 | 分子量: 15828.328 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #5: タンパク質 | 分子量: 11667.380 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #6: タンパク質 | 分子量: 16788.453 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #7: タンパク質 | 分子量: 14146.557 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #8: タンパク質 | 分子量: 14580.919 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #9: タンパク質 | 分子量: 11221.034 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #10: タンパク質 | 分子量: 12513.237 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #11: タンパク質 | 分子量: 13768.157 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #12: タンパク質 | 分子量: 12657.843 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #13: タンパク質 | 分子量: 11606.560 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #14: タンパク質 | 分子量: 10290.816 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #15: タンパク質 | 分子量: 9063.426 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #16: タンパク質 | 分子量: 9287.992 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #17: タンパク質 | 分子量: 6490.523 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #18: タンパク質 | 分子量: 9081.672 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #19: タンパク質 | 分子量: 9532.228 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #20: タンパク質 | 分子量: 6091.125 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
-RNA鎖 , 6種, 6分子 AAA1A2A3BABB
| #21: RNA鎖 | 分子量: 496892.375 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
|---|---|
| #22: RNA鎖 | 分子量: 24617.768 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #23: RNA鎖 | 分子量: 4693.746 Da / 分子数: 1 / 由来タイプ: 合成 / 詳細: mRNA |
| #24: RNA鎖 | 分子量: 24848.918 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #54: RNA鎖 | 分子量: 941306.188 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
| #55: RNA鎖 | 分子量: 38177.762 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  |
+50S ribosomal protein ... , 30種, 30分子 BCBDBEBFBGBHBIBJBKBLBMBNBOBPBQBRBSBTBUBVBWBXBYBZB0B1B2B3B4B5
-非ポリマー , 2種, 2分子 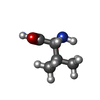
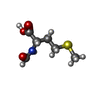

| #57: 化合物 | ChemComp-VAL / |
|---|---|
| #58: 化合物 | ChemComp-FME / |
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: E. coli 70S-fMetVal-tRNAVal-tRNAfMet complex / タイプ: RIBOSOME |
|---|---|
| 緩衝液 | 名称: 50 mM Tris-HCl, 70 mM NH4Cl, 30 mM KCl, 7 mM MgCl2, 0.6 mM spermine, 0.4 mM spermidine pH: 7.5 詳細: 50 mM Tris-HCl, 70 mM NH4Cl, 30 mM KCl, 7 mM MgCl2, 0.6 mM spermine, 0.4 mM spermidine |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE / 湿度: 75 % 詳細: Manual blotting for about 2 seconds prior to plunging into liquid ethane (custom-built CEVS vitrification instrument with dew point temperature adjusted to 18 degrees C) 手法: Manual blotting for about 2 seconds Time resolved state: Samples were vitrified at different time points along the reaction coordinate (1, 2, 5 and 20 minutes after addition of deacylated tRNAfMet to 70S-fMetVal-tRNAVal complexes). |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 顕微鏡 | モデル: FEI/PHILIPS CM200FEG / 日付: 2008年5月11日 詳細: Objective lens astigmatism was corrected at 200,000 times magnification |
|---|---|
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 160 kV / 照射モード: SPOT SCAN FIELD EMISSION GUN / 加速電圧: 160 kV / 照射モード: SPOT SCAN |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 161000 X / 倍率(補正後): 162740 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 500 nm / Cs: 2 mm |
| 試料ホルダ | 試料ホルダーモデル: GATAN LIQUID NITROGEN / 資料ホルダタイプ: Eucentric / 温度: 77 K |
| 撮影 | 電子線照射量: 20 e/Å2 フィルム・検出器のモデル: GENERIC TVIPS (4k x 4k) 詳細: 4k CCD camera (TVIPS) |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTF補正 | 詳細: local | |||||||||||||||
| 対称性 | 点対称性: C1 (非対称) | |||||||||||||||
| 3次元再構成 | 手法: Projection matching / 解像度: 13 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 8375 / ピクセルサイズ(公称値): 2.8 Å / ピクセルサイズ(実測値): 2.8 Å 詳細: Final maps were calculated from 13 datasets acquired at different time points, computationally sorted into distinct substates. 対称性のタイプ: POINT | |||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL / Target criteria: Cross-correlation coefficient 詳細: METHOD--Cross-correlation gradient based REFINEMENT PROTOCOL--flexible fitting, dynamic elastic network | |||||||||||||||
| 原子モデル構築 |
|
 ムービー
ムービー コントローラー
コントローラー































 PDBj
PDBj