| 登録情報 | データベース: PDB / ID: 4my6
|
|---|
| タイトル | EnaH-EVH1 in complex with peptidomimetic low-molecular weight inhibitor Ac-[2-Cl-F]-[ProM-2]-[ProM-1]-OH |
|---|
 要素 要素 | Protein enabled homolog |
|---|
 キーワード キーワード | Cell adhesion/inhibitor / MOLECULAR RECOGNITION / ACTIN DYNAMICS / Cell adhesion-inhibitor complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
postsynaptic cytoskeleton organization / actin polymerization-dependent cell motility / profilin binding / Signaling by ROBO receptors / actin polymerization or depolymerization / WW domain binding / Generation of second messenger molecules / axon guidance / filopodium / GABA-ergic synapse ...postsynaptic cytoskeleton organization / actin polymerization-dependent cell motility / profilin binding / Signaling by ROBO receptors / actin polymerization or depolymerization / WW domain binding / Generation of second messenger molecules / axon guidance / filopodium / GABA-ergic synapse / SH3 domain binding / lamellipodium / actin binding / cytoskeleton / postsynapse / focal adhesion / plasma membrane / cytosol類似検索 - 分子機能 VASP tetramerisation / VASP tetramerisation domain superfamily / VASP tetramerisation domain / WH1/EVH1 domain / WH1 domain / WH1 domain profile. / WASP homology region 1 / Pleckstrin-homology domain (PH domain)/Phosphotyrosine-binding domain (PTB) / PH-domain like / PH-like domain superfamily ...VASP tetramerisation / VASP tetramerisation domain superfamily / VASP tetramerisation domain / WH1/EVH1 domain / WH1 domain / WH1 domain profile. / WASP homology region 1 / Pleckstrin-homology domain (PH domain)/Phosphotyrosine-binding domain (PTB) / PH-domain like / PH-like domain superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 (3AR,5AS,8S,10AS)-1-[(3S,6R,8AS)-1'-[(2S)-2-ACETAMIDO-3-(2-CHLOROPHENYL)PROPANOYL]-5-OXIDANYLIDENE-SPIRO[1,2,3,8A-TETRAHYDROINDOLIZINE-6,2'-PYRROLIDINE]-3-YL]CARBONYL-10-OXIDANYLIDENE-2,3,3A,5A,8,10A-HEXAHYDRODIPYRROLO[3,2-B:3',1'-F]AZEPINE-8-CARBOXYLIC ACID / : / BROMIDE ION / Protein enabled homolog類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.7 Å 分子置換 / 解像度: 1.7 Å |
|---|
 データ登録者 データ登録者 | Barone, M. / Roske, Y. / Kuehne, R. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2015 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2015
タイトル: A modular toolkit to inhibit proline-rich motif-mediated protein-protein interactions.
著者: Opitz, R. / Muller, M. / Reuter, C. / Barone, M. / Soicke, A. / Roske, Y. / Piotukh, K. / Huy, P. / Beerbaum, M. / Wiesner, B. / Beyermann, M. / Schmieder, P. / Freund, C. / Volkmer, R. / ...著者: Opitz, R. / Muller, M. / Reuter, C. / Barone, M. / Soicke, A. / Roske, Y. / Piotukh, K. / Huy, P. / Beerbaum, M. / Wiesner, B. / Beyermann, M. / Schmieder, P. / Freund, C. / Volkmer, R. / Oschkinat, H. / Schmalz, H.G. / Kuhne, R. |
|---|
| 履歴 | | 登録 | 2013年9月27日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年10月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2015年4月15日 | Group: Database references |
|---|
| 改定 1.2 | 2015年4月22日 | Group: Database references |
|---|
| 改定 1.3 | 2015年5月6日 | Group: Database references |
|---|
| 改定 1.4 | 2015年7月22日 | Group: Structure summary |
|---|
| 改定 1.5 | 2023年9月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.7 Å
分子置換 / 解像度: 1.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2015
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2015 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4my6.cif.gz
4my6.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4my6.ent.gz
pdb4my6.ent.gz PDB形式
PDB形式 4my6.json.gz
4my6.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4my6_validation.pdf.gz
4my6_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4my6_full_validation.pdf.gz
4my6_full_validation.pdf.gz 4my6_validation.xml.gz
4my6_validation.xml.gz 4my6_validation.cif.gz
4my6_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/my/4my6
https://data.pdbj.org/pub/pdb/validation_reports/my/4my6 ftp://data.pdbj.org/pub/pdb/validation_reports/my/4my6
ftp://data.pdbj.org/pub/pdb/validation_reports/my/4my6
 リンク
リンク 集合体
集合体
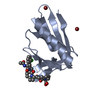

 要素
要素 Homo sapiens (ヒト) / 遺伝子: ENAH, MENA / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ENAH, MENA / 発現宿主: 

 タイプ: peptide-like, Peptide-like / クラス: 阻害剤 / 分子量: 676.159 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C35H38ClN5O7
タイプ: peptide-like, Peptide-like / クラス: 阻害剤 / 分子量: 676.159 Da / 分子数: 2 / 由来タイプ: 合成 / 式: C35H38ClN5O7 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  BESSY
BESSY  / ビームライン: 14.1 / 波長: 0.91841 Å
/ ビームライン: 14.1 / 波長: 0.91841 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー




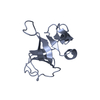





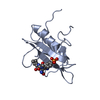
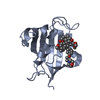

 PDBj
PDBj










