[English] 日本語
 Yorodumi
Yorodumi- PDB-3imj: Crystal Structure of the Grb2 SH2 Domain in Complex with a Cyclop... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 3imj | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of the Grb2 SH2 Domain in Complex with a Cyclopropyl-constrained Ac-pTyr-Ile-Asn-NH2 Tripeptide Mimic | ||||||
 Components Components | Growth factor receptor-bound protein 2 | ||||||
 Keywords Keywords | SIGNALING PROTEIN/PEPTIDE / ligand preorganization / peptide mimics / Golgi apparatus / Host-virus interaction / Phosphoprotein / SH2 domain / SH3 domain / Signaling protein-pseudopeptide ligand complex / SIGNALING PROTEIN-PEPTIDE COMPLEX | ||||||
| Function / homology |  Function and homology information Function and homology informationguanyl-nucleotide exchange factor adaptor activity / Grb2-EGFR complex / branching involved in labyrinthine layer morphogenesis / STAT5 Activation / Co-inhibition by BTLA / neurotrophin TRKA receptor binding / Activated NTRK2 signals through PI3K / COP9 signalosome / transmembrane receptor protein tyrosine kinase adaptor activity / MET receptor recycling ...guanyl-nucleotide exchange factor adaptor activity / Grb2-EGFR complex / branching involved in labyrinthine layer morphogenesis / STAT5 Activation / Co-inhibition by BTLA / neurotrophin TRKA receptor binding / Activated NTRK2 signals through PI3K / COP9 signalosome / transmembrane receptor protein tyrosine kinase adaptor activity / MET receptor recycling / Interleukin-15 signaling / Signaling by cytosolic FGFR1 fusion mutants / negative regulation of natural killer cell mediated cytotoxicity / MET activates PTPN11 / MET activates RAP1 and RAC1 / vesicle membrane / Signaling by LTK / CD28 dependent Vav1 pathway / Signal regulatory protein family interactions / MET activates PI3K/AKT signaling / epidermal growth factor receptor binding / Regulation of KIT signaling / natural killer cell mediated cytotoxicity / PI-3K cascade:FGFR3 / STAT5 activation downstream of FLT3 ITD mutants / PI-3K cascade:FGFR2 / PI-3K cascade:FGFR4 / PI-3K cascade:FGFR1 / endodermal cell differentiation / positive regulation of actin filament polymerization / GRB2:SOS provides linkage to MAPK signaling for Integrins / RHOU GTPase cycle / regulation of MAPK cascade / RET signaling / insulin receptor substrate binding / Interleukin-3, Interleukin-5 and GM-CSF signaling / PI3K events in ERBB2 signaling / PI3K Cascade / SOS-mediated signalling / Role of LAT2/NTAL/LAB on calcium mobilization / fibroblast growth factor receptor signaling pathway / Activated NTRK3 signals through RAS / Activated NTRK2 signals through RAS / Interleukin receptor SHC signaling / SHC1 events in ERBB4 signaling / RHO GTPases Activate WASPs and WAVEs / Signal attenuation / Signalling to RAS / GAB1 signalosome / SHC-related events triggered by IGF1R / Activated NTRK2 signals through FRS2 and FRS3 / Schwann cell development / SHC-mediated cascade:FGFR3 / MET activates RAS signaling / SHC-mediated cascade:FGFR2 / SHC-mediated cascade:FGFR4 / Signaling by PDGFRA transmembrane, juxtamembrane and kinase domain mutants / Signaling by PDGFRA extracellular domain mutants / Erythropoietin activates RAS / SHC-mediated cascade:FGFR1 / Signaling by FGFR4 in disease / ephrin receptor binding / Signaling by CSF3 (G-CSF) / FRS-mediated FGFR3 signaling / Signaling by FLT3 ITD and TKD mutants / FRS-mediated FGFR2 signaling / signal transduction in response to DNA damage / FRS-mediated FGFR4 signaling / FRS-mediated FGFR1 signaling / Signaling by FGFR3 in disease / Tie2 Signaling / phosphotyrosine residue binding / Signaling by FGFR2 in disease / myelination / GRB2 events in EGFR signaling / Signaling by FLT3 fusion proteins / SHC1 events in EGFR signaling / FLT3 Signaling / Signaling by FGFR1 in disease / EGFR Transactivation by Gastrin / FCERI mediated Ca+2 mobilization / NCAM signaling for neurite out-growth / GRB2 events in ERBB2 signaling / Downstream signal transduction / insulin-like growth factor receptor signaling pathway / Insulin receptor signalling cascade / SHC1 events in ERBB2 signaling / Constitutive Signaling by Overexpressed ERBB2 / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants / T cell activation / InlB-mediated entry of Listeria monocytogenes into host cell / cellular response to ionizing radiation / B cell receptor signaling pathway / FCGR3A-mediated phagocytosis / FCERI mediated MAPK activation / Regulation of signaling by CBL / Negative regulation of FGFR3 signaling / Negative regulation of FGFR2 signaling / Negative regulation of FGFR4 signaling Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / MOLECULAR REPLACEMENT /  molecular replacement / Resolution: 2.02 Å molecular replacement / Resolution: 2.02 Å | ||||||
 Authors Authors | Clements, J.H. | ||||||
 Citation Citation |  Journal: J.Am.Chem.Soc. / Year: 2009 Journal: J.Am.Chem.Soc. / Year: 2009Title: Thermodynamic and Structural Effects of Conformational Constraints in Protein-Ligand Interactions. Entropic Paradoxy Associated with Ligand Preorganization. Authors: Delorbe, J.E. / Clements, J.H. / Teresk, M.G. / Benfield, A.P. / Plake, H.R. / Millspaugh, L.E. / Martin, S.F. #1:  Journal: Angew.Chem.Int.Ed.Engl. / Year: 2006 Journal: Angew.Chem.Int.Ed.Engl. / Year: 2006Title: Ligand Preorganization May Be Accompanied by Entropic Penalties in Protein-Ligand Interactions Authors: Benfield, A.P. / Teresk, M.G. / Plake, H.R. / DeLorbe, J.E. / Millspaugh, L.E. / Martin, S.F. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  3imj.cif.gz 3imj.cif.gz | 61.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb3imj.ent.gz pdb3imj.ent.gz | 44.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  3imj.json.gz 3imj.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/im/3imj https://data.pdbj.org/pub/pdb/validation_reports/im/3imj ftp://data.pdbj.org/pub/pdb/validation_reports/im/3imj ftp://data.pdbj.org/pub/pdb/validation_reports/im/3imj | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  3imdC  3in7C  3in8C  3kfjC  2huwS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 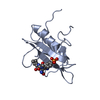
| ||||||||
| 2 | 
| ||||||||
| 3 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 13758.543 Da / Num. of mol.: 2 / Fragment: SH2 domain Source method: isolated from a genetically manipulated source Details: residues 53-163 were expressed in addition to a C-terminal 6-his tag Source: (gene. exp.)  Homo sapiens (human) / Gene: GRB2, ASH / Plasmid: pQE-60 / Production host: Homo sapiens (human) / Gene: GRB2, ASH / Plasmid: pQE-60 / Production host:  #2: Chemical | #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.02 Å3/Da / Density % sol: 39.17 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, hanging drop / pH: 4.6 Details: Ligand in lyophilized powder form was dissolved in a 11.2 mg/mL solution of Grb2 SH2 in water such to give a protein/ligand molar ratio of 2:1. 4 uL of this solution was mixed with 3 uL of 0. ...Details: Ligand in lyophilized powder form was dissolved in a 11.2 mg/mL solution of Grb2 SH2 in water such to give a protein/ligand molar ratio of 2:1. 4 uL of this solution was mixed with 3 uL of 0.2 M ammonium acetate, 0.1 M sodium acetate, 30% v/v PEG MW4000, pH 4.6 to create the hanging drop, which yielded crystals of the protein-ligand complex in the presence of the above-mentioned solution after four weeks at room temperature., VAPOR DIFFUSION, HANGING DROP, temperature 298K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  ROTATING ANODE / Type: RIGAKU RU200 / Wavelength: 1.5418 Å ROTATING ANODE / Type: RIGAKU RU200 / Wavelength: 1.5418 Å |
| Detector | Type: RIGAKU RAXIS IV++ / Detector: IMAGE PLATE / Date: Oct 1, 2006 |
| Radiation | Monochromator: blue max-flux confocal / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.5418 Å / Relative weight: 1 |
| Reflection | Resolution: 2.02→50 Å / Num. all: 14334 / Num. obs: 13832 / % possible obs: 96.5 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 2.1 % / Rmerge(I) obs: 0.047 / Χ2: 1.606 / Net I/σ(I): 16.7 |
| Reflection shell | Resolution: 2.02→2.09 Å / Redundancy: 1.7 % / Rmerge(I) obs: 0.265 / Num. unique all: 1220 / Χ2: 1.553 / % possible all: 84.7 |
-Phasing
| Phasing | Method:  molecular replacement molecular replacement |
|---|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: pdb entry 2HUW Resolution: 2.02→50 Å / Occupancy max: 1 / Occupancy min: 0.5 / Cross valid method: THROUGHOUT / σ(F): 0 / σ(I): 0 / Stereochemistry target values: Engh & Huber
| ||||||||||||||||||||||||||||
| Solvent computation | Bsol: 31.59 Å2 | ||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 55.85 Å2 / Biso mean: 26.942 Å2 / Biso min: 13.47 Å2
| ||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.02→50 Å
| ||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||
| Xplor file |
|
 Movie
Movie Controller
Controller













 PDBj
PDBj


























