[English] 日本語
 Yorodumi
Yorodumi- PDB-4arb: Mus musculus Acetylcholinesterase in complex with (S)-C5685 at 2.... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4arb | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Mus musculus Acetylcholinesterase in complex with (S)-C5685 at 2.25 A resolution. | |||||||||
 Components Components | ACETYLCHOLINESTERASE | |||||||||
 Keywords Keywords | HYDROLASE / ENATIOMERS / INHIBITOR / CHEMICAL LEAD | |||||||||
| Function / homology |  Function and homology information Function and homology informationacetylcholine metabolic process / serine hydrolase activity / acetylcholine catabolic process / acetylcholinesterase / positive regulation of dendrite morphogenesis / cholinesterase activity / choline metabolic process / acetylcholine binding / osteoblast development / acetylcholine receptor signaling pathway ...acetylcholine metabolic process / serine hydrolase activity / acetylcholine catabolic process / acetylcholinesterase / positive regulation of dendrite morphogenesis / cholinesterase activity / choline metabolic process / acetylcholine binding / osteoblast development / acetylcholine receptor signaling pathway / acetylcholinesterase activity / positive regulation of axonogenesis / basement membrane / regulation of receptor recycling / synaptic cleft / side of membrane / collagen binding / synapse assembly / laminin binding / neuromuscular junction / receptor internalization / response to insulin / nuclear envelope / positive regulation of cold-induced thermogenesis / retina development in camera-type eye / presynaptic membrane / postsynaptic membrane / cell adhesion / membrane raft / endoplasmic reticulum lumen / hydrolase activity / axon / neuronal cell body / synapse / dendrite / perinuclear region of cytoplasm / cell surface / Golgi apparatus / protein homodimerization activity / extracellular space / extracellular region / identical protein binding / membrane / plasma membrane Similarity search - Function | |||||||||
| Biological species |  | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.25 Å MOLECULAR REPLACEMENT / Resolution: 2.25 Å | |||||||||
 Authors Authors | Berg, L. / Niemiec, M.S. / Qian, W. / Andersson, C.D. / WittungStafshede, P. / Ekstrom, F. / Linusson, A. | |||||||||
 Citation Citation |  Journal: Angew.Chem.Int.Ed.Engl. / Year: 2012 Journal: Angew.Chem.Int.Ed.Engl. / Year: 2012Title: Similar But Different: Thermodynamic and Structural Characterization of a Pair of Enantiomers Binding to Acetylcholinesterase. Authors: Berg, L. / Niemiec, M.S. / Qian, W. / Andersson, C.D. / Wittung-Stafshede, P. / Ekstrom, F. / Linusson, A. #1:  Journal: Plos One / Year: 2011 Journal: Plos One / Year: 2011Title: Targeting Acetylcholinesterase: Identification of Chemical Leads by High Throughput Screening, Structure Determination and Molecular Modeling. Authors: Berg, L. / Andersson, C.D. / Artursson, E. / Hornberg, A. / Tunemalm, A. / Linusson, A. / Ekstrom, F. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4arb.cif.gz 4arb.cif.gz | 446.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4arb.ent.gz pdb4arb.ent.gz | 367.4 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4arb.json.gz 4arb.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ar/4arb https://data.pdbj.org/pub/pdb/validation_reports/ar/4arb ftp://data.pdbj.org/pub/pdb/validation_reports/ar/4arb ftp://data.pdbj.org/pub/pdb/validation_reports/ar/4arb | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  4araC  1j06S S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein / Sugars , 2 types, 5 molecules AB

| #1: Protein | Mass: 60233.984 Da / Num. of mol.: 2 / Fragment: CATALYTIC DOMAIN, RESIDUES 32-574 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   Homo sapiens (human) / References: UniProt: P21836, acetylcholinesterase Homo sapiens (human) / References: UniProt: P21836, acetylcholinesterase#4: Sugar | |
|---|
-Non-polymers , 4 types, 998 molecules 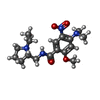






| #2: Chemical | | #3: Chemical | ChemComp-PEG / #5: Chemical | ChemComp-P6G / | #6: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|---|
| Nonpolymer details | 4-(DIMETHYLAMINO)-N-{[(2R)-1-ETHYLPYRROLIDIN- 2-YL]METHYL}-2-METHOXY-5-NITROBENZAMIDE (568): ...4-(DIMETHYLAM |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.6 Å3/Da / Density % sol: 65 % / Description: NONE |
|---|---|
| Crystal grow | pH: 7 Details: 0.1 M HEPES, 30 % (V/V) POLYETHELENEGLYCOLEMONOMETHYLETHER, pH 7 |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  MAX II MAX II  / Beamline: I911-3 / Wavelength: 1.039 / Beamline: I911-3 / Wavelength: 1.039 |
| Detector | Type: MARMOSAIC 225 mm CCD / Detector: CCD / Date: Jan 29, 2011 / Details: MIRRORS |
| Radiation | Monochromator: RH-COATED SI MIRROR, BENT FOR VERTICAL COLLIMATION Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.039 Å / Relative weight: 1 |
| Reflection | Resolution: 2.25→29.1 Å / Num. obs: 96077 / % possible obs: 99.9 % / Observed criterion σ(I): 3 / Redundancy: 5.7 % / Biso Wilson estimate: 41.84 Å2 / Rmerge(I) obs: 0.07 / Net I/σ(I): 16 |
| Reflection shell | Resolution: 2.25→2.37 Å / Redundancy: 5.7 % / Rmerge(I) obs: 0.48 / Mean I/σ(I) obs: 3.7 / % possible all: 99.9 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 1J06 Resolution: 2.25→28.806 Å / SU ML: 0.3 / σ(F): 1.33 / Phase error: 20.24 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.86 Å / VDW probe radii: 1.1 Å / Solvent model: FLAT BULK SOLVENT MODEL / Bsol: 70.323 Å2 / ksol: 0.342 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.25→28.806 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller








































































 PDBj
PDBj




