[English] 日本語
 Yorodumi
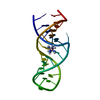
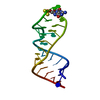
Yorodumi- PDB-483d: CRYSTAL STRUCTURE OF THE SARCIN/RICIN DOMAIN FROM E. COLI 23 S RRNA -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 483d | ||||||
|---|---|---|---|---|---|---|---|
| Title | CRYSTAL STRUCTURE OF THE SARCIN/RICIN DOMAIN FROM E. COLI 23 S RRNA | ||||||
 Components Components | SARCIN/RICIN RRNA DOMAIN | ||||||
 Keywords Keywords | RNA / SACIN/RICIN DOMAIN RNA RECOGNITION RIBOSOMES ELONGATION FACTORS CRYSTAL STRUCTURE | ||||||
| Function / homology | RNA / RNA (> 10) Function and homology information Function and homology information | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.11 Å MOLECULAR REPLACEMENT / Resolution: 1.11 Å | ||||||
 Authors Authors | Correll, C.C. / Wool, I.G. / Munishkin, A. | ||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 1999 Journal: J.Mol.Biol. / Year: 1999Title: The two faces of the Escherichia coli 23 S rRNA sarcin/ricin domain: the structure at 1.11 A resolution. Authors: Correll, C.C. / Wool, I.G. / Munishkin, A. #1:  Journal: Proc.Natl.Acad.Sci.USA / Year: 1998 Journal: Proc.Natl.Acad.Sci.USA / Year: 1998Title: Crystal Structure of the Ribosomal RNA Domain Essential for Binding Elongation Factors Authors: Correll, C.C. / Munishkin, A. / Chan, Y.-L. / Ren, Z. / Wool, I.G. #2:  Journal: Biophys.J. / Year: 1999 Journal: Biophys.J. / Year: 1999Title: Comparison of the Crystal and Solution Structures of Two RNA Oligonucleotides Authors: Rife, J.P. / Stallings, S.G. / Correll, C.C. / Dallas, A. / Steitz, T.A. / Moore, P.B. #3:  Journal: J.Mol.Biol. / Year: 1995 Journal: J.Mol.Biol. / Year: 1995Title: The Sarcin/Ricin Loop, a Modular RNA Authors: Szewczak, A.A. / Moore, P.B. #4:  Journal: Proc.Natl.Acad.Sci.USA / Year: 1993 Journal: Proc.Natl.Acad.Sci.USA / Year: 1993Title: The Conformation of the Sarcin/Ricin Loop from 28 S Ribosomal RNA Authors: Szewczak, A.A. / Moore, P.B. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  483d.cif.gz 483d.cif.gz | 50.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb483d.ent.gz pdb483d.ent.gz | 37.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  483d.json.gz 483d.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  483d_validation.pdf.gz 483d_validation.pdf.gz | 396.6 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  483d_full_validation.pdf.gz 483d_full_validation.pdf.gz | 397.4 KB | Display | |
| Data in XML |  483d_validation.xml.gz 483d_validation.xml.gz | 5.4 KB | Display | |
| Data in CIF |  483d_validation.cif.gz 483d_validation.cif.gz | 7.4 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/83/483d https://data.pdbj.org/pub/pdb/validation_reports/83/483d ftp://data.pdbj.org/pub/pdb/validation_reports/83/483d ftp://data.pdbj.org/pub/pdb/validation_reports/83/483d | HTTPS FTP |
-Related structure data
| Related structure data |  480dC  430dS S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| Unit cell |
|
- Components
Components
| #1: RNA chain | Mass: 8744.255 Da / Num. of mol.: 1 / Fragment: 27 NA LONG SARCIN/RICIN DOMAIN / Source method: obtained synthetically Details: SEQUENCE OCCURS NATURALLY IN E.COLI 28 S RIBOSOMAL RNA |
|---|---|
| #2: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.97 Å3/Da / Density % sol: 50 % | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Method: vapor diffusion, hanging drop / pH: 7 Details: 3.0 - 3.2 M (NH4)2SO4, (50 MM KMOPS, 10 MM MGCL2, 10MM MNCL2), 2.5 MG/ML RNA, pH 7.00, VAPOR DIFFUSION, HANGING DROP | ||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions |
| ||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Temperature: 19 ℃ / pH: 8 / Method: vapor diffusion | ||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 14-BM-D / Wavelength: 0.9188 / Beamline: 14-BM-D / Wavelength: 0.9188 |
| Detector | Type: ADSC / Detector: CCD / Date: May 14, 1998 / Details: BENT CYLINDRICAL SI-MIRROR |
| Radiation | Monochromator: SI(111) DOUBLE CRYSTAL / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.9188 Å / Relative weight: 1 |
| Reflection | Resolution: 1.11→20 Å / Num. obs: 25407 / % possible obs: 97.9 % / Observed criterion σ(I): -3 / Redundancy: 4.7 % / Rmerge(I) obs: 0.042 / Rsym value: 0.042 / Net I/σ(I): 35.4 |
| Reflection shell | Resolution: 1.11→1.12 Å / Redundancy: 2.2 % / Rmerge(I) obs: 0.487 / Mean I/σ(I) obs: 2.2 / Rsym value: 0.487 / % possible all: 2.3 |
| Reflection | *PLUS Highest resolution: 1.11 Å / Lowest resolution: 20 Å / Observed criterion σ(I): -3 / Redundancy: 4.7 % |
| Reflection shell | *PLUS % possible obs: 84.5 % / Redundancy: 2.2 % |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: THE RELATED RAT SARCIN/RICIN STRUCTURE (430D) Resolution: 1.11→20 Å / Num. parameters: 7554 / Num. restraintsaints: 10247 / Cross valid method: FREE R / σ(F): 0 Stereochemistry target values: G. PARKINSON, J. VOJTECHOVSKY, L. CLOWNEY, A.T. BRUNGER, H.M. BERMAN: ACTA CRYST.D, 52, 57 (1996) Details: ANISOTROPIC SCALING APPLIED BY THE METHOD OF PARKIN, MOEZZI & HOPE, J.APPL.CRYST. 28 (1995) 53-56
| |||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: MOEWS & KRETSINGER, J.MOL.BIOL. 91 (1973) 201-228 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 8 / Occupancy sum hydrogen: 0 / Occupancy sum non hydrogen: 732 | |||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.11→20 Å
| |||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||
| Software | *PLUS Name: SHELXL-97 / Classification: refinement | |||||||||||||||||||||||||||||||||
| Refinement | *PLUS % reflection Rfree: 5.3 % / Rfactor Rwork: 0.152 | |||||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS Biso mean: 13.5 Å2 | |||||||||||||||||||||||||||||||||
| Refine LS restraints | *PLUS
| |||||||||||||||||||||||||||||||||
| LS refinement shell | *PLUS Rfactor obs: 0.295 |
 Movie
Movie Controller
Controller






 PDBj
PDBj






























