+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 3uh8 | ||||||
|---|---|---|---|---|---|---|---|
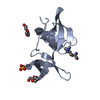
| Title | N-terminal domain of phage TP901-1 ORF48 | ||||||
 Components Components | ORF48 | ||||||
 Keywords Keywords | VIRAL PROTEIN / all beta protein / immunoglobulin fold / structural protein / TP901-1 ORF46 and ORF49 / phage baseplate | ||||||
| Function / homology |  Function and homology information Function and homology information | ||||||
| Biological species |  Lactococcus phage TP901-1 (virus) Lactococcus phage TP901-1 (virus) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  SAD / Resolution: 2.3 Å SAD / Resolution: 2.3 Å | ||||||
 Authors Authors | Veesler, D. / Spinelli, S. / Mahony, J. / Lichiere, J. / Blangy, S. / Bricogne, G. / Legrand, P. / Ortiz-Lombardia, M. / Campanacci, V.I. / van Sinderen, D. / Cambillau, C. | ||||||
 Citation Citation |  Journal: Proc.Natl.Acad.Sci.USA / Year: 2012 Journal: Proc.Natl.Acad.Sci.USA / Year: 2012Title: Structure of the phage TP901-1 1.8 MDa baseplate suggests an alternative host adhesion mechanism. Authors: Veesler, D. / Spinelli, S. / Mahony, J. / Lichiere, J. / Blangy, S. / Bricogne, G. / Legrand, P. / Ortiz-Lombardia, M. / Campanacci, V. / van Sinderen, D. / Cambillau, C. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  3uh8.cif.gz 3uh8.cif.gz | 57.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb3uh8.ent.gz pdb3uh8.ent.gz | 42 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  3uh8.json.gz 3uh8.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Summary document |  3uh8_validation.pdf.gz 3uh8_validation.pdf.gz | 423 KB | Display |  wwPDB validaton report wwPDB validaton report |
|---|---|---|---|---|
| Full document |  3uh8_full_validation.pdf.gz 3uh8_full_validation.pdf.gz | 422.9 KB | Display | |
| Data in XML |  3uh8_validation.xml.gz 3uh8_validation.xml.gz | 7.4 KB | Display | |
| Data in CIF |  3uh8_validation.cif.gz 3uh8_validation.cif.gz | 9.4 KB | Display | |
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/uh/3uh8 https://data.pdbj.org/pub/pdb/validation_reports/uh/3uh8 ftp://data.pdbj.org/pub/pdb/validation_reports/uh/3uh8 ftp://data.pdbj.org/pub/pdb/validation_reports/uh/3uh8 | HTTPS FTP |
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: Protein | Mass: 14443.050 Da / Num. of mol.: 1 / Fragment: ORF48 N-terminus, UNP residues 1-128 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Lactococcus phage TP901-1 (virus) / Plasmid: pETG20A / Production host: Lactococcus phage TP901-1 (virus) / Plasmid: pETG20A / Production host:  |
|---|---|
| #2: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 1.85 Å3/Da / Density % sol: 33.37 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 8 Details: Protein 33 mg/ml, 1.5 M K2HPO4/0.4 M NaH2PO4, 0.2M NaCl, 0.1 M imidazole, pH 8.0 , VAPOR DIFFUSION, SITTING DROP, temperature 298K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SOLEIL SOLEIL  / Beamline: PROXIMA 1 / Wavelength: 0.98011 Å / Beamline: PROXIMA 1 / Wavelength: 0.98011 Å |
| Detector | Type: ADSC QUANTUM 315r / Detector: CCD / Date: May 9, 2011 |
| Radiation | Monochromator: mirrors / Protocol: SAD / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.98011 Å / Relative weight: 1 |
| Reflection | Resolution: 2.3→45 Å / Num. all: 4537 / Num. obs: 4537 / % possible obs: 99.1 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 6.3 % / Biso Wilson estimate: 40.2 Å2 / Rmerge(I) obs: 0.068 / Net I/σ(I): 33 |
| Reflection shell | Resolution: 2.3→2.5 Å / Rmerge(I) obs: 0.142 / Mean I/σ(I) obs: 27 / % possible all: 99.5 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  SAD / Resolution: 2.3→35.56 Å / Cor.coef. Fo:Fc: 0.8972 / Cor.coef. Fo:Fc free: 0.8536 / Cross valid method: THROUGHOUT / σ(F): 0 / σ(I): 0 / Stereochemistry target values: Engh & Huber SAD / Resolution: 2.3→35.56 Å / Cor.coef. Fo:Fc: 0.8972 / Cor.coef. Fo:Fc free: 0.8536 / Cross valid method: THROUGHOUT / σ(F): 0 / σ(I): 0 / Stereochemistry target values: Engh & Huber
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 38.16 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.38 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.3→35.56 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.3→2.57 Å / Total num. of bins used: 5
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller
















 PDBj
PDBj
