[English] 日本語
 Yorodumi
Yorodumi- PDB-3p4p: Crystal structure of Menaquinol:fumarate oxidoreductase in comple... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 3p4p | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of Menaquinol:fumarate oxidoreductase in complex with fumarate | |||||||||
 Components Components | (Fumarate reductase ...) x 4 | |||||||||
 Keywords Keywords | OXIDOREDUCTASE | |||||||||
| Function / homology |  Function and homology information Function and homology informationfumarate reductase complex / fumarate reductase (quinol) / : / fermentation / succinate dehydrogenase activity / fumarate metabolic process / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / anaerobic electron transport chain / anaerobic respiration ...fumarate reductase complex / fumarate reductase (quinol) / : / fermentation / succinate dehydrogenase activity / fumarate metabolic process / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / anaerobic electron transport chain / anaerobic respiration / 3 iron, 4 sulfur cluster binding / bacterial-type flagellum assembly / iron-sulfur cluster binding / tricarboxylic acid cycle / FAD binding / electron transport chain / 2 iron, 2 sulfur cluster binding / flavin adenine dinucleotide binding / 4 iron, 4 sulfur cluster binding / electron transfer activity / DNA damage response / metal ion binding / membrane / plasma membrane / cytosol Similarity search - Function | |||||||||
| Biological species |    | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / Resolution: 2.8 Å SYNCHROTRON / Resolution: 2.8 Å | |||||||||
 Authors Authors | Tomasiak, T.M. / Archuleta, T.L. / Andr ll, J. / Luna-Ch vez, C. / Davis, T.A. / Sarwar, M. / Ham, A.J. / McDonald, W.H. / Yankowskaya, V. / Stern, H.A. ...Tomasiak, T.M. / Archuleta, T.L. / Andr ll, J. / Luna-Ch vez, C. / Davis, T.A. / Sarwar, M. / Ham, A.J. / McDonald, W.H. / Yankowskaya, V. / Stern, H.A. / Johnston, J.N. / Maklashina, E. / Cecchini, G. / Iverson, T.M. | |||||||||
 Citation Citation |  Journal: J.Biol.Chem. / Year: 2011 Journal: J.Biol.Chem. / Year: 2011Title: Geometric restraint drives on- and off-pathway catalysis by the Escherichia coli menaquinol:fumarate reductase. Authors: Tomasiak, T.M. / Archuleta, T.L. / Andrell, J. / Luna-Chavez, C. / Davis, T.A. / Sarwar, M. / Ham, A.J. / McDonald, W.H. / Yankovskaya, V. / Stern, H.A. / Johnston, J.N. / Maklashina, E. / ...Authors: Tomasiak, T.M. / Archuleta, T.L. / Andrell, J. / Luna-Chavez, C. / Davis, T.A. / Sarwar, M. / Ham, A.J. / McDonald, W.H. / Yankovskaya, V. / Stern, H.A. / Johnston, J.N. / Maklashina, E. / Cecchini, G. / Iverson, T.M. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  3p4p.cif.gz 3p4p.cif.gz | 847.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb3p4p.ent.gz pdb3p4p.ent.gz | 705 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  3p4p.json.gz 3p4p.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/p4/3p4p https://data.pdbj.org/pub/pdb/validation_reports/p4/3p4p ftp://data.pdbj.org/pub/pdb/validation_reports/p4/3p4p ftp://data.pdbj.org/pub/pdb/validation_reports/p4/3p4p | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
|
- Components
Components
-Fumarate reductase ... , 4 types, 8 molecules AMBNCODP
| #1: Protein | Mass: 63477.707 Da / Num. of mol.: 2 / Fragment: UNP residues 1-577 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   References: UniProt: D3GV56, UniProt: P00363*PLUS, EC: 1.3.99.1 #2: Protein | Mass: 27021.885 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   References: UniProt: Q0T9N6, UniProt: P0AC47*PLUS, EC: 1.3.99.1 #3: Protein | Mass: 14898.773 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   #4: Protein | Mass: 13118.870 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   |
|---|
-Non-polymers , 6 types, 88 molecules 
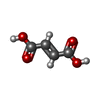









| #5: Chemical | | #6: Chemical | ChemComp-FUM / | #7: Chemical | #8: Chemical | #9: Chemical | #10: Water | ChemComp-HOH / | |
|---|
-Details
| Has protein modification | Y |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.78 Å3/Da / Density % sol: 67.5 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion / pH: 5.8 Details: 12.5% PEG 5000 mme, 85mM MgAc, 100mM Citrate pH 5.8, 0.1% w/v DTT, 0.1mM EDTA, 5mM fumarate, vapor diffusion, temperature 298K, VAPOR DIFFUSION |
-Data collection
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SSRL SSRL  / Beamline: BL9-2 / Wavelength: 1 Å / Beamline: BL9-2 / Wavelength: 1 Å |
|---|---|
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 2.8→90.76 Å / Num. obs: 84789 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Resolution: 2.8→38.92 Å / Cor.coef. Fo:Fc: 0.904 / Cor.coef. Fo:Fc free: 0.896 / SU B: 31.801 / SU ML: 0.298
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: BABINET MODEL WITH MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 104.52 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.8→38.92 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.8→2.87 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller





















 PDBj
PDBj

















