| 登録情報 | データベース: PDB / ID: 3f57
|
|---|
| タイトル | Crystal structure of human erythroid beta spectrin repeats 14 and 15 (ankyrin binding domain) |
|---|
 要素 要素 | Spectrin beta chain, erythrocyte |
|---|
 キーワード キーワード | STRUCTURAL PROTEIN / spectrin / spectrin repeat / three-helix-bundle / ankyrin binding / Actin capping / Actin-binding / Cytoskeleton / Disease mutation / Elliptocytosis / Hereditary hemolytic anemia / Phosphoprotein |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
spectrin / spectrin-associated cytoskeleton / modification of postsynaptic actin cytoskeleton / actin filament capping / Interaction between L1 and Ankyrins / ankyrin binding / cortical actin cytoskeleton / COPI-mediated anterograde transport / NCAM signaling for neurite out-growth / cell projection ...spectrin / spectrin-associated cytoskeleton / modification of postsynaptic actin cytoskeleton / actin filament capping / Interaction between L1 and Ankyrins / ankyrin binding / cortical actin cytoskeleton / COPI-mediated anterograde transport / NCAM signaling for neurite out-growth / cell projection / structural constituent of cytoskeleton / cytoplasmic side of plasma membrane / actin filament binding / cell junction / actin cytoskeleton / actin binding / RAF/MAP kinase cascade / actin cytoskeleton organization / postsynapse / glutamatergic synapse / cell surface / protein-containing complex / plasma membrane / cytosol類似検索 - 分子機能 Spectrin, beta subunit / Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #60 / Spectrin repeat / Spectrin repeat / Actinin-type actin-binding domain signature 1. / Actinin-type actin-binding domain signature 2. / Actinin-type actin-binding domain, conserved site / Spectrin/alpha-actinin / Spectrin repeats / Calponin homology domain ...Spectrin, beta subunit / Methane Monooxygenase Hydroxylase; Chain G, domain 1 - #60 / Spectrin repeat / Spectrin repeat / Actinin-type actin-binding domain signature 1. / Actinin-type actin-binding domain signature 2. / Actinin-type actin-binding domain, conserved site / Spectrin/alpha-actinin / Spectrin repeats / Calponin homology domain / Calponin homology (CH) domain / Calponin homology domain / CH domain superfamily / Calponin homology (CH) domain profile. / Methane Monooxygenase Hydroxylase; Chain G, domain 1 / Up-down Bundle / Mainly Alpha類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2.9 Å 単波長異常分散 / 解像度: 2.9 Å |
|---|
 データ登録者 データ登録者 | Ipsaro, J.J. / Mondragon, A. |
|---|
 引用 引用 |  ジャーナル: Blood / 年: 2009 ジャーナル: Blood / 年: 2009
タイトル: Structures of the spectrin-ankyrin interaction binding domains.
著者: Ipsaro, J.J. / Huang, L. / Mondragon, A. |
|---|
| 履歴 | | 登録 | 2008年11月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年2月24日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Advisory / Version format compliance |
|---|
| 改定 1.2 | 2017年10月25日 | Group: Refinement description / カテゴリ: software / Item: _software.name |
|---|
| 改定 1.3 | 2023年12月27日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 2.9 Å
単波長異常分散 / 解像度: 2.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Blood / 年: 2009
ジャーナル: Blood / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3f57.cif.gz
3f57.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3f57.ent.gz
pdb3f57.ent.gz PDB形式
PDB形式 3f57.json.gz
3f57.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/f5/3f57
https://data.pdbj.org/pub/pdb/validation_reports/f5/3f57 ftp://data.pdbj.org/pub/pdb/validation_reports/f5/3f57
ftp://data.pdbj.org/pub/pdb/validation_reports/f5/3f57 リンク
リンク 集合体
集合体
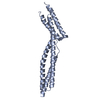

 要素
要素 Homo sapiens (ヒト) / 遺伝子: SPTB, SPTB1 / プラスミド: pMCSG7 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: SPTB, SPTB1 / プラスミド: pMCSG7 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 21-ID-G / 波長: 0.97857 Å
/ ビームライン: 21-ID-G / 波長: 0.97857 Å 解析
解析 単波長異常分散 / 解像度: 2.9→19.84 Å / Cor.coef. Fo:Fc: 0.914 / Cor.coef. Fo:Fc free: 0.874 / SU B: 43.539 / SU ML: 0.359 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 1 / ESU R Free: 0.443 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
単波長異常分散 / 解像度: 2.9→19.84 Å / Cor.coef. Fo:Fc: 0.914 / Cor.coef. Fo:Fc free: 0.874 / SU B: 43.539 / SU ML: 0.359 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 1 / ESU R Free: 0.443 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー









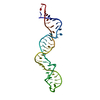



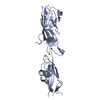






 PDBj
PDBj







