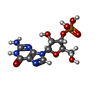
| ソフトウェア | | 名称 | バージョン | 分類 |
|---|
| REFMAC | 5.2.0019| 精密化 | | DENZO | | データ削減 | | SCALEPACK | | データスケーリング | | MOLREP | | 位相決定 | |
|
|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換
開始モデル: PDB entry 1py3
解像度: 2.2→19.88 Å / Cor.coef. Fo:Fc: 0.933 / Cor.coef. Fo:Fc free: 0.912 / SU B: 12.601 / SU ML: 0.178 / TLS residual ADP flag: LIKELY RESIDUAL / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 / ESU R: 0.322 / ESU R Free: 0.241 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| Rfactor | 反射数 | %反射 | Selection details |
|---|
| Rfree | 0.2646 | 869 | 5.2 % | RANDOM |
|---|
| Rwork | 0.22047 | - | - | - |
|---|
| all | 0.2228 | 16671 | - | - |
|---|
| obs | 0.2228 | 15801 | 88.31 % | - |
|---|
|
|---|
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK |
|---|
| 原子変位パラメータ | Biso mean: 31.703 Å2
| Baniso -1 | Baniso -2 | Baniso -3 |
|---|
| 1- | -1.46 Å2 | 0 Å2 | -0.54 Å2 |
|---|
| 2- | - | 3.82 Å2 | 0 Å2 |
|---|
| 3- | - | - | -2.57 Å2 |
|---|
|
|---|
| Refine analyze | | Free | Obs |
|---|
| Luzzati coordinate error | 0.235 Å | 0.014 Å |
|---|
| Luzzati d res low | - | 0 Å |
|---|
| Luzzati sigma a | 0.332 Å | - |
|---|
|
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 2.2→19.88 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 2232 | 0 | 34 | 133 | 2399 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | Dev ideal target | 数 |
|---|
| X-RAY DIFFRACTION | r_bond_refined_d| 0.014 | 0.022 | 2340 | | X-RAY DIFFRACTION | r_angle_refined_deg| 1.52 | 1.969 | 3186 | | X-RAY DIFFRACTION | r_dihedral_angle_1_deg| 6.926 | 5 | 274 | | X-RAY DIFFRACTION | r_dihedral_angle_2_deg| 30.583 | 22.602 | 123 | | X-RAY DIFFRACTION | r_dihedral_angle_3_deg| 19.548 | 15 | 349 | | X-RAY DIFFRACTION | r_dihedral_angle_4_deg| 21.128 | 15 | 26 | | X-RAY DIFFRACTION | r_chiral_restr| 0.108 | 0.2 | 320 | | X-RAY DIFFRACTION | r_gen_planes_refined| 0.005 | 0.02 | 1867 | | X-RAY DIFFRACTION | r_nbd_refined| 0.208 | 0.2 | 1049 | | X-RAY DIFFRACTION | r_nbtor_refined| 0.302 | 0.2 | 1557 | | X-RAY DIFFRACTION | r_xyhbond_nbd_refined| 0.186 | 0.2 | 146 | | X-RAY DIFFRACTION | r_symmetry_vdw_refined| 0.304 | 0.2 | 52 | | X-RAY DIFFRACTION | r_symmetry_hbond_refined| 0.28 | 0.2 | 10 | | X-RAY DIFFRACTION | r_mcbond_it| 0.618 | 1.5 | 1407 | | X-RAY DIFFRACTION | r_mcangle_it| 1.041 | 2 | 2245 | | X-RAY DIFFRACTION | r_scbond_it| 1.538 | 3 | 1061 | | X-RAY DIFFRACTION | r_scangle_it| 2.327 | 4.5 | 941 | | | | | | | | | | | | | | | | | |
|
|---|
| LS精密化 シェル | 解像度: 2.2→2.257 Å / Total num. of bins used: 20
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.332 | 49 | - |
|---|
| Rwork | 0.235 | 790 | - |
|---|
| obs | - | 456 | 61.15 % |
|---|
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 3.4822 | -1.6176 | -0.0193 | 8.7593 | 1.0642 | 4.6643 | 0.0687 | 0.0011 | 0.285 | -0.0062 | -0.1204 | 0.1755 | -0.6898 | 0.4855 | 0.0517 | 0.0642 | -0.1353 | 0.0237 | 0.0571 | -0.0055 | -0.0381 | 68.0254 | 5.884 | -27.7112 | | 2 | 2.6412 | -0.0023 | -0.3765 | 1.8382 | 0.3677 | 2.3333 | 0.0859 | -0.2551 | -0.0319 | 0.1799 | -0.1007 | -0.0197 | 0.0131 | 0.2324 | 0.0147 | 0.0231 | -0.0193 | 0.0031 | 0.0672 | 0.0038 | -0.004 | 61.7789 | -4.6101 | -18.2193 | | 3 | 1.1078 | 4.8496 | 0.7424 | 21.6434 | 7.3712 | 41.5159 | 2.4659 | 1.404 | 1.9866 | 0.7683 | -0.6541 | -3.8134 | 0.5927 | 4.8435 | -1.8117 | 0.299 | 0.0284 | -0.1822 | 0.364 | 0.2223 | 0.794 | 79.1009 | -7.372 | -19.0699 | | 4 | 2.4955 | 1.0691 | -0.2913 | 2.075 | 0.0671 | 2.189 | 0.1457 | -0.0698 | 0.2486 | -0.0215 | -0.1564 | -0.0566 | -0.3983 | 0.2444 | 0.0107 | 0.0418 | -0.0657 | 0.0105 | 0.1294 | -0.0008 | -0.0108 | 63.7492 | 3.3203 | -20.5736 | | 5 | 5.055 | -1.5501 | 1.8775 | 0.6239 | -1.4635 | 6.002 | -0.0159 | 0.6878 | -0.2423 | -0.0689 | 0.0456 | 0.0685 | 0.6554 | 0.0153 | -0.0297 | -0.0044 | 0.1102 | 0.0079 | -0.0401 | -0.0401 | 0.0362 | 65.8225 | -25.5388 | 12.965 | | 6 | 7.8747 | -0.9993 | 2.3132 | 5.1486 | -1.1446 | 5.7262 | -0.0107 | -0.2315 | -0.4829 | 0.3513 | 0.2355 | 0.5522 | 0.145 | -0.3887 | -0.2248 | -0.0656 | 0.0678 | 0.0339 | 0.0185 | 0.0561 | 0.0268 | 61.3631 | -20.8848 | 25.0703 | | 7 | 2.7917 | -1.8163 | 0.357 | 3.7553 | 0.0819 | 5.3265 | 0.0373 | 0.2924 | -0.5184 | 0.2007 | -0.147 | 0.509 | 0.7455 | -0.0421 | 0.1097 | -0.0138 | 0.0666 | 0.0104 | 0.1064 | 0.009 | 0.0618 | 62.7189 | -22.807 | 20.9308 | | 8 | 6.7565 | 1.7472 | -1.8533 | 2.0343 | -5.1207 | 21.6018 | 0.4779 | 0.1795 | -0.4197 | -0.1564 | -0.232 | 0.2828 | 0.3323 | 0.5868 | -0.2459 | -0.0628 | 0.1828 | 0.0173 | -0.0483 | -0.021 | 0.1525 | 69.0251 | -26.432 | 17.561 | | 9 | 7.2661 | -0.6202 | 0.0395 | 1.761 | 0.3693 | 2.6377 | 0.0915 | 0.402 | 0.047 | 0.0333 | -0.1866 | -0.1414 | -0.3675 | 0.1774 | 0.0951 | -0.0159 | 0.0321 | 0.0014 | 0.1445 | 0.0655 | -0.06 | 64.3181 | -5.1274 | 11.4941 | | 10 | 3.0398 | -1.3813 | -1.691 | 2.4161 | 0.0709 | 4.5533 | 0.1872 | 0.6865 | -0.1218 | -0.1608 | -0.2661 | 0.0552 | -0.3115 | -0.5653 | 0.0789 | -0.0145 | 0.1142 | -0.004 | 0.3005 | 0.0123 | -0.0539 | 59.9154 | -9.2867 | -0.1144 | | 11 | 6.033 | 2.1122 | -2.9938 | 9.1277 | -4.3852 | 6.4182 | 0.6438 | 0.5704 | 0.3194 | 0.2934 | -0.4852 | 0.2111 | -0.6852 | 0.1712 | -0.1586 | 0.0123 | 0.0446 | 0.0117 | 0.2765 | 0.0576 | -0.0563 | 64.211 | -6.8802 | 5.0201 | | 12 | 16.0837 | -6.6068 | 13.0876 | 3.4086 | -2.074 | 26.3462 | 0.7331 | 1.1679 | -0.3097 | -0.1247 | -0.8617 | -0.0044 | -0.3101 | 0.768 | 0.1286 | -0.1693 | 0.0531 | -0.0098 | 0.214 | 0.1161 | -0.0256 | 70.0593 | -8.7779 | 3.4965 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Auth asym-ID | Label asym-ID | Auth seq-ID | Label seq-ID |
|---|
| 1 | X-RAY DIFFRACTION | 1 | AA| 5 - 19 | 5 - 19 | | 2 | X-RAY DIFFRACTION | 2 | AA| 20 - 61 | 20 - 61 | | 3 | X-RAY DIFFRACTION | 3 | AA| 62 - 67 | 62 - 67 | | 4 | X-RAY DIFFRACTION | 4 | AA| 68 - 97 | 68 - 97 | | 5 | X-RAY DIFFRACTION | 5 | BB| 1 - 25 | 1 - 25 | | 6 | X-RAY DIFFRACTION | 6 | BB| 26 - 49 | 26 - 49 | | 7 | X-RAY DIFFRACTION | 7 | BB| 50 - 82 | 50 - 82 | | 8 | X-RAY DIFFRACTION | 8 | BB| 83 - 97 | 83 - 97 | | 9 | X-RAY DIFFRACTION | 9 | CC| 5 - 37 | 5 - 37 | | 10 | X-RAY DIFFRACTION | 10 | CC| 38 - 62 | 38 - 62 | | 11 | X-RAY DIFFRACTION | 11 | CC| 67 - 84 | 67 - 84 | | 12 | X-RAY DIFFRACTION | 12 | CC| 85 - 97 | 85 - 97 | | | | | | | | | | | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Streptomyces aureofaciens (バクテリア)
Streptomyces aureofaciens (バクテリア) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Febs J. / 年: 2009
ジャーナル: Febs J. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3d4a.cif.gz
3d4a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3d4a.ent.gz
pdb3d4a.ent.gz PDB形式
PDB形式 3d4a.json.gz
3d4a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3d4a_validation.pdf.gz
3d4a_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3d4a_full_validation.pdf.gz
3d4a_full_validation.pdf.gz 3d4a_validation.xml.gz
3d4a_validation.xml.gz 3d4a_validation.cif.gz
3d4a_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/d4/3d4a
https://data.pdbj.org/pub/pdb/validation_reports/d4/3d4a ftp://data.pdbj.org/pub/pdb/validation_reports/d4/3d4a
ftp://data.pdbj.org/pub/pdb/validation_reports/d4/3d4a リンク
リンク 集合体
集合体



 要素
要素 Streptomyces aureofaciens (バクテリア)
Streptomyces aureofaciens (バクテリア)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  EMBL/DESY, HAMBURG
EMBL/DESY, HAMBURG  / ビームライン: X31 / 波長: 1.1 Å
/ ビームライン: X31 / 波長: 1.1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj