Entry Database : PDB / ID : 3bxkTitle Crystal structure of the P/Q-type calcium channel (CaV2.1) IQ domain and CA2+calmodulin complex Calmodulin Voltage-dependent P/Q-type calcium channel subunit alpha-1A peptide Keywords / / / / / / / / / / / / / / / / / / / / / / Function / homology Function Domain/homology Component
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / Biological species Rattus norvegicus (Norway rat)Method / / Resolution : 2.55 Å Authors Mori, M.X. / Vander Kooi, C.W. / Leahy, D.J. / Yue, D.T. Journal : To be Published Title : Crystal structure of the P/Q-type calcium channel (CaV2.1) IQ domain and CA2+calmodulin complexAuthors : Mori, M.X. / Vander Kooi, C.W. / Leahy, D.J. / Yue, D.T. History Deposition Jan 14, 2008 Deposition site / Processing site Revision 1.0 Mar 25, 2008 Provider / Type Revision 1.1 Jul 13, 2011 Group / Version format complianceRevision 1.2 Feb 21, 2024 Group / Database references / Derived calculationsCategory chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
Show all Show less
 Yorodumi
Yorodumi Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
Function and homology information
 X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / Resolution: 2.55 Å
MOLECULAR REPLACEMENT / Resolution: 2.55 Å  Authors
Authors Citation
Citation Journal: To be Published
Journal: To be Published Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 3bxk.cif.gz
3bxk.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb3bxk.ent.gz
pdb3bxk.ent.gz PDB format
PDB format 3bxk.json.gz
3bxk.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/bx/3bxk
https://data.pdbj.org/pub/pdb/validation_reports/bx/3bxk ftp://data.pdbj.org/pub/pdb/validation_reports/bx/3bxk
ftp://data.pdbj.org/pub/pdb/validation_reports/bx/3bxk Links
Links Assembly
Assembly
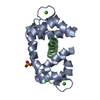

 Components
Components

 X-RAY DIFFRACTION / Number of used crystals: 2
X-RAY DIFFRACTION / Number of used crystals: 2  Sample preparation
Sample preparation ROTATING ANODE / Type: RIGAKU RU200 / Wavelength: 1.5418 Å
ROTATING ANODE / Type: RIGAKU RU200 / Wavelength: 1.5418 Å Processing
Processing MOLECULAR REPLACEMENT / Resolution: 2.55→24.87 Å / Cor.coef. Fo:Fc: 0.946 / Cor.coef. Fo:Fc free: 0.905 / SU B: 11.546 / SU ML: 0.251 / TLS residual ADP flag: LIKELY RESIDUAL / Cross valid method: THROUGHOUT / ESU R: 0.655 / ESU R Free: 0.321 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
MOLECULAR REPLACEMENT / Resolution: 2.55→24.87 Å / Cor.coef. Fo:Fc: 0.946 / Cor.coef. Fo:Fc free: 0.905 / SU B: 11.546 / SU ML: 0.251 / TLS residual ADP flag: LIKELY RESIDUAL / Cross valid method: THROUGHOUT / ESU R: 0.655 / ESU R Free: 0.321 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS Movie
Movie Controller
Controller







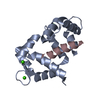





 PDBj
PDBj













