+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2vj5 | ||||||
|---|---|---|---|---|---|---|---|
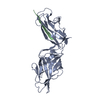
| Title | Shigella flexneri MxiC | ||||||
 Components Components | PROTEIN MXIC | ||||||
 Keywords Keywords | TRANSPORT PROTEIN / SECRETION REGULATION / T3SS / VIRULENCE / TRANSPORT / TYPE THREE SECRETION SYSTEM | ||||||
| Function / homology |  Function and homology information Function and homology informationprotein secretion by the type III secretion system / outer membrane / negative regulation of protein secretion / host cell / cell surface / extracellular region Similarity search - Function | ||||||
| Biological species |  SHIGELLA FLEXNERI (bacteria) SHIGELLA FLEXNERI (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 3 Å MOLECULAR REPLACEMENT / Resolution: 3 Å | ||||||
 Authors Authors | Deane, J.E. / Roversi, P. / King, C. / Johnson, S. / Lea, S.M. | ||||||
 Citation Citation |  Journal: J.Mol.Biol. / Year: 2008 Journal: J.Mol.Biol. / Year: 2008Title: Structures of the Shigella Flexneri Type 3 Secretion System Protein Mxic Reveal Conformational Variability Amongst Homologues. Authors: Deane, J.E. / Roversi, P. / King, C. / Johnson, S. / Lea, S.M. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2vj5.cif.gz 2vj5.cif.gz | 122.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2vj5.ent.gz pdb2vj5.ent.gz | 95.8 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2vj5.json.gz 2vj5.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/vj/2vj5 https://data.pdbj.org/pub/pdb/validation_reports/vj/2vj5 ftp://data.pdbj.org/pub/pdb/validation_reports/vj/2vj5 ftp://data.pdbj.org/pub/pdb/validation_reports/vj/2vj5 | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  2vixSC  2vj4C S: Starting model for refinement C: citing same article ( |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
| ||||||||
| Noncrystallographic symmetry (NCS) | NCS oper: (Code: given Matrix: (0.818, 0.009, 0.576), Vector: |
- Components
Components
| #1: Protein | Mass: 33643.301 Da / Num. of mol.: 2 / Fragment: RESIDUES 74-355 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  SHIGELLA FLEXNERI (bacteria) / Strain: PWR100 / Production host: SHIGELLA FLEXNERI (bacteria) / Strain: PWR100 / Production host:  #2: Water | ChemComp-HOH / | Sequence details | THE SEQUENCE HSSGLVPRGSH COMES FROM THE EXPRESSION VECTOR. THE NTERMINUS IS ABSENT FROM THE ...THE SEQUENCE HSSGLVPRGS | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.58 Å3/Da / Density % sol: 65.6 % Description: DATA INDEXED WITH LABELIT AND INTEGRATED IN XDS USING THE SUITE OF PROGRAMS XIA2 |
|---|---|
| Crystal grow | pH: 6.5 Details: 0.2 M NA2SO4, 0.1 M BISTRISPROPANE PH 6.5, 20% PEG3350 |
-Data collection
| Diffraction | Mean temperature: 173 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ESRF ESRF  / Beamline: ID29 / Wavelength: 0.976 / Beamline: ID29 / Wavelength: 0.976 |
| Detector | Type: ADSC CCD / Detector: CCD / Date: Jun 22, 2007 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.976 Å / Relative weight: 1 |
| Reflection | Resolution: 3→38.6 Å / Num. obs: 18676 / % possible obs: 98.5 % / Observed criterion σ(I): 0 / Redundancy: 6.9 % / Biso Wilson estimate: 171 Å2 / Rmerge(I) obs: 0.07 / Net I/σ(I): 14 |
| Reflection shell | Resolution: 3→3.16 Å / Redundancy: 7.1 % / Rmerge(I) obs: 0.43 / Mean I/σ(I) obs: 4.5 / % possible all: 98.4 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 2VIX Resolution: 3→38.6 Å / Isotropic thermal model: TNT BCORREL / Cross valid method: THROUGHOUT / σ(F): 0 / Stereochemistry target values: TNT PROTGEO Details: REFINED IN BUSTER-TNT 2.1.1 WITH RESTRAINTS TO A STRUCTURE REFINED IN REFMAC5
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Solvent model: BABINET SCALING / Bsol: 255 Å2 / ksol: 0.38 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 3→38.6 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj
