+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2oed | ||||||
|---|---|---|---|---|---|---|---|
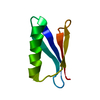
| タイトル | GB3 solution structure obtained by refinement of X-ray structure with dipolar couplings | ||||||
 要素 要素 | Immunoglobulin G-binding protein G | ||||||
 キーワード キーワード | IMMUNE SYSTEM / residual dipolar couplings | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Streptococcus sp. 'group G' (バクテリア) Streptococcus sp. 'group G' (バクテリア) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Ulmer, T.S. / Ramirez, B.E. / Delaglio, F. / Bax, A. / Grishaev, A. | ||||||
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2003 ジャーナル: J.Am.Chem.Soc. / 年: 2003タイトル: Evaluation of backbone proton positions and dynamics in a small protein by liquid crystal NMR spectroscopy 著者: Ulmer, T.S. / Ramirez, B.E. / Delaglio, F. / Bax, A. #1:  ジャーナル: J.Mol.Biol. / 年: 1994 ジャーナル: J.Mol.Biol. / 年: 1994タイトル: The third IGG-binding domain from Streptococcal protein G. An analysis by X-ray crystallography of the structure alone and in a complex with FAB 著者: Derrick, J.P. / Wiggley, D.B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2oed.cif.gz 2oed.cif.gz | 27 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2oed.ent.gz pdb2oed.ent.gz | 17.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2oed.json.gz 2oed.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2oed_validation.pdf.gz 2oed_validation.pdf.gz | 240.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2oed_full_validation.pdf.gz 2oed_full_validation.pdf.gz | 240.6 KB | 表示 | |
| XML形式データ |  2oed_validation.xml.gz 2oed_validation.xml.gz | 2.2 KB | 表示 | |
| CIF形式データ |  2oed_validation.cif.gz 2oed_validation.cif.gz | 2.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/oe/2oed https://data.pdbj.org/pub/pdb/validation_reports/oe/2oed ftp://data.pdbj.org/pub/pdb/validation_reports/oe/2oed ftp://data.pdbj.org/pub/pdb/validation_reports/oe/2oed | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: 抗体 | 分子量: 6214.848 Da / 分子数: 1 / 断片: THIRD IGG-BINDING DOMAIN / 由来タイプ: 組換発現 由来: (組換発現)  Streptococcus sp. 'group G' (バクテリア) Streptococcus sp. 'group G' (バクテリア)遺伝子: spg / プラスミド: PET-11A / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 | 内容: 25 MM NAH2PO4/NA2HPO4, 0.2 MG/ML NAN3, 1.5 MM GB3 PROTEIN AND ALIGNING MEDIA, 90% H2O, 10% D2O 溶媒系: 90% H2O/10% D2O |
|---|---|
| 試料状態 | イオン強度: 0.05 / pH: 6.5 / 圧: ambient / 温度: 300 K |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射波長 | 相対比: 1 | |||||||||||||||
| NMRスペクトロメーター |
|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: GB3 solution structure is obtained by refinement of the x-ray structure of gb3 (1igd) with c(alpha)-c', c'-n, c(alpha)-h(alpha) and N-H dipolar couplings measured in five aligning media ...詳細: GB3 solution structure is obtained by refinement of the x-ray structure of gb3 (1igd) with c(alpha)-c', c'-n, c(alpha)-h(alpha) and N-H dipolar couplings measured in five aligning media (bicelle, peg, pf1 phage, negatively and positively charged polyacrylamide gels). Dipolar couplings originating on residues 10-11, 24-26, 39-41 and the n- and c-terminal residues were excluded, resulting in near-identity with the x-ray structure for these residues. This deposition corresponds to the refined-II structure of the primary citation that was refined against all 4 types of dipolar couplings in 5 media. The related depositions 1P7E and 1P7F correspond to the refined-III and refined-IV structures, respectively. The deposited structure was calculated with DYNAMO 2.1 following a short low-temperature molecular dynamics simulated annealing run starting from the regularized coordinates of the X-ray structure 1IGD using atomic coordinate restraints to the overlapping 3-residue segments of 1IGD. The force constants for the HN-N-C-Ca impropers were softened 10-fold relative to their standard settings. See the primary citation and the corresponding supporting information file for a detailed description of the refinement procedure. The deposited restraints file includes, in addition to the dipolar couplings and backbone H-bond distance restraints (XPLOR/CNS format), an Xplor-NIH script designed to mimic the deposited structure, and the force field parameter files. A set of overlapping 3-residue NCS restraint terms is used in this script in place of the Dynamo atomic coordinate restraints. | ||||||||||||
| 代表構造 | 選択基準: best agreement with dipolar restraints | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: all calculated structures submitted 計算したコンフォーマーの数: 1 / 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

 HSQC
HSQC