+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2kg4 | ||||||
|---|---|---|---|---|---|---|---|
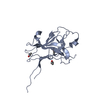
| タイトル | Three-dimensional structure of human Gadd45alpha in solution by NMR | ||||||
 要素 要素 | Growth arrest and DNA-damage-inducible protein GADD45 alpha | ||||||
 キーワード キーワード | CELL CYCLE / gadd45 / growth arrest / DNA damage / flexible regions / monomer | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of peptidyl-serine phosphorylation of STAT protein / negative regulation of protein serine/threonine kinase activity / FOXO-mediated transcription of cell cycle genes / regulation of cyclin-dependent protein serine/threonine kinase activity / positive regulation of p38MAPK cascade / centrosome cycle / negative regulation of blood vessel endothelial cell migration / signal transduction in response to DNA damage / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / negative regulation of angiogenesis ...negative regulation of peptidyl-serine phosphorylation of STAT protein / negative regulation of protein serine/threonine kinase activity / FOXO-mediated transcription of cell cycle genes / regulation of cyclin-dependent protein serine/threonine kinase activity / positive regulation of p38MAPK cascade / centrosome cycle / negative regulation of blood vessel endothelial cell migration / signal transduction in response to DNA damage / TP53 Regulates Transcription of Genes Involved in G2 Cell Cycle Arrest / negative regulation of angiogenesis / cellular response to ionizing radiation / positive regulation of JNK cascade / promoter-specific chromatin binding / cellular response to mechanical stimulus / kinase binding / positive regulation of reactive oxygen species metabolic process / regulation of cell cycle / nuclear speck / positive regulation of apoptotic process / protein heterodimerization activity / DNA repair / apoptotic process / negative regulation of transcription by RNA polymerase II / protein homodimerization activity / nucleoplasm / nucleus / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / molecular dynamics | ||||||
| Model details | lowest energy, model 1 | ||||||
 データ登録者 データ登録者 | Sanchez, R. / Pantoja-Uceda, D. / Prieto, J. / Diercks, T. / Campos-Olivas, R. / Blanco, F.J. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2010 ジャーナル: J.Biol.Chem. / 年: 2010タイトル: Solution structure of human growth arrest and DNA damage 45alpha (Gadd45alpha) and its interactions with proliferating cell nuclear antigen (PCNA) and Aurora A kinase 著者: Sanchez, R. / Pantoja-Uceda, D. / Prieto, J. / Diercks, T. / Marcaida, M.J. / Montoya, G. / Campos-Olivas, R. / Blanco, F.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2kg4.cif.gz 2kg4.cif.gz | 986 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2kg4.ent.gz pdb2kg4.ent.gz | 827.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2kg4.json.gz 2kg4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2kg4_validation.pdf.gz 2kg4_validation.pdf.gz | 473.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2kg4_full_validation.pdf.gz 2kg4_full_validation.pdf.gz | 829.8 KB | 表示 | |
| XML形式データ |  2kg4_validation.xml.gz 2kg4_validation.xml.gz | 118.7 KB | 表示 | |
| CIF形式データ |  2kg4_validation.cif.gz 2kg4_validation.cif.gz | 110.8 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/kg/2kg4 https://data.pdbj.org/pub/pdb/validation_reports/kg/2kg4 ftp://data.pdbj.org/pub/pdb/validation_reports/kg/2kg4 ftp://data.pdbj.org/pub/pdb/validation_reports/kg/2kg4 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 18351.723 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: GADD45A / 発現宿主: Homo sapiens (ヒト) / 遺伝子: GADD45A / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj












 HSQC
HSQC