+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2jbl | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PHOTOSYNTHETIC REACTION CENTER FROM BLASTOCHLORIS VIRIDIS | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード | ELECTRON TRANSPORT / CHROMOPHORE / FORMYLATION / CHLOROPHYLL / LIPOPROTEIN / STIGMATELLIN / METAL-BINDING / TRANSMEMBRANE / IRON / HEME / MEMBRANE / TRANSPORT / MAGNESIUM / PHOTOSYNTHESIS / REACTION CENTER / BACTERIOCHLOROPHYLL / PHOTOSYNTHETIC REACTION CENTER | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / photosynthesis, light reaction / : / photosynthesis / electron transfer activity / iron ion binding / heme binding / metal ion binding 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  BLASTOCHLORIS VIRIDIS (バクテリア) BLASTOCHLORIS VIRIDIS (バクテリア) | ||||||||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.4 Å 分子置換 / 解像度: 2.4 Å | ||||||||||||
 データ登録者 データ登録者 | Lancaster, C.R.D. | ||||||||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2007 ジャーナル: J.Mol.Biol. / 年: 2007タイトル: A Comparison of Stigmatellin Conformations, Free and Bound to the Photosynthetic Reaction Center and the Cytochrome Bc(1) Complex. 著者: Lancaster, C.R.D. / Hunte, C. / Kelley, J. / Trumpower, B.L. / Ditchfield, R. #1:  ジャーナル: J.Biol.Chem. / 年: 2000 ジャーナル: J.Biol.Chem. / 年: 2000タイトル: Structural Basis of the Drastically Increased Initial Electron Transfer Rate in the Reaction Center from a Rhodopseudomonas Viridis Mutant Described at 2.00-A Resolution 著者: Lancaster, C.R.D. / Bibikova, M. / Sabatino, P. / Oesterhelt, D. / Michel, H. #2:  ジャーナル: J.Mol.Biol. / 年: 1999 ジャーナル: J.Mol.Biol. / 年: 1999タイトル: Refined Crystal Structures of Reaction Centres from Rhodopseudomonas Viridis in Complexes with the Herbicide Atrazine and Two Chiral Atrazine Derivatives Also Lead to a New Model of the Bound Carotenoid 著者: Lancaster, C.R. / Michel, H. #3:  ジャーナル: Biochim.Biophys.Acta / 年: 1998 ジャーナル: Biochim.Biophys.Acta / 年: 1998タイトル: Ubiquinone Reduction and Protonation in the Reaction Centre of Rhodopseudomonas Viridis: X-Ray Structures and Their Functional Implications 著者: Lancaster, C.R.D. #4:  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: The Coupling of Light-Induced Electron Transfer and Proton Uptake as Derived from Crystal Structures of Reaction Centres from Rhodopseudomonas Viridis Modified at the Binding Site of ...タイトル: The Coupling of Light-Induced Electron Transfer and Proton Uptake as Derived from Crystal Structures of Reaction Centres from Rhodopseudomonas Viridis Modified at the Binding Site of the Secondary Quinone, Qb 著者: Lancaster, C.R. / Michel, H. #5:  ジャーナル: J.Mol.Biol. / 年: 1995 ジャーナル: J.Mol.Biol. / 年: 1995タイトル: Crystallographic Refinement at 2.3 A Resolution and Refined Model of the Photosynthetic Reaction Centre from Rhodopseudomonas Viridis 著者: Deisenhofer, J. / Epp, O. / Sinning, I. / Michel, H. #6: ジャーナル: Science / 年: 1989 タイトル: The Photosynthetic Reaction Center from the Purple Bacterium Rhodopseudomonas Viridis 著者: Deisenhofer, J. / Michel, H. #7: ジャーナル: Nature / 年: 1985 タイトル: Structure of the Protein Subunits in the Photosynthetic Reaction Centre of Rhodopseudomonas Viridis at 3 Angstroms Resolution 著者: Deisenhofer, J. / Epp, O. / Miki, K. / Huber, R. / Michel, H. #8: ジャーナル: J.Mol.Biol. / 年: 1984 タイトル: X-Ray Structure Analysis of a Membrane Protein Complex. Electron Density Map at 3 A Resolution and a Model of the Chromophores of the Photosynthetic Reaction Center from Rhodopseudomonas Viridis 著者: Deisenhofer, J. / Epp, O. / Miki, K. / Huber, R. / Michel, H. #9: ジャーナル: J.Mol.Biol. / 年: 1982 タイトル: Three-Dimensional Crystals of a Membrane Protein Complex. The Photosynthetic Reaction Centre from Rhodopseudomonas Viridis 著者: Michel, H. | ||||||||||||
| 履歴 |
| ||||||||||||
| Remark 700 | SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN ... SHEET THE SHEET STRUCTURE OF THIS MOLECULE IS BIFURCATED. IN ORDER TO REPRESENT THIS FEATURE IN THE SHEET RECORDS BELOW, TWO SHEETS ARE DEFINED. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2jbl.cif.gz 2jbl.cif.gz | 287.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2jbl.ent.gz pdb2jbl.ent.gz | 224.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2jbl.json.gz 2jbl.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2jbl_validation.pdf.gz 2jbl_validation.pdf.gz | 3.7 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2jbl_full_validation.pdf.gz 2jbl_full_validation.pdf.gz | 3.7 MB | 表示 | |
| XML形式データ |  2jbl_validation.xml.gz 2jbl_validation.xml.gz | 52 KB | 表示 | |
| CIF形式データ |  2jbl_validation.cif.gz 2jbl_validation.cif.gz | 71.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jb/2jbl https://data.pdbj.org/pub/pdb/validation_reports/jb/2jbl ftp://data.pdbj.org/pub/pdb/validation_reports/jb/2jbl ftp://data.pdbj.org/pub/pdb/validation_reports/jb/2jbl | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 C
| #1: タンパク質 | 分子量: 39419.176 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM 133) / 由来: (天然)  BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P07173 BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P07173 |
|---|
-REACTION CENTER PROTEIN ... , 3種, 3分子 HLM
| #2: タンパク質 | 分子量: 28557.453 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM 133) / 由来: (天然)  BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P06008 BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P06008 |
|---|---|
| #3: タンパク質 | 分子量: 30469.104 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM 133) / 由来: (天然)  BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P06009 BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P06009 |
| #4: タンパク質 | 分子量: 35932.188 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: GERMAN COLLECTION OF MICROORGANISMS (DSM 133) / 由来: (天然)  BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P06010 BLASTOCHLORIS VIRIDIS (バクテリア) / 参照: UniProt: P06010 |
-非ポリマー , 10種, 366分子 


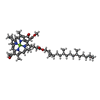
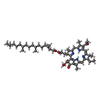
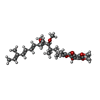













| #5: 化合物 | ChemComp-HEC / #6: 化合物 | ChemComp-LDA / #7: 化合物 | ChemComp-SO4 / #8: 化合物 | ChemComp-BCB / #9: 化合物 | #10: 化合物 | ChemComp-SMA / | #11: 化合物 | ChemComp-FE / | #12: 化合物 | ChemComp-MQ7 / | #13: 化合物 | ChemComp-NS5 / | #14: 水 | ChemComp-HOH / | |
|---|
-詳細
| Has protein modification | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 5 X線回折 / 使用した結晶の数: 5 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.82 Å3/Da / 溶媒含有率: 74.28 % |
|---|---|
| 結晶化 | pH: 6 / 詳細: pH 6.00 |
-データ収集
| 回折 | 平均測定温度: 263 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  EMBL/DESY, HAMBURG EMBL/DESY, HAMBURG  / ビームライン: X11 / 波長: 0.92 / ビームライン: X11 / 波長: 0.92 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1992年11月13日 / 詳細: FOCUSING MIRROR |
| 放射 | モノクロメーター: BENT SINGLE-CRYSTAL GERMANIUM TRIANGULAR MONOCHROMATOR プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.92 Å / 相対比: 1 |
| 反射 | 解像度: 2.4→26.56 Å / Num. obs: 103499 / % possible obs: 92.3 % / Observed criterion σ(I): -3 / 冗長度: 1.9 % / Biso Wilson estimate: 28.1 Å2 / Rmerge(I) obs: 0.09 / Net I/σ(I): 6.9 |
| 反射 シェル | 解像度: 2.4→2.46 Å / 冗長度: 1.4 % / Rmerge(I) obs: 0.31 / Mean I/σ(I) obs: 1.6 / % possible all: 86.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 4PRC  4prc 解像度: 2.4→26.56 Å / Rfactor Rfree error: 0.005 / Data cutoff high absF: 3307055.41 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: MLF
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 56.1587 Å2 / ksol: 0.285982 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 37.7 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.4→26.56 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.4→2.55 Å / Rfactor Rfree error: 0.02 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー



























 PDBj
PDBj




















