+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2hhn | ||||||
|---|---|---|---|---|---|---|---|
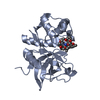
| Title | Cathepsin S in complex with non covalent arylaminoethyl amide. | ||||||
 Components Components | Cathepsin S | ||||||
 Keywords Keywords | HYDROLASE / cathepsin S / noncovalent / inhibition / arylaminoethyl amide / protease | ||||||
| Function / homology |  Function and homology information Function and homology informationcathepsin S / regulation of antigen processing and presentation / basement membrane disassembly / positive regulation of cation channel activity / antigen processing and presentation of peptide antigen / endolysosome lumen / cellular response to thyroid hormone stimulus / Trafficking and processing of endosomal TLR / proteoglycan binding / response to acidic pH ...cathepsin S / regulation of antigen processing and presentation / basement membrane disassembly / positive regulation of cation channel activity / antigen processing and presentation of peptide antigen / endolysosome lumen / cellular response to thyroid hormone stimulus / Trafficking and processing of endosomal TLR / proteoglycan binding / response to acidic pH / Assembly of collagen fibrils and other multimeric structures / toll-like receptor signaling pathway / antigen processing and presentation / collagen catabolic process / fibronectin binding / extracellular matrix disassembly / collagen binding / phagocytic vesicle / Degradation of the extracellular matrix / laminin binding / cysteine-type peptidase activity / MHC class II antigen presentation / lysosomal lumen / : / Endosomal/Vacuolar pathway / protein processing / : / antigen processing and presentation of exogenous peptide antigen via MHC class II / tertiary granule lumen / late endosome / ficolin-1-rich granule lumen / adaptive immune response / lysosome / immune response / intracellular membrane-bounded organelle / serine-type endopeptidase activity / cysteine-type endopeptidase activity / Neutrophil degranulation / proteolysis / extracellular space / extracellular region Similarity search - Function | ||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / RIGID BODY REFINEMENT / Resolution: 1.55 Å SYNCHROTRON / RIGID BODY REFINEMENT / Resolution: 1.55 Å | ||||||
 Authors Authors | Spraggon, G. / Hornsby, M. / Lesley, S.A. / Tully, D.C. / Harris, J.L. / Karenewsky, D.S. / Kulathila, R. / Clark, K. | ||||||
 Citation Citation |  Journal: Bioorg.Med.Chem.Lett. / Year: 2006 Journal: Bioorg.Med.Chem.Lett. / Year: 2006Title: Synthesis and SAR of arylaminoethyl amides as noncovalent inhibitors of cathepsin S: P3 cyclic ethers Authors: Tully, D.C. / Liu, H. / Chatterjee, A.K. / Alper, P.B. / Epple, R. / Williams, J.A. / Roberts, M.J. / Woodmansee, D.H. / Masick, B.T. / Tumanut, C. / Li, J. / Spraggon, G. / Hornsby, M. / ...Authors: Tully, D.C. / Liu, H. / Chatterjee, A.K. / Alper, P.B. / Epple, R. / Williams, J.A. / Roberts, M.J. / Woodmansee, D.H. / Masick, B.T. / Tumanut, C. / Li, J. / Spraggon, G. / Hornsby, M. / Chang, J. / Tuntland, T. / Hollenbeck, T. / Gordon, P. / Harris, J.L. / Karanewsky, D.S. #1:  Journal: Nat.Struct.Mol.Biol. / Year: 1999 Journal: Nat.Struct.Mol.Biol. / Year: 1999Title: Structural and mechanistic basis of immunity towards endonuclease colicins Authors: Kleanthous, C. / Kuhlmann, U.C. / Pommer, A.J. / Ferguson, N. / Radford, S.E. / Moore, G.R. / James, R. / Hemmings, A.M. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2hhn.cif.gz 2hhn.cif.gz | 113.6 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2hhn.ent.gz pdb2hhn.ent.gz | 87.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2hhn.json.gz 2hhn.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/hh/2hhn https://data.pdbj.org/pub/pdb/validation_reports/hh/2hhn ftp://data.pdbj.org/pub/pdb/validation_reports/hh/2hhn ftp://data.pdbj.org/pub/pdb/validation_reports/hh/2hhn | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  2hh5C  2f1gS C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||
| 2 | 
| |||||||||
| 3 | 
| |||||||||
| 4 | 
| |||||||||
| 5 | 
| |||||||||
| Unit cell |
| |||||||||
| Components on special symmetry positions |
| |||||||||
| Details | The natural state of the molecule is a monomer - there are two molecules of Cathepsin S in the asymmetric unit |
- Components
Components
| #1: Protein | Mass: 24391.393 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: CTSS / Production host: Homo sapiens (human) / Gene: CTSS / Production host:  #2: Chemical | ChemComp-SO4 / #3: Chemical | ChemComp-GNQ / | #4: Water | ChemComp-HOH / | Has protein modification | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.82 Å3/Da / Density % sol: 56.36 % |
|---|---|
| Crystal grow | Temperature: 278 K / Method: vapor diffusion, hanging drop / pH: 6.5 Details: 20% PEG-8000, 0.4M Lithium Sulfate, pH 6.5, VAPOR DIFFUSION, HANGING DROP, temperature 278K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  ALS ALS  / Beamline: 5.0.3 / Wavelength: 1 Å / Beamline: 5.0.3 / Wavelength: 1 Å |
| Detector | Type: ADSC QUANTUM 4 / Detector: CCD / Date: Aug 28, 2003 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 1.55→84.2 Å / Num. all: 73915 / Num. obs: 73915 / % possible obs: 96.2 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 3.54 % / Rmerge(I) obs: 0.058 / Rsym value: 0.058 / Net I/σ(I): 21.5 |
| Reflection shell | Resolution: 1.55→1.62 Å / Rmerge(I) obs: 0.509 / Mean I/σ(I) obs: 2.33 / Num. unique all: 6804 / Rsym value: 0.509 / % possible all: 86.3 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure: RIGID BODY REFINEMENT Starting model: 2F1G Resolution: 1.55→84.2 Å / Cor.coef. Fo:Fc: 0.967 / Cor.coef. Fo:Fc free: 0.959 / SU B: 1.184 / SU ML: 0.043 / Cross valid method: THROUGHOUT / σ(F): 0 / σ(I): 0 / ESU R: 0.067 / ESU R Free: 0.068 / Stereochemistry target values: MAXIMUM LIKELIHOOD Details: Hydrogens have been added in the riding positions. The density for chain A is too weak to reliably place the ligand (a low occupancy ligand). For chain B all atoms are present but density ...Details: Hydrogens have been added in the riding positions. The density for chain A is too weak to reliably place the ligand (a low occupancy ligand). For chain B all atoms are present but density for the 4-(TRIFLUOROMETHOXY)PHENYL AMINO group is largely missing. Those atoms have been labelled as occupancy 0.00 and are included on for comparison with 2HH5 structure.
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: BABINET MODEL WITH MASK | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 12.839 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.55→84.2 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 1.554→1.594 Å / Total num. of bins used: 20
|
 Movie
Movie Controller
Controller












 PDBj
PDBj