| 登録情報 | データベース: PDB / ID: 1wa7
|
|---|
| タイトル | SH3 DOMAIN OF HUMAN LYN TYROSINE KINASE IN COMPLEX WITH A HERPESVIRAL LIGAND |
|---|
 要素 要素 | - HYPOTHETICAL 28.7 KDA PROTEIN IN DHFR 3'REGION (ORF1)
- TYROSINE-PROTEIN KINASE LYN
|
|---|
 キーワード キーワード | SH3 DOMAIN / LIGAND / TYROSINE KINASE / SIGNAL TRANSDUCTION / LYN / TIP / PROTO-ONCOGENE / HYPOTHETICAL PROTEIN |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of myeloid leukocyte differentiation / C-X-C chemokine receptor CXCR4 signaling pathway / regulation of monocyte chemotaxis / response to sterol depletion / regulation of mast cell activation / eosinophil differentiation / positive regulation of dendritic cell apoptotic process / Fc receptor mediated stimulatory signaling pathway / positive regulation of Fc receptor mediated stimulatory signaling pathway / negative regulation of intracellular signal transduction ...negative regulation of myeloid leukocyte differentiation / C-X-C chemokine receptor CXCR4 signaling pathway / regulation of monocyte chemotaxis / response to sterol depletion / regulation of mast cell activation / eosinophil differentiation / positive regulation of dendritic cell apoptotic process / Fc receptor mediated stimulatory signaling pathway / positive regulation of Fc receptor mediated stimulatory signaling pathway / negative regulation of intracellular signal transduction / Fc receptor mediated inhibitory signaling pathway / regulation of B cell receptor signaling pathway / negative regulation of toll-like receptor 2 signaling pathway / positive regulation of oligodendrocyte progenitor proliferation / integrin alpha2-beta1 complex / tolerance induction to self antigen / negative regulation of mast cell proliferation / immune response-regulating cell surface receptor signaling pathway / positive regulation of mast cell proliferation / glycosphingolipid binding / regulation of mast cell degranulation / platelet degranulation / negative regulation of toll-like receptor 4 signaling pathway / positive regulation of amyloid precursor protein catabolic process / negative regulation of immune response / regulation of platelet aggregation / phosphorylation-dependent protein binding / dendritic cell differentiation / regulation of B cell apoptotic process / neuroinflammatory response / Signaling by Erythropoietin / phosphatidylinositol 3-kinase activator activity / Erythropoietin activates STAT5 / interleukin-5-mediated signaling pathway / Erythropoietin activates Phospholipase C gamma (PLCG) / Platelet Adhesion to exposed collagen / histamine secretion by mast cell / CD22 mediated BCR regulation / regulation of release of sequestered calcium ion into cytosol / oligodendrocyte development / gamma-tubulin binding / response to carbohydrate / platelet-derived growth factor receptor binding / Co-stimulation by CD28 / Fc epsilon receptor (FCERI) signaling / EPH-Ephrin signaling / postsynaptic specialization, intracellular component / Regulation of KIT signaling / toll-like receptor 4 signaling pathway / leukocyte migration / Co-inhibition by CTLA4 / Erythropoietin activates Phosphoinositide-3-kinase (PI3K) / EPHA-mediated growth cone collapse / negative regulation of B cell proliferation / Dectin-2 family / mitochondrial crista / Fc-epsilon receptor signaling pathway / regulation of cell adhesion mediated by integrin / stimulatory C-type lectin receptor signaling pathway / Fc-gamma receptor signaling pathway involved in phagocytosis / negative regulation of protein phosphorylation / negative regulation of MAP kinase activity / regulation of protein phosphorylation / PECAM1 interactions / B cell homeostasis / signaling receptor activator activity / FCGR activation / positive regulation of phosphorylation / positive regulation of glial cell proliferation / growth hormone receptor signaling pathway via JAK-STAT / EPH-ephrin mediated repulsion of cells / Role of LAT2/NTAL/LAB on calcium mobilization / ephrin receptor signaling pathway / response to amino acid / fatty acid transport / Growth hormone receptor signaling / regulation of ERK1 and ERK2 cascade / hematopoietic progenitor cell differentiation / response to axon injury / Erythropoietin activates RAS / ephrin receptor binding / GPVI-mediated activation cascade / Signaling by CSF3 (G-CSF) / T cell costimulation / response to hormone / cellular response to retinoic acid / EPHB-mediated forward signaling / FCERI mediated Ca+2 mobilization / CD209 (DC-SIGN) signaling / regulation of cytokine production / FCGR3A-mediated IL10 synthesis / cell surface receptor protein tyrosine kinase signaling pathway / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / peptidyl-tyrosine phosphorylation / lipopolysaccharide-mediated signaling pathway / Signaling by phosphorylated juxtamembrane, extracellular and kinase domain KIT mutants / DNA damage checkpoint signaling / erythrocyte differentiation / Cell surface interactions at the vascular wall / B cell receptor signaling pathway類似検索 - 分子機能 Tyrosine-protein kinase Lyn, SH3 domain / Tyrosine-protein kinase Lyn, SH2 domain / SH3 Domains / : / SH3 domain / SH2 domain / Src homology 2 (SH2) domain profile. / Src homology 2 domains / SH2 domain / SH3 type barrels. ...Tyrosine-protein kinase Lyn, SH3 domain / Tyrosine-protein kinase Lyn, SH2 domain / SH3 Domains / : / SH3 domain / SH2 domain / Src homology 2 (SH2) domain profile. / Src homology 2 domains / SH2 domain / SH3 type barrels. / Src homology 3 domains / SH2 domain superfamily / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. / SH3 domain / Tyrosine-protein kinase, catalytic domain / Tyrosine kinase, catalytic domain / Tyrosine protein kinases specific active-site signature. / Tyrosine-protein kinase, active site / Serine-threonine/tyrosine-protein kinase, catalytic domain / Protein tyrosine and serine/threonine kinase / Roll / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily / Mainly Beta類似検索 - ドメイン・相同性 Tyrosine-protein kinase Lyn / Tyrosine-protein kinase-interacting protein類似検索 - 構成要素 |
|---|
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト)
 SAIMIRIINE HERPESVIRUS 2 (ヘルペスウイルス) SAIMIRIINE HERPESVIRUS 2 (ヘルペスウイルス) |
|---|
| 手法 | 溶液NMR / XPLOR |
|---|
 データ登録者 データ登録者 | Bauer, F. / Schweimer, K. / Hoffmann, S. / Roesch, P. / Sticht, H. |
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2002 ジャーナル: Biochemistry / 年: 2002
タイトル: Structural Investigation of the Binding of a Herpesviral Protein to the SH3 Domain of Tyrosine Kinase Lck.
著者: Schweimer, K. / Hoffmann, S. / Bauer, F. / Friedrich, U. / Kardinal, C. / Feller, S.M. / Biesinger, B. / Sticht, H. |
|---|
| 履歴 | | 登録 | 2004年10月25日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2005年7月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年5月8日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2024年5月15日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) SAIMIRIINE HERPESVIRUS 2 (ヘルペスウイルス)
SAIMIRIINE HERPESVIRUS 2 (ヘルペスウイルス) データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2002
ジャーナル: Biochemistry / 年: 2002 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1wa7.cif.gz
1wa7.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1wa7.ent.gz
pdb1wa7.ent.gz PDB形式
PDB形式 1wa7.json.gz
1wa7.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1wa7_validation.pdf.gz
1wa7_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1wa7_full_validation.pdf.gz
1wa7_full_validation.pdf.gz 1wa7_validation.xml.gz
1wa7_validation.xml.gz 1wa7_validation.cif.gz
1wa7_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/wa/1wa7
https://data.pdbj.org/pub/pdb/validation_reports/wa/1wa7 ftp://data.pdbj.org/pub/pdb/validation_reports/wa/1wa7
ftp://data.pdbj.org/pub/pdb/validation_reports/wa/1wa7 リンク
リンク 集合体
集合体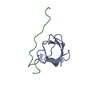
 要素
要素 HOMO SAPIENS (ヒト) / Cell: INTRACELLULAR / プラスミド: PGEX-6P-2 / 発現宿主:
HOMO SAPIENS (ヒト) / Cell: INTRACELLULAR / プラスミド: PGEX-6P-2 / 発現宿主: 
 SAIMIRIINE HERPESVIRUS 2 (ヘルペスウイルス)
SAIMIRIINE HERPESVIRUS 2 (ヘルペスウイルス)
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj




















 X-PLOR
X-PLOR