| 登録情報 | データベース: PDB / ID: 1tf0
|
|---|
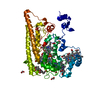
| タイトル | Crystal structure of the GA module complexed with human serum albumin |
|---|
 要素 要素 | - Peptostreptococcal albumin-binding protein
- Serum albumin
|
|---|
 キーワード キーワード | PROTEIN BINDING / PROTEIN-PROTEIN COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Ciprofloxacin ADME / exogenous protein binding / cellular response to calcium ion starvation / enterobactin binding / Heme biosynthesis / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME ...Ciprofloxacin ADME / exogenous protein binding / cellular response to calcium ion starvation / enterobactin binding / Heme biosynthesis / HDL remodeling / negative regulation of mitochondrial depolarization / Prednisone ADME / Heme degradation / Aspirin ADME / antioxidant activity / toxic substance binding / Scavenging of heme from plasma / Recycling of bile acids and salts / platelet alpha granule lumen / cellular response to starvation / fatty acid binding / Post-translational protein phosphorylation / Cytoprotection by HMOX1 / Regulation of Insulin-like Growth Factor (IGF) transport and uptake by Insulin-like Growth Factor Binding Proteins (IGFBPs) / pyridoxal phosphate binding / Platelet degranulation / protein-folding chaperone binding / blood microparticle / copper ion binding / endoplasmic reticulum lumen / endoplasmic reticulum / Golgi apparatus / protein-containing complex / DNA binding / extracellular space / extracellular exosome / extracellular region / identical protein binding / nucleus / cytoplasm類似検索 - 分子機能 Albumin-binding domain / Repeat of unknown function DUF5633 / Family of unknown function (DUF5633) / GA-like domain / GA-like domain / Serum Albumin; Chain A, Domain 1 - #10 / Serum albumin/Alpha-fetoprotein/Afamin / ALB/AFP/VDB / Serum albumin, N-terminal / Serum albumin, conserved site ...Albumin-binding domain / Repeat of unknown function DUF5633 / Family of unknown function (DUF5633) / GA-like domain / GA-like domain / Serum Albumin; Chain A, Domain 1 - #10 / Serum albumin/Alpha-fetoprotein/Afamin / ALB/AFP/VDB / Serum albumin, N-terminal / Serum albumin, conserved site / Serum albumin-like / Serum albumin family / Albumin domain signature. / Albumin domain profile. / serum albumin / Immunoglobulin/albumin-binding domain superfamily / Serum Albumin; Chain A, Domain 1 / LPXTG cell wall anchor motif / Gram-positive cocci surface proteins LPxTG motif profile. / LPXTG cell wall anchor domain / Helicase, Ruva Protein; domain 3 / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 CITRIC ACID / DECANOIC ACID / Albumin / Peptostreptococcal albumin-binding protein類似検索 - 構成要素 |
|---|
| 生物種 |  Finegoldia magna (バクテリア) Finegoldia magna (バクテリア)
 Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.7 Å 分子置換 / 解像度: 2.7 Å |
|---|
 データ登録者 データ登録者 | Lejon, S. / Svensson, S. / Bjorck, L. / Frick, I.-M. / Wikstrom, M. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2004 ジャーナル: J.Biol.Chem. / 年: 2004
タイトル: Crystal structure and biological implications of a bacterial albumin binding module in complex with human serum albumin
著者: Lejon, S. / Frick, I.-M. / Bjorck, L. / Wikstrom, M. / Svensson, S. |
|---|
| 履歴 | | 登録 | 2004年5月26日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2004年9月7日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月30日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Advisory / Source and taxonomy / Version format compliance |
|---|
| 改定 1.3 | 2017年10月11日 | Group: Refinement description / カテゴリ: software / Item: _software.classification / _software.name |
|---|
| 改定 1.4 | 2023年8月23日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
| 改定 1.5 | 2024年10月9日 | Group: Structure summary
カテゴリ: pdbx_entry_details / pdbx_modification_feature |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Finegoldia magna (バクテリア)
Finegoldia magna (バクテリア) Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.7 Å
分子置換 / 解像度: 2.7 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2004
ジャーナル: J.Biol.Chem. / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1tf0.cif.gz
1tf0.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1tf0.ent.gz
pdb1tf0.ent.gz PDB形式
PDB形式 1tf0.json.gz
1tf0.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/tf/1tf0
https://data.pdbj.org/pub/pdb/validation_reports/tf/1tf0 ftp://data.pdbj.org/pub/pdb/validation_reports/tf/1tf0
ftp://data.pdbj.org/pub/pdb/validation_reports/tf/1tf0 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / Tissue fraction: plasma / 参照: UniProt: P02768
Homo sapiens (ヒト) / Tissue fraction: plasma / 参照: UniProt: P02768 Finegoldia magna (バクテリア) / 株: ATCC 29328 / 遺伝子: PAB / プラスミド: pHD389 / 発現宿主:
Finegoldia magna (バクテリア) / 株: ATCC 29328 / 遺伝子: PAB / プラスミド: pHD389 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-4 / 波長: 1 Å
/ ビームライン: ID14-4 / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj