+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1ovz | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Crystal structure of human FcaRI | |||||||||
 Components Components | Immunoglobulin alpha Fc receptor | |||||||||
 Keywords Keywords | IMMUNE SYSTEM / FcaRI / CD89 / IgA / Fc receptor / immunoglobulin-like domain | |||||||||
| Function / homology |  Function and homology information Function and homology informationIgA receptor activity / cellular response to interferon-alpha / Fc receptor signaling pathway / IgA binding / immune response-regulating signaling pathway / positive regulation of neutrophil apoptotic process / neutrophil activation / cellular response to granulocyte macrophage colony-stimulating factor stimulus / neutrophil mediated immunity / cellular response to interleukin-6 ...IgA receptor activity / cellular response to interferon-alpha / Fc receptor signaling pathway / IgA binding / immune response-regulating signaling pathway / positive regulation of neutrophil apoptotic process / neutrophil activation / cellular response to granulocyte macrophage colony-stimulating factor stimulus / neutrophil mediated immunity / cellular response to interleukin-6 / tertiary granule membrane / ficolin-1-rich granule membrane / specific granule membrane / cellular response to type II interferon / cellular response to tumor necrosis factor / cellular response to lipopolysaccharide / immune response / Neutrophil degranulation / extracellular region / plasma membrane Similarity search - Function | |||||||||
| Biological species |  Homo sapiens (human) Homo sapiens (human) | |||||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MIR / Resolution: 3 Å MIR / Resolution: 3 Å | |||||||||
 Authors Authors | Herr, A.B. / Ballister, E.R. / Bjorkman, P.J. | |||||||||
 Citation Citation |  Journal: Nature / Year: 2003 Journal: Nature / Year: 2003Title: Insights into IgA-mediated immune responses from the crystal structures of human Fc-alpha-RI and its complex with IgA1-Fc Authors: Herr, A.B. / Ballister, E.R. / Bjorkman, P.J. #1:  Journal: J.Mol.Biol. / Year: 2003 Journal: J.Mol.Biol. / Year: 2003Title: Bivalent Binding of IgA1 to FcaRI Suggests a Mechanism for Cytokine Activation of IgA Phagocytosis Authors: Herr, A.B. / White, C.L. / Milburn, C. / Wu, C. / Bjorkman, P.J. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1ovz.cif.gz 1ovz.cif.gz | 82.1 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1ovz.ent.gz pdb1ovz.ent.gz | 61.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1ovz.json.gz 1ovz.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ov/1ovz https://data.pdbj.org/pub/pdb/validation_reports/ov/1ovz ftp://data.pdbj.org/pub/pdb/validation_reports/ov/1ovz ftp://data.pdbj.org/pub/pdb/validation_reports/ov/1ovz | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 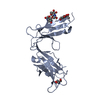
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
| ||||||||
| Details | The biological assembly state is a monomer. |
- Components
Components
| #1: Protein | Mass: 23615.742 Da / Num. of mol.: 2 / Fragment: ectodomain Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Homo sapiens (human) / Gene: FCAR OR CD89 / Plasmid: pAcGP67 / Production host: Homo sapiens (human) / Gene: FCAR OR CD89 / Plasmid: pAcGP67 / Production host:  Trichoplusia ni (cabbage looper) / Strain (production host): High 5 insect cells / References: UniProt: P24071 Trichoplusia ni (cabbage looper) / Strain (production host): High 5 insect cells / References: UniProt: P24071#2: Polysaccharide | 2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose | Source method: isolated from a genetically manipulated source #3: Sugar | #4: Chemical | ChemComp-TRS / | Has protein modification | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.36 Å3/Da / Density % sol: 49.1 % | ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 296 K / Method: vapor diffusion, hanging drop / pH: 6 Details: PEG 8000, MES, pH 6.0, VAPOR DIFFUSION, HANGING DROP, temperature 296K | ||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Method: vapor diffusion, hanging drop | ||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  SSRL SSRL  / Beamline: BL9-2 / Wavelength: 1 Å / Beamline: BL9-2 / Wavelength: 1 Å |
| Detector | Type: ADSC QUANTUM 4 / Detector: CCD / Date: Apr 26, 2001 |
| Radiation | Monochromator: double crystal monochromator / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 3→25 Å / Num. all: 10246 / Num. obs: 10204 / % possible obs: 99.6 % / Observed criterion σ(I): -3 / Redundancy: 15.1 % / Biso Wilson estimate: 56.4 Å2 / Rmerge(I) obs: 0.077 / Net I/σ(I): 22.4 |
| Reflection shell | Resolution: 3→3.05 Å / Rmerge(I) obs: 0.404 / Mean I/σ(I) obs: 4.9 / Num. unique all: 517 / % possible all: 100 |
| Reflection | *PLUS Num. obs: 10246 / % possible obs: 99.7 % / Num. measured all: 154491 |
| Reflection shell | *PLUS % possible obs: 100 % |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MIR / Resolution: 3→24.79 Å / Rfactor Rfree error: 0.009 / Isotropic thermal model: GROUP / Cross valid method: THROUGHOUT / σ(F): 0 / Stereochemistry target values: Engh & Huber MIR / Resolution: 3→24.79 Å / Rfactor Rfree error: 0.009 / Isotropic thermal model: GROUP / Cross valid method: THROUGHOUT / σ(F): 0 / Stereochemistry target values: Engh & Huber
| |||||||||||||||||||||||||
| Solvent computation | Solvent model: FLAT MODEL / Bsol: 38.5159 Å2 / ksol: 0.335763 e/Å3 | |||||||||||||||||||||||||
| Displacement parameters | Biso mean: 52.3 Å2
| |||||||||||||||||||||||||
| Refine analyze |
| |||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 3→24.79 Å
| |||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: CONSTR | |||||||||||||||||||||||||
| LS refinement shell | Resolution: 3→3.11 Å / Rfactor Rfree error: 0.046 / Total num. of bins used: 10
| |||||||||||||||||||||||||
| Xplor file |
| |||||||||||||||||||||||||
| Refinement | *PLUS Highest resolution: 3 Å / Lowest resolution: 25 Å / Rfactor Rfree: 0.291 | |||||||||||||||||||||||||
| Solvent computation | *PLUS | |||||||||||||||||||||||||
| Displacement parameters | *PLUS | |||||||||||||||||||||||||
| Refine LS restraints | *PLUS
|
 Movie
Movie Controller
Controller














 PDBj
PDBj








