| 登録情報 | データベース: PDB / ID: 1jei
|
|---|
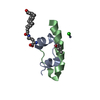
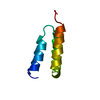
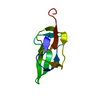
| タイトル | LEM DOMAIN OF HUMAN INNER NUCLEAR MEMBRANE PROTEIN EMERIN |
|---|
 要素 要素 | EMERIN |
|---|
 キーワード キーワード | MEMBRANE PROTEIN / emerin nucleus membrane domain dystrophy |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
TMEM240-body / nuclear membrane organization / Depolymerization of the Nuclear Lamina / Nuclear Envelope Breakdown / Initiation of Nuclear Envelope (NE) Reformation / RHOD GTPase cycle / nuclear inner membrane / regulation of canonical Wnt signaling pathway / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / muscle organ development ...TMEM240-body / nuclear membrane organization / Depolymerization of the Nuclear Lamina / Nuclear Envelope Breakdown / Initiation of Nuclear Envelope (NE) Reformation / RHOD GTPase cycle / nuclear inner membrane / regulation of canonical Wnt signaling pathway / Insertion of tail-anchored proteins into the endoplasmic reticulum membrane / muscle organ development / nuclear outer membrane / skeletal muscle cell differentiation / RHOG GTPase cycle / RAC2 GTPase cycle / RAC3 GTPase cycle / beta-tubulin binding / negative regulation of fibroblast proliferation / RAC1 GTPase cycle / muscle contraction / positive regulation of protein export from nucleus / negative regulation of canonical Wnt signaling pathway / cellular response to growth factor stimulus / spindle / nuclear envelope / actin binding / nuclear membrane / amyloid fibril formation / microtubule / cadherin binding / endoplasmic reticulum / nucleoplasm / membrane / cytosol / cytoplasm類似検索 - 分子機能 Emerin, LEM domain / Emerin / Transcription Termination Factor Rho, Rna-binding Domain; Chain A, Domain 1 - #40 / LEM domain / LEM domain / LEM domain profile. / in nuclear membrane-associated proteins / LEM/LEM-like domain superfamily / Transcription Termination Factor Rho, Rna-binding Domain; Chain A, Domain 1 / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 |
|---|
| 手法 | 溶液NMR / semi-automated iterative procedure using X-PLOR, home-written C-shell programs |
|---|
 データ登録者 データ登録者 | Wolff, N. / Gilquin, B. / Courchay, K. / Callebaut, I. / Zinn-Justin, S. |
|---|
 引用 引用 | |
|---|
| 履歴 | | 登録 | 2001年6月18日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2001年7月4日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2007年10月21日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2022年2月23日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name |
|---|
| 改定 1.4 | 2024年5月29日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: FEBS LETT. / 年: 2001
ジャーナル: FEBS LETT. / 年: 2001 ジャーナル: Structure / 年: 2001
ジャーナル: Structure / 年: 2001 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1jei.cif.gz
1jei.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1jei.ent.gz
pdb1jei.ent.gz PDB形式
PDB形式 1jei.json.gz
1jei.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1jei_validation.pdf.gz
1jei_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1jei_full_validation.pdf.gz
1jei_full_validation.pdf.gz 1jei_validation.xml.gz
1jei_validation.xml.gz 1jei_validation.cif.gz
1jei_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/je/1jei
https://data.pdbj.org/pub/pdb/validation_reports/je/1jei ftp://data.pdbj.org/pub/pdb/validation_reports/je/1jei
ftp://data.pdbj.org/pub/pdb/validation_reports/je/1jei リンク
リンク 集合体
集合体
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







 X-PLOR
X-PLOR