+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1dh3 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURE OF A CREB BZIP-CRE COMPLEX REVEALS THE BASIS FOR CREB FAIMLY SELECTIVE DIMERIZATION AND DNA BINDING | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSCRIPTION/DNA / PROTEIN-DNA COMPLEX / TRANSCRIPTION-DNA COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報AKT phosphorylates targets in the nucleus / NGF-stimulated transcription / chemotaxis to arachidonate / positive regulation of membrane depolarization / response to erythropoietin / ATF4-CREB1 transcription factor complex / CREB phosphorylation / CREB1 phosphorylation through NMDA receptor-mediated activation of RAS signaling / Gastrin-CREB signalling pathway via PKC and MAPK / Estrogen-dependent nuclear events downstream of ESR-membrane signaling ...AKT phosphorylates targets in the nucleus / NGF-stimulated transcription / chemotaxis to arachidonate / positive regulation of membrane depolarization / response to erythropoietin / ATF4-CREB1 transcription factor complex / CREB phosphorylation / CREB1 phosphorylation through NMDA receptor-mediated activation of RAS signaling / Gastrin-CREB signalling pathway via PKC and MAPK / Estrogen-dependent nuclear events downstream of ESR-membrane signaling / positive regulation of transforming growth factor beta3 production / lung saccule development / response to dehydroepiandrosterone / NCAM signaling for neurite out-growth / cAMP response element binding / response to hypobaric hypoxia / secretory granule organization / positive regulation of cardiac muscle tissue development / regulation of glial cell proliferation / lung epithelium development / inhibition of non-skeletal tissue mineralization / pituitary gland development / regulation of fibroblast proliferation / regulation of testosterone biosynthetic process / response to purine-containing compound / negative regulation of transcription by competitive promoter binding / hormone secretion / positive regulation of hormone secretion / mammary gland development / positive regulation of multicellular organism growth / arrestin family protein binding / positive regulation of osteoclast differentiation / cellular response to fatty acid / response to L-glutamate / response to glucagon / response to morphine / cellular response to hepatocyte growth factor stimulus / cellular response to insulin-like growth factor stimulus / histone acetyltransferase binding / regulation of cell size / cellular response to zinc ion / type I pneumocyte differentiation / cAMP/PKA signal transduction / positive regulation of RNA polymerase II transcription preinitiation complex assembly / positive regulation of fat cell differentiation / positive regulation of lipid biosynthetic process / cellular response to retinoic acid / cellular response to platelet-derived growth factor stimulus / lactation / Hsp70 protein binding / cellular response to forskolin / transforming growth factor beta receptor signaling pathway / axonogenesis / osteoclast differentiation / cellular response to leukemia inhibitory factor / positive regulation of long-term synaptic potentiation / response to activity / response to nicotine / response to cocaine / RNA polymerase II transcription regulatory region sequence-specific DNA binding / mRNA transcription by RNA polymerase II / euchromatin / circadian rhythm / cellular response to nerve growth factor stimulus / visual learning / cellular response to growth factor stimulus / RNA polymerase II transcription regulator complex / transcription coactivator binding / memory / DNA-binding transcription activator activity, RNA polymerase II-specific / response to ethanol / transcription regulator complex / sequence-specific DNA binding / RNA polymerase II-specific DNA-binding transcription factor binding / protein stabilization / ciliary basal body / positive regulation of apoptotic process / mitochondrial matrix / response to xenobiotic stimulus / DNA-binding transcription factor activity / axon / negative regulation of gene expression / centrosome / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / negative regulation of apoptotic process / chromatin / positive regulation of DNA-templated transcription / positive regulation of transcription by RNA polymerase II / DNA binding / nucleoplasm / identical protein binding / nucleus / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / 解像度: 3 Å シンクロトロン / 解像度: 3 Å | ||||||
 データ登録者 データ登録者 | Schumacher, M.A. / Goodman, R.H. / Brennan, R.G. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2000 ジャーナル: J.Biol.Chem. / 年: 2000タイトル: The structure of a CREB bZIP.somatostatin CRE complex reveals the basis for selective dimerization and divalent cation-enhanced DNA binding 著者: Schumacher, M.A. / Goodman, R.H. / Brennan, R.G. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1dh3.cif.gz 1dh3.cif.gz | 60.1 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1dh3.ent.gz pdb1dh3.ent.gz | 41.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1dh3.json.gz 1dh3.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1dh3_validation.pdf.gz 1dh3_validation.pdf.gz | 386 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1dh3_full_validation.pdf.gz 1dh3_full_validation.pdf.gz | 415.3 KB | 表示 | |
| XML形式データ |  1dh3_validation.xml.gz 1dh3_validation.xml.gz | 8.2 KB | 表示 | |
| CIF形式データ |  1dh3_validation.cif.gz 1dh3_validation.cif.gz | 11.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/dh/1dh3 https://data.pdbj.org/pub/pdb/validation_reports/dh/1dh3 ftp://data.pdbj.org/pub/pdb/validation_reports/dh/1dh3 ftp://data.pdbj.org/pub/pdb/validation_reports/dh/1dh3 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 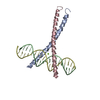
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 6424.148 Da / 分子数: 2 / 由来タイプ: 合成 #2: タンパク質 | 分子量: 6686.831 Da / 分子数: 2 / Fragment: RESIDUES 201-255 / 由来タイプ: 組換発現 / 由来: (組換発現)   #3: 化合物 | ChemComp-MG / | #4: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.42 Å3/Da / 溶媒含有率: 64.01 % | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 298 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.6 詳細: PEG 8000, MGCL2, (NH4)2SO4, MES, pH 5.6, VAPOR DIFFUSION, HANGING DROP, temperature 298.K | |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| |||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 手法: 蒸気拡散法 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 298 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL7-1 / 波長: 1.08 / ビームライン: BL7-1 / 波長: 1.08 |
| 検出器 | 検出器: IMAGE PLATE |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.08 Å / 相対比: 1 |
| 反射 | 解像度: 3→10 Å / Num. all: 7436 / Num. obs: 7316 / % possible obs: 90 % / Observed criterion σ(I): 1 / 冗長度: 5.7 % / Biso Wilson estimate: 40 Å2 / Rmerge(I) obs: 0.079 / Net I/σ(I): 17 |
| 反射 シェル | 解像度: 3→3.2 Å / 冗長度: 5 % / Rmerge(I) obs: 0.258 / % possible all: 95 |
| 反射 | *PLUS % possible obs: 95 % / 冗長度: 4.3 % / Rmerge(I) obs: 0.065 |
| 反射 シェル | *PLUS % possible obs: 87.4 % / Mean I/σ(I) obs: 3.6 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 3→10 Å / 交差検証法: THROUGHOUT / σ(F): 2 / σ(I): 1 詳細: THE FIRST AND LAST TWO RESIDUES ARE DISORDERED AND NOT MODELED. THE DENSITY IS WEAK FOR RESIDUES 334-339 IN BOTH PROTEIN CHAINS. PROCHECK REVEALED NO BAD CONTACTS AND NO RAMACHANDRAN OUTLIERS
| ||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3→10 Å
| ||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: TNT / 分類: refinement | ||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 3 Å / 最低解像度: 10 Å / σ(F): 2 / % reflection Rfree: 10 % / Rfactor Rfree: 0.28 | ||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj










































