+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1d4w | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURE OF THE XLP PROTEIN SAP IN COMPLEX WITH SLAM PHOSPHOPEPTIDE | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | SIGNALING PROTEIN / SH2 DOMAIN / PHOSPHOTYROSINE RECOGNIITON / PEPTIDE RECOGNITION / SIGNAL TRANSDUCTION / LYMPHOPROLIFERATIVE DISEASE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報natural killer cell proliferation / negative regulation of CD40 signaling pathway / myeloid dendritic cell activation involved in immune response / regulation of vesicle fusion / negative regulation of T cell cytokine production / leukocyte chemotaxis involved in inflammatory response / positive regulation of dendritic cell chemotaxis / positive regulation of T-helper 1 cell cytokine production / natural killer cell differentiation / positive regulation of natural killer cell mediated cytotoxicity ...natural killer cell proliferation / negative regulation of CD40 signaling pathway / myeloid dendritic cell activation involved in immune response / regulation of vesicle fusion / negative regulation of T cell cytokine production / leukocyte chemotaxis involved in inflammatory response / positive regulation of dendritic cell chemotaxis / positive regulation of T-helper 1 cell cytokine production / natural killer cell differentiation / positive regulation of natural killer cell mediated cytotoxicity / negative regulation of interleukin-12 production / natural killer cell activation / negative regulation of T cell receptor signaling pathway / natural killer cell mediated cytotoxicity / positive regulation of macrophage chemotaxis / positive regulation of activated T cell proliferation / negative regulation of type II interferon production / negative regulation of interleukin-6 production / humoral immune response / negative regulation of tumor necrosis factor production / phagocytosis / regulation of immune response / cellular defense response / antigen binding / phagocytic vesicle / SH2 domain binding / positive regulation of JNK cascade / positive regulation of type II interferon production / Immunoregulatory interactions between a Lymphoid and a non-Lymphoid cell / transmembrane signaling receptor activity / cell-cell signaling / signaling receptor activity / virus receptor activity / protein-macromolecule adaptor activity / adaptive immune response / positive regulation of ERK1 and ERK2 cascade / cell adhesion / immune response / innate immune response / external side of plasma membrane / positive regulation of cell population proliferation / extracellular exosome / identical protein binding / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / 解像度: 1.8 Å X線回折 / 解像度: 1.8 Å | ||||||
 データ登録者 データ登録者 | Poy, F. / Yaffe, M.B. / Sayos, J. / Saxena, K. / Eck, M.J. | ||||||
 引用 引用 |  ジャーナル: Mol.Cell / 年: 1999 ジャーナル: Mol.Cell / 年: 1999タイトル: Crystal structures of the XLP protein SAP reveal a class of SH2 domains with extended, phosphotyrosine-independent sequence recognition. 著者: Poy, F. / Yaffe, M.B. / Sayos, J. / Saxena, K. / Morra, M. / Sumegi, J. / Cantley, L.C. / Terhorst, C. / Eck, M.J. #1:  ジャーナル: Nature / 年: 1998 ジャーナル: Nature / 年: 1998タイトル: The X-linked lymphoproliferative disease gene product SAP regulates signals induced through the co-receptor SLAM 著者: Sayos, J. / Wu, C. / Morra, M. / Wang, N. / Terhorst, C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1d4w.cif.gz 1d4w.cif.gz | 66.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1d4w.ent.gz pdb1d4w.ent.gz | 48.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1d4w.json.gz 1d4w.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1d4w_validation.pdf.gz 1d4w_validation.pdf.gz | 443.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1d4w_full_validation.pdf.gz 1d4w_full_validation.pdf.gz | 444.8 KB | 表示 | |
| XML形式データ |  1d4w_validation.xml.gz 1d4w_validation.xml.gz | 18.4 KB | 表示 | |
| CIF形式データ |  1d4w_validation.cif.gz 1d4w_validation.cif.gz | 26.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/d4/1d4w https://data.pdbj.org/pub/pdb/validation_reports/d4/1d4w ftp://data.pdbj.org/pub/pdb/validation_reports/d4/1d4w ftp://data.pdbj.org/pub/pdb/validation_reports/d4/1d4w | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 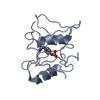
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 11702.393 Da / 分子数: 2 / 断片: SH2 DOMAIN / 変異: RESIDUES 1-104 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 組織: BLOOD / Cell: T CELL / プラスミド: T7-PRSET / 発現宿主: Homo sapiens (ヒト) / 組織: BLOOD / Cell: T CELL / プラスミド: T7-PRSET / 発現宿主:  #2: タンパク質・ペプチド | 分子量: 1360.491 Da / 分子数: 2 / 断片: CYTOPLASMIC TAIL SYNTHETIC PHOSPOPEPTIDE / 由来タイプ: 合成 / 詳細: THIS PEPTIDE WAS CHEMICALLY SYNTHESIZED. / 参照: UniProt: Q13291 #3: 水 | ChemComp-HOH / | Has protein modification | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.6 Å3/Da / 溶媒含有率: 52.77 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 295 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 5.6 詳細: 30% PEG 8000, 20% GLYCEROL, 100 MM SODIUM CITRATE, 10MM DTT, pH 5.6, VAPOR DIFFUSION, HANGING DROP, temperature 22K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS pH: 7.5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RU300 / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RU300 / 波長: 1.5418 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.8→20 Å / Num. all: 23946 / % possible obs: 94.8 % / Rmerge(I) obs: 0.076 |
| 反射 | *PLUS Num. obs: 23946 / Num. measured all: 113987 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.8→10 Å / σ(F): 4 / 立体化学のターゲット値: ENGH& HUBER
| |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.8→10 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL-97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 1.8 Å / 最低解像度: 10 Å / σ(F): 4 / Rfactor Rfree: 0.24 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj

