+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1alx | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
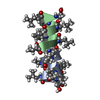
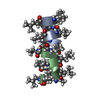
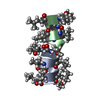
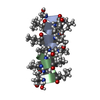
| タイトル | GRAMICIDIN D FROM BACILLUS BREVIS (METHANOL SOLVATE) | |||||||||
 要素 要素 | (GRAMICIDIN A) x 2 | |||||||||
 キーワード キーワード | ANTIBIOTIC / GRAMICIDIN / ANTIFUNGAL / ANTIBACTERIAL / ABTIBIOTIC / MEMBRANE ION CHANNEL / LINEAR GRAMICIDIN | |||||||||
| 機能・相同性 | GRAMICIDIN D / METHANOL / :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  BREVIBACILLUS BREVIS (バクテリア) BREVIBACILLUS BREVIS (バクテリア) | |||||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.2 Å 分子置換 / 解像度: 1.2 Å | |||||||||
 データ登録者 データ登録者 | Burkhart, B.M. / Langs, D.A. / Smith, G.D. / Courseille, C. / Precigoux, G. / Hospital, M. / Pangborn, W.A. / Duax, W.L. | |||||||||
 引用 引用 |  ジャーナル: Biophys.J. / 年: 1998 ジャーナル: Biophys.J. / 年: 1998タイトル: Heterodimer Formation and Crystal Nucleation of Gramicidin D 著者: Burkhart, B.M. / Gassman, R.M. / Langs, D.A. / Pangborn, W.A. / Duax, W.L. #1: ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 1991 タイトル: Monoclinic Uncomplexed Double-Stranded, Antiparallel, Left-Handed Beta 5.6-Helix (Increases Decreases Beta 5.6) Structure of Gramicidin A: Alternate Patterns of Helical Association and Deformation 著者: Langs, D.A. / Smith, G.D. / Courseille, C. / Precigoux, G. / Hospital, M. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1alx.cif.gz 1alx.cif.gz | 46.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1alx.ent.gz pdb1alx.ent.gz | 37.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1alx.json.gz 1alx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1alx_validation.pdf.gz 1alx_validation.pdf.gz | 450.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1alx_full_validation.pdf.gz 1alx_full_validation.pdf.gz | 453.2 KB | 表示 | |
| XML形式データ |  1alx_validation.xml.gz 1alx_validation.xml.gz | 5.1 KB | 表示 | |
| CIF形式データ |  1alx_validation.cif.gz 1alx_validation.cif.gz | 6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/al/1alx https://data.pdbj.org/pub/pdb/validation_reports/al/1alx ftp://data.pdbj.org/pub/pdb/validation_reports/al/1alx ftp://data.pdbj.org/pub/pdb/validation_reports/al/1alx | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 非結晶学的対称性 (NCS) | NCS oper: (Code: given Matrix: (0.96861, 0.1753, -0.17624), ベクター: |
- 要素
要素
| #1: タンパク質・ペプチド |   タイプ: Polypeptide / クラス: 抗生剤 / 分子量: 1859.258 Da / 分子数: 1 / 由来タイプ: 天然 タイプ: Polypeptide / クラス: 抗生剤 / 分子量: 1859.258 Da / 分子数: 1 / 由来タイプ: 天然詳細: GRAMICIDIN D IS A HEXADECAMERIC HELICAL PEPTIDE WITH ALTERNATING D,L CHARACTERISTICS. THE N-TERM IS FORMYLATED (RESIDUE 1). THE C-TERM IS CAPPED WITH ETHANOLAMINE (RESIDUE 16). 由来: (天然)  BREVIBACILLUS BREVIS (バクテリア) / 参照: NOR: NOR00243, GRAMICIDIN D BREVIBACILLUS BREVIS (バクテリア) / 参照: NOR: NOR00243, GRAMICIDIN D | ||||
|---|---|---|---|---|---|
| #2: タンパク質・ペプチド |   タイプ: Polypeptide / クラス: 抗生剤 / 分子量: 1882.294 Da / 分子数: 1 / 由来タイプ: 天然 タイプ: Polypeptide / クラス: 抗生剤 / 分子量: 1882.294 Da / 分子数: 1 / 由来タイプ: 天然詳細: GRAMICIDIN D IS A HEXADECAMERIC HELICAL PEPTIDE WITH ALTERNATING D,L CHARACTERISTICS. THE N-TERM IS FORMYLATED (RESIDUE 1). THE C-TERM IS CAPPED WITH ETHANOLAMINE (RESIDUE 16). 由来: (天然)  BREVIBACILLUS BREVIS (バクテリア) / 参照: NOR: NOR00243, GRAMICIDIN D BREVIBACILLUS BREVIS (バクテリア) / 参照: NOR: NOR00243, GRAMICIDIN D | ||||
| #3: 化合物 | ChemComp-MOH / 構成要素の詳細 | GRAMICIDIN IS A HETEROGENEOUS MIXTURE OF SEVERAL COMPOUNDS INCLUDING GRAMICIDIN A, B AND C WHICH ...GRAMICIDIN | Has protein modification | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.65 Å3/Da / 溶媒含有率: 25.57 % | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7 詳細: CRYSTALLIZED BY BATCH METHODS FROM A SATURATED SOLUTION OF GRAMICIDIN D IN METHANOL., PH 7.0, BATCH METHOD | ||||||||||||||||||||
| 結晶化 | *PLUS 手法: unknown | ||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 120 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 回転陽極 / タイプ: RIGAKU RUH2R / 波長: 1.5418 |
| 検出器 | タイプ: ENRAF-NONIUS FAST / 検出器: DIFFRACTOMETER / 日付: 1990年1月1日 / 詳細: COLLIMATOR |
| 放射 | モノクロメーター: GRAPHITE(002) / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.25→100 Å / Num. obs: 7726 / % possible obs: 99.9 % / Observed criterion σ(I): -3 / 冗長度: 1 % / Net I/σ(I): 85.41 |
| 反射 シェル | 解像度: 1.2→1.24 Å / 冗長度: 1 % / Mean I/σ(I) obs: 35.53 / % possible all: 99 |
| 反射 | *PLUS Rmerge(I) obs: 0.0891 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1GMA  1gma 解像度: 1.2→100 Å / Num. parameters: 3422 / Num. restraintsaints: 5704 / 交差検証法: FREE R-VALUE / σ(F): 0 StereochEM target val spec case: MODIFIED ENGH AND HUBER FOR ETHANOLAMINE BASED ON SERINE 立体化学のターゲット値: ENGH AND HUBER
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER (G = 0.12792 U = 0.82322) | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 5 / Occupancy sum hydrogen: 367.54 / Occupancy sum non hydrogen: 315.41 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.2→100 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL-97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS Rfactor Rwork: 0.1 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー




















 PDBj
PDBj