+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-9317 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Dihedral oligomeric complex of GyrA N-terminal fragment, solved by cryoEM in D2 symmetry | ||||||||||||||||||
 マップデータ マップデータ | Dihedral oligomeric complex of GyrA N-terminal fragment, solved by cryoEM in D2 symmetry | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
 キーワード キーワード | topoisomerase / oligomeric complex / gyrase / ISOMERASE | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) complex / DNA negative supercoiling activity / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / DNA-templated DNA replication / chromosome / DNA binding / ATP binding / cytoplasm 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  Streptococcus pneumoniae G54 (肺炎レンサ球菌) Streptococcus pneumoniae G54 (肺炎レンサ球菌) | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 5.16 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Soczek KM / Grant T | ||||||||||||||||||
| 資金援助 |  米国, 米国,  英国, 5件 英国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2018 ジャーナル: Elife / 年: 2018タイトル: CryoEM structures of open dimers of gyrase A in complex with DNA illuminate mechanism of strand passage. 著者: Katarzyna M Soczek / Tim Grant / Peter B Rosenthal / Alfonso Mondragón /   要旨: Gyrase is a unique type IIA topoisomerase that uses ATP hydrolysis to maintain the negatively supercoiled state of bacterial DNA. In order to perform its function, gyrase undergoes a sequence of ...Gyrase is a unique type IIA topoisomerase that uses ATP hydrolysis to maintain the negatively supercoiled state of bacterial DNA. In order to perform its function, gyrase undergoes a sequence of conformational changes that consist of concerted gate openings, DNA cleavage, and DNA strand passage events. Structures where the transported DNA molecule (T-segment) is trapped by the A subunit have not been observed. Here we present the cryoEM structures of two oligomeric complexes of open gyrase A dimers and DNA. The protein subunits in these complexes were solved to 4 Å and 5.2 Å resolution. One of the complexes traps a linear DNA molecule, a putative T-segment, which interacts with the open gyrase A dimers in two states, representing steps either prior to or after passage through the DNA-gate. The structures locate the T-segment in important intermediate conformations of the catalytic cycle and provide insights into gyrase-DNA interactions and mechanism. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_9317.map.gz emd_9317.map.gz | 28.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-9317-v30.xml emd-9317-v30.xml emd-9317.xml emd-9317.xml | 16.6 KB 16.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_9317_fsc.xml emd_9317_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_9317.png emd_9317.png | 45.3 KB | ||
| Filedesc metadata |  emd-9317.cif.gz emd-9317.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-9317 http://ftp.pdbj.org/pub/emdb/structures/EMD-9317 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9317 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-9317 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_9317_validation.pdf.gz emd_9317_validation.pdf.gz | 562.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_9317_full_validation.pdf.gz emd_9317_full_validation.pdf.gz | 562.1 KB | 表示 | |
| XML形式データ |  emd_9317_validation.xml.gz emd_9317_validation.xml.gz | 9.4 KB | 表示 | |
| CIF形式データ |  emd_9317_validation.cif.gz emd_9317_validation.cif.gz | 12 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9317 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9317 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9317 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-9317 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_9317.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_9317.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
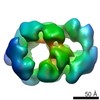
| 注釈 | Dihedral oligomeric complex of GyrA N-terminal fragment, solved by cryoEM in D2 symmetry | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Complex of DNA Gyrase A subunit in D2 symmetry
| 全体 | 名称: Complex of DNA Gyrase A subunit in D2 symmetry |
|---|---|
| 要素 |
|
-超分子 #1: Complex of DNA Gyrase A subunit in D2 symmetry
| 超分子 | 名称: Complex of DNA Gyrase A subunit in D2 symmetry / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pneumoniae G54 (肺炎レンサ球菌) Streptococcus pneumoniae G54 (肺炎レンサ球菌) |
| 分子量 | 理論値: 460 KDa |
-分子 #1: DNA gyrase subunit A
| 分子 | 名称: DNA gyrase subunit A / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: ec: 5.99.1.3 |
|---|---|
| 由来(天然) | 生物種:  Streptococcus pneumoniae G54 (肺炎レンサ球菌) Streptococcus pneumoniae G54 (肺炎レンサ球菌)株: G |
| 分子量 | 理論値: 57.977578 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHSSG VDLGTENLYF QSIAMQDKNL VNVNLTKEMK ASFIDYAMSV IVARALPDVR DGLKPVHRRI LYGMNELGVT PDKPHKKSA RITGDVMGKY HPHGDSSIYE AMVRMAQWWS YRYMLVDGHG NFGSMDGDSA AAQRYTEARM SKIALEMLRD I NKNTVDFV ...文字列: MHHHHHHSSG VDLGTENLYF QSIAMQDKNL VNVNLTKEMK ASFIDYAMSV IVARALPDVR DGLKPVHRRI LYGMNELGVT PDKPHKKSA RITGDVMGKY HPHGDSSIYE AMVRMAQWWS YRYMLVDGHG NFGSMDGDSA AAQRYTEARM SKIALEMLRD I NKNTVDFV DNYDANEREP LVLPARFPNL LVNGATGIAV GMATNIPPHN LGETIDAVKL VMDNPEVTTK DLMEVLPGPD FP TGALVMG KSGIHKAYET GKGSIVLRSR TEIETTKTGR ERIVVTEFPY MVNKTKVHEH IVRLVQEKRI EGITAVRDES NRE GVRFVI EVKRDASANV ILNNLFKMTQ MQTNFGFNML AIQNGIPKIL SLRQILDAYI EHQKEVVVRR TRFDKEKAEA RAHI LEGLL IALDHIDEVI RIIRASETDA EAQAELMSKF KLSERQSQAI LDMRLRRLTG LERDKIQSEY DDLLALIADL ADILA KPER VSQIIKDELD EVKRKFSDKR RTELMVG UniProtKB: DNA gyrase subunit A |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-1.2/1.3 4C / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 3200FS |
|---|---|
| 温度 | 最低: 100.0 K / 最高: 100.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: In-column Omega Filter エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3838 pixel / デジタル化 - 画像ごとのフレーム数: 1-40 / 実像数: 718 / 平均露光時間: 12.0 sec. / 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 最小 デフォーカス(補正後): 1.0 µm / 倍率(補正後): 40323 / 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 倍率(公称値): 30000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 626 SINGLE TILT LIQUID NITROGEN CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 2-486 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 当てはまり具合の基準: correlation coefficient |
| 得られたモデル |  PDB-6n1q: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)