+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8510 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative-stain electron microscopy reconstructions of a dimodular nonribosomal peptide synthetase | |||||||||
 マップデータ マップデータ | dimodular nonribosomal peptide synthetase | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Anoxybacillus kamchatkensis G10 (バクテリア) Anoxybacillus kamchatkensis G10 (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 25.8 Å | |||||||||
 データ登録者 データ登録者 | Haque AS / Bui KH / Schmeing TM | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2017 ジャーナル: Structure / 年: 2017タイトル: X-Ray Crystallography and Electron Microscopy of Cross- and Multi-Module Nonribosomal Peptide Synthetase Proteins Reveal a Flexible Architecture. 著者: Michael J Tarry / Asfarul S Haque / Khanh Huy Bui / T Martin Schmeing /  要旨: Nonribosomal peptide synthetases (NRPS) are macromolecular machines that produce peptides with diverse activities. Structural information exists for domains, didomains, and even modules, but little ...Nonribosomal peptide synthetases (NRPS) are macromolecular machines that produce peptides with diverse activities. Structural information exists for domains, didomains, and even modules, but little is known about higher-order organization. We performed a multi-technique study on constructs from the dimodular NRPS DhbF. We determined a crystal structure of a cross-module construct including the adenylation (A) and peptidyl carrier protein (PCP) domains from module 1 and the condensation domain from module 2, complexed with an adenosine-vinylsulfonamide inhibitor and an MbtH-like protein (MLP). The action of the inhibitor and the role of the MLP were investigated using adenylation reactions and isothermal titration calorimetry. In the structure, the PCP and A domains adopt a novel conformation, and noncovalent, cross-module interactions are limited. We calculated envelopes of dimodular DhbF using negative-stain electron microscopy. The data show large conformational variability between modules. Together, our results suggest that NRPSs lack a uniform, rigid supermodular architecture. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8510.map.gz emd_8510.map.gz | 9.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8510-v30.xml emd-8510-v30.xml emd-8510.xml emd-8510.xml | 22.3 KB 22.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_8510_fsc_1.xml emd_8510_fsc_1.xml emd_8510_fsc_2.xml emd_8510_fsc_2.xml emd_8510_fsc_3.xml emd_8510_fsc_3.xml emd_8510_fsc_4.xml emd_8510_fsc_4.xml emd_8510_fsc_5.xml emd_8510_fsc_5.xml | 6.4 KB 6.4 KB 6.4 KB 6.4 KB 6.4 KB | 表示 表示 表示 表示 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_8510_1.png emd_8510_1.png emd_8510_2.png emd_8510_2.png emd_8510_3.png emd_8510_3.png emd_8510_4.png emd_8510_4.png emd_8510_5.png emd_8510_5.png | 36.5 KB 42.2 KB 40.5 KB 62.8 KB 41.8 KB | ||
| その他 |  emd_8510_additional_1.map.gz emd_8510_additional_1.map.gz emd_8510_additional_2.map.gz emd_8510_additional_2.map.gz emd_8510_additional_3.map.gz emd_8510_additional_3.map.gz emd_8510_additional_4.map.gz emd_8510_additional_4.map.gz | 9.7 MB 9.7 MB 9.7 MB 9.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8510 http://ftp.pdbj.org/pub/emdb/structures/EMD-8510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8510 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_8510_validation.pdf.gz emd_8510_validation.pdf.gz | 78.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_8510_full_validation.pdf.gz emd_8510_full_validation.pdf.gz | 77.6 KB | 表示 | |
| XML形式データ |  emd_8510_validation.xml.gz emd_8510_validation.xml.gz | 494 B | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8510 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8510 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8510 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-8510 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8510.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8510.map.gz / 形式: CCP4 / 大きさ: 12.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dimodular nonribosomal peptide synthetase | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.21 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: dimodular nonribosomal peptide synthetase, additional map #1
| ファイル | emd_8510_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dimodular nonribosomal peptide synthetase, additional map #1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
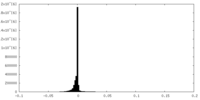
| 密度ヒストグラム |
-追加マップ: dimodular nonribosomal peptide synthetase, additional map #2
| ファイル | emd_8510_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dimodular nonribosomal peptide synthetase, additional map #2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
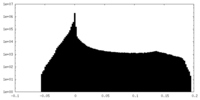
| 密度ヒストグラム |
-追加マップ: dimodular nonribosomal peptide synthetase, additional map #3
| ファイル | emd_8510_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dimodular nonribosomal peptide synthetase, additional map #3 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
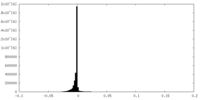
| 密度ヒストグラム |
-追加マップ: dimodular nonribosomal peptide synthetase, additional map #4
| ファイル | emd_8510_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dimodular nonribosomal peptide synthetase, additional map #4 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
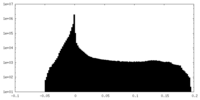
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dimodule of DhbF, a bacillibactin-synthesizing nonribosomal pepti...
| 全体 | 名称: Dimodule of DhbF, a bacillibactin-synthesizing nonribosomal peptide synthetase |
|---|---|
| 要素 |
|
-超分子 #1: Dimodule of DhbF, a bacillibactin-synthesizing nonribosomal pepti...
| 超分子 | 名称: Dimodule of DhbF, a bacillibactin-synthesizing nonribosomal peptide synthetase タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Anoxybacillus kamchatkensis G10 (バクテリア) Anoxybacillus kamchatkensis G10 (バクテリア) |
| 組換発現 | 生物種:  |
| 分子量 | 理論値: 238 KDa |
-分子 #1: DhbF
| 分子 | 名称: DhbF / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Anoxybacillus kamchatkensis G10 (バクテリア) Anoxybacillus kamchatkensis G10 (バクテリア) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ASMNQRLRLP LSGAQAGIWF AQQLDPANPI YNTGEYVEIH GPVDPARFEQ ALRHVLVQAE SLHAQFGED ENGPWQIIDP SPDFPFYFID VSTAANPESE ALFWMKKDLS KPVDLKCDPL F TEALFKLS DQRFFWYQRI HHIAIDGFGF SLIAQKVAET YTALMNDRMI ...文字列: ASMNQRLRLP LSGAQAGIWF AQQLDPANPI YNTGEYVEIH GPVDPARFEQ ALRHVLVQAE SLHAQFGED ENGPWQIIDP SPDFPFYFID VSTAANPESE ALFWMKKDLS KPVDLKCDPL F TEALFKLS DQRFFWYQRI HHIAIDGFGF SLIAQKVAET YTALMNDRMI AADEAFASFR EV IEEEKVY HASEQYERDR QFWLERFRDQ PEAVSLSDRA ARASHTFIRK TVHLSVAQTE RLK QSTQYW KAGWHELFLA ATALYLHRLT SATDVILGLP MMNRLGSVAL NVPAMVMNLV PFRL HLHAD MKMSQLLSAV RQEIKEIKQH HKYRHEQLRR DLKLLGENQR LFGVQVNIMP FDYGL HFDG YQGITHNLSA GPVDDLAINV YDRTDGNGLR IDFDANPEIY QADDLAIHQS RFLQIL EML TVLQEDVTIG SVELLLEQER RQVLEIWNET ARHHSEASFL QLFEKQAQQH PEAMAVI CE NQMLRYQELN EQANRLAHLL VEKGAGPEQY VALALPRSAD MVIAMLAVLK AGAAYLPI D PEYPKERLAF MLDDAEPLCI ITNSATQPKL SAISPLSMIV LDHPETEEAI KRYPSTNVE NGRSALHPAY VIYTSGSTGK PKGVVVPFQN LNNFLFAMQD KFMLNERDRW LAVTTIAFDI AALEIFLPL MSGARLIVAK KEAIHDPQTL AALISNEEIT IMQATPTLWH MLVTYHPDSI A GLRVLVGG EALPSSLASA LDQLGCEITN LYGPTETTIW STMATLQQDD IVAPVIGKPI WN TQVYVLD RHLQPVPPGV AGELYIAGEG VARGYLKRPD LTAERFVANP YGPPGSRMYR TGD LVRWRK DGSLDYIGRV DYQVKLRGFR IEIGEIEAIL TKYDEVERAV VVAREDQPGA PRLV AYLIP RGSKGALDLA ELRLYVSEKL PDYMVPSAFM ILEEFPLTPN GKIDRKALPV PDWTV TMKG RKPRTPQEEI LCELFAEVLD LSAVGIDDNF FELGGHSLLA ARLISRIRDV LGVELA IGK LFESPTVASL VHHLKEAAHG KPPVKAYAHK DEIPLSFAQR RLWFLHHLEG PSPTYNI PV VVHLTGALQI AALRQALYDV VERHETLRTI FPDRSGTSRQ IVLEPHQARP ELIVQEIS E NELSRVLNEA VRYSFDLAKE PPIRAQLFVL GENRYVLLLL MHHIAADGWS LTPLTRDLA SAYQAHCQQQ KVDWPPLPVT YADYALWQQE FLGNENHPDH LIAKQLDYWK QTLADLPEEL EWSTDYPRP AESSYEGGVV EFKLDAELHQ RLLALARENK TSLFMVLQAG FAALLTRLGA G TDIPIGSP IAGRNDDALE DLVGMFINTL VLRMDTSGNP SFRELLARVK QVNLSAYENQ DL PFERLVE VLNPVRSRAK HPLFQVMFVF QNTPEPKLEL QGLESRLEVR SVGSAKFDLT LEL RENRMG DGSPAGLIGL FEYSRDLFQQ ETVERFAKRL CQLLKEVVTN PELPIGQIHM LLPE ERKLL LNQAEHRQRH LSAETLPALF EKQVQRVPDA TAVVFEDQKL SYAELNKKAN QLAHF LISK GVGPERVVAL ALPRSLEMVI GILAVLKAGG AYLPLDPNYP EDRIAYMMED AQPMYV IAN QQMAVKLPHT PNVEHIVVDA EHVVGQIGHY PETNPNDADR IEALSPYHMA YIIYTSG ST GKPKGVMIPH QNVVRLFRST EHWFQFSEND SWTLFHSYAF DFSVWEIWGP FLYGGRLV V VPHHVSRSPK EFLQLLVEQN VTVLNQTPSA FYQLMQADRE NSEWSKQLSL RFVIFGGEA LELSRLEDWY ERHPENQPKL INMYGITETT VHVSYIELDR TILSLKGNSL IGCSIPDLEV YVLDDQLQP VPPGVIGEMY VAGAGLARGY LGRPDLTAER FIANPFGPPG SRMYRTGDLA K WRVDGTLD YIGRADHQVK IRGYRIELGE IEAVLAKHPQ IAQVAVIVRE DQPGDKRLVA YI VPAKDAV FESSELRKYV AASLPDYMIP SAFVMIEALP LTPNGKLDRK ALPMPDLAIE VNG RGPRTP QEEMLCDLFM EILRLPRVGI DDGFFELGGH SLLAVQLMSR IRETFGVELS IGDL FEAPT VAGLAEKLEM GSS |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.01 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl formate | ||||||||||||
| グリッド | 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 7.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 雰囲気: AIR |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 12.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)