+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-7772 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Seneca Valley Virus-Anthrax Toxin Receptor 1 complex | |||||||||
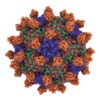
 マップデータ マップデータ | Seneca Valley Virus-Anthrax Toxin Receptor 1 complex | |||||||||
 試料 試料 | Seneca Valley Virus-Anthrax Toxin Receptor 1 complex != Senecavirus A Seneca Valley Virus-Anthrax Toxin Receptor 1 complex
| |||||||||
 キーワード キーワード | Virus-receptor complex / Picornavirus / Senecavirus / Anthrax Toxin Receptor / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF7 activity / host cell nucleolus / adhesion receptor-mediated virion attachment to host cell / filopodium membrane / negative regulation of extracellular matrix assembly / symbiont-mediated suppression of host TRAF-mediated signal transduction / blood vessel development / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / Uptake and function of anthrax toxins / lamellipodium membrane ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF7 activity / host cell nucleolus / adhesion receptor-mediated virion attachment to host cell / filopodium membrane / negative regulation of extracellular matrix assembly / symbiont-mediated suppression of host TRAF-mediated signal transduction / blood vessel development / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / Uptake and function of anthrax toxins / lamellipodium membrane / collagen binding / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of MAVS activity / substrate adhesion-dependent cell spreading / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / actin filament binding / transmembrane signaling receptor activity / channel activity / actin cytoskeleton organization / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of TBK1 activity / monoatomic ion transmembrane transport / symbiont-mediated suppression of host toll-like receptor signaling pathway / symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of IRF3 activity / ubiquitinyl hydrolase 1 / entry receptor-mediated virion attachment to host cell / cysteine-type deubiquitinase activity / RNA helicase activity / endosome membrane / RNA helicase / external side of plasma membrane / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / symbiont entry into host cell / virion attachment to host cell / host cell nucleus / structural molecule activity / cell surface / ATP hydrolysis activity / proteolysis / RNA binding / ATP binding / metal ion binding / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Senecavirus A (ウイルス) Senecavirus A (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Jayawardena N / Burga L | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Structural basis for anthrax toxin receptor 1 recognition by Seneca Valley Virus. 著者: Nadishka Jayawardena / Laura N Burga / Richard A Easingwood / Yoshimasa Takizawa / Matthias Wolf / Mihnea Bostina /   要旨: Recently, the use of oncolytic viruses in cancer therapy has become a realistic therapeutic option. Seneca Valley Virus (SVV) is a newly discovered picornavirus, which has earned a significant ...Recently, the use of oncolytic viruses in cancer therapy has become a realistic therapeutic option. Seneca Valley Virus (SVV) is a newly discovered picornavirus, which has earned a significant reputation as a potent oncolytic agent. Anthrax toxin receptor 1 (ANTXR1), one of the cellular receptors for the protective antigen secreted by , has been identified as the high-affinity cellular receptor for SVV. Here, we report the structure of the SVV-ANTXR1 complex determined by single-particle cryo-electron microscopy analysis at near-atomic resolution. This is an example of a shared receptor structure between a mammalian virus and a bacterial toxin. Our structure shows that ANTXR1 decorates the outer surface of the SVV capsid and interacts with the surface-exposed BC loop and loop II of VP1, "the puff" of VP2 and "the knob" of VP3. Comparison of the receptor-bound capsid structure with the native capsid structure reveals that receptor binding induces minor conformational changes in SVV capsid structure, suggesting the role of ANTXR1 as an attachment receptor. Furthermore, our results demonstrate that the capsid footprint on the receptor is not conserved in anthrax toxin receptor 2 (ANTXR2), thereby providing a molecular mechanism for explaining the exquisite selectivity of SVV for ANTXR1. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_7772.map.gz emd_7772.map.gz | 64.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-7772-v30.xml emd-7772-v30.xml emd-7772.xml emd-7772.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_7772.png emd_7772.png | 244.1 KB | ||
| Filedesc metadata |  emd-7772.cif.gz emd-7772.cif.gz | 6.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-7772 http://ftp.pdbj.org/pub/emdb/structures/EMD-7772 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7772 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-7772 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_7772_validation.pdf.gz emd_7772_validation.pdf.gz | 619.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_7772_full_validation.pdf.gz emd_7772_full_validation.pdf.gz | 618.9 KB | 表示 | |
| XML形式データ |  emd_7772_validation.xml.gz emd_7772_validation.xml.gz | 7.5 KB | 表示 | |
| CIF形式データ |  emd_7772_validation.cif.gz emd_7772_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7772 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7772 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7772 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-7772 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_7772.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_7772.map.gz / 形式: CCP4 / 大きさ: 282.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Seneca Valley Virus-Anthrax Toxin Receptor 1 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.42 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Seneca Valley Virus-Anthrax Toxin Receptor 1 complex
| 全体 | 名称: Seneca Valley Virus-Anthrax Toxin Receptor 1 complex |
|---|---|
| 要素 |
|
-超分子 #1: Senecavirus A
| 超分子 | 名称: Senecavirus A / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 390157 / 生物種: Senecavirus A / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 分子量 | 理論値: 6.5 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Capsid / 直径: 300.0 Å / T番号(三角分割数): 3 |
-分子 #1: Anthrax toxin receptor 1
| 分子 | 名称: Anthrax toxin receptor 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 21.026668 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: CYGGFDLYFI LDKSGSVLHH WNEIYYFVEQ LAHKFISPQL RMSFIVFSTR GTTLMKLTED REQIRQGLEE LQKVLPGGDT YMHEGFERA SEQIYYENRQ GYRTASVIIA LTDGELHEDL FFYSEREANR SRDLGAIVYA VGVKDFNETQ LARIADSKDH V FPVNDGFQ ALQGIIHSIL KKSC UniProtKB: Anthrax toxin receptor 1 |
-分子 #2: Capsid protein VP1
| 分子 | 名称: Capsid protein VP1 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Senecavirus A (ウイルス) Senecavirus A (ウイルス) |
| 分子量 | 理論値: 28.459969 KDa |
| 配列 | 文字列: STDNAETGVI EAGNTDTDFS GELAAPGSNH TNVKFLFDRS RLLNVIKVLE KDAVFPRPFP TQEGAQQDDG YFCLLTPRPT VASRPATRF GLYANPSGSG VLANTSLDFN FYSLACFTYF RSDLEVTVVS LEPDLEFAVG WFPSGSEYQA SSFVYDQLHV P FHFTGRTP ...文字列: STDNAETGVI EAGNTDTDFS GELAAPGSNH TNVKFLFDRS RLLNVIKVLE KDAVFPRPFP TQEGAQQDDG YFCLLTPRPT VASRPATRF GLYANPSGSG VLANTSLDFN FYSLACFTYF RSDLEVTVVS LEPDLEFAVG WFPSGSEYQA SSFVYDQLHV P FHFTGRTP RAFASKGGKV SFVLPWNSVS SVLPVRWGGA SKLSSATRGL PAHADWGTIY AFVPRPNEKK STAVKHVAVY IR YKNARAW CPSMLPFRSY K UniProtKB: Genome polyprotein |
-分子 #3: Capsid protein VP2
| 分子 | 名称: Capsid protein VP2 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Senecavirus A (ウイルス) Senecavirus A (ウイルス) |
| 分子量 | 理論値: 29.870316 KDa |
| 配列 | 文字列: DRVTTQTAGN TAINTQSSLG VLCAYVEDPT KSDPPSSSTD QPTTTFTAID RWYTGRLNSW TKAVKTFSFQ AVPLPGAFLS RQGGLNGGA FTATLHRHFL MKCGWQVQVQ CNLTQFHQGA LLVAMVPETT LDVKPDGKAK SLQELNEEQW VEMSDDYRTG K NMPFQSLG ...文字列: DRVTTQTAGN TAINTQSSLG VLCAYVEDPT KSDPPSSSTD QPTTTFTAID RWYTGRLNSW TKAVKTFSFQ AVPLPGAFLS RQGGLNGGA FTATLHRHFL MKCGWQVQVQ CNLTQFHQGA LLVAMVPETT LDVKPDGKAK SLQELNEEQW VEMSDDYRTG K NMPFQSLG TYYRPPNWTW GPNFINPYQV TVFPHQILNA RTSTSVDINV PYIGETPTQS SETQNSWTLL VMVLVPLDYK EG ATTDPEI TFSVRPTSPY FNGLRNRYTA G UniProtKB: Genome polyprotein |
-分子 #4: Capsid protein VP3
| 分子 | 名称: Capsid protein VP3 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Senecavirus A (ウイルス) Senecavirus A (ウイルス) |
| 分子量 | 理論値: 26.353938 KDa |
| 配列 | 文字列: GPIPTAPREN SLMFLSTLPD DTVPAYGNVR TPPVNYLPGE ITDLLQLARI PTLMAFERVP EPVPASDTYV PYVAVPTQFD DRPLISFPI TLSDPVYQNT LVGAISSNFA NYRGCIQITL TFCGPMMARG KFLLSYSPPN GTQPQTLSEA MQCTYSIWDI G LNSSWTFV ...文字列: GPIPTAPREN SLMFLSTLPD DTVPAYGNVR TPPVNYLPGE ITDLLQLARI PTLMAFERVP EPVPASDTYV PYVAVPTQFD DRPLISFPI TLSDPVYQNT LVGAISSNFA NYRGCIQITL TFCGPMMARG KFLLSYSPPN GTQPQTLSEA MQCTYSIWDI G LNSSWTFV VPYISPSDYR ETRAITNSVY SADGWFSLHK LTKITLPPDC PQSPCILFFA SAGEDYTLRL PVDCNPSYVF UniProtKB: Genome polyprotein |
-分子 #5: Capsid protein VP4
| 分子 | 名称: Capsid protein VP4 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Senecavirus A (ウイルス) Senecavirus A (ウイルス) |
| 分子量 | 理論値: 5.998467 KDa |
| 配列 | 文字列: RGNNGNMTFN YYANTYQNSV DFSTSSSASG AGPGNSRGGL AGLLTNFSGI LNPLGYLK UniProtKB: Genome polyprotein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 構成要素 - 名称: Phosphate Buffer |
| グリッド | モデル: Quantifoil R1.2/1.3 / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 12 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 293 K / 装置: FEI VITROBOT MARK IV / 詳細: Blot force 0, 3 sec blotting time. |
| 詳細 | A homogenous sample containing full capsids |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 39.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 73000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 100 / 当てはまり具合の基準: CC |
|---|---|
| 得られたモデル |  PDB-6cx1: |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















