+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4945 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | In situ structure of a pentameric IgM complex bound to complement components C1 and two molecules of C4b | |||||||||
 マップデータ マップデータ | Pentameric IgM in complex with complement component C1 and two molecules of C4b | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
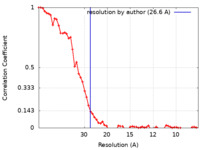
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 26.6 Å | |||||||||
 データ登録者 データ登録者 | Sharp TH | |||||||||
| 資金援助 |  オランダ, 1件 オランダ, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2019 ジャーナル: Proc Natl Acad Sci U S A / 年: 2019タイトル: Insights into IgM-mediated complement activation based on in situ structures of IgM-C1-C4b. 著者: Thomas H Sharp / Aimee L Boyle / Christoph A Diebolder / Alexander Kros / Abraham J Koster / Piet Gros /  要旨: Antigen binding by serum Ig-M (IgM) protects against microbial infections and helps to prevent autoimmunity, but causes life-threatening diseases when mistargeted. How antigen-bound IgM activates ...Antigen binding by serum Ig-M (IgM) protects against microbial infections and helps to prevent autoimmunity, but causes life-threatening diseases when mistargeted. How antigen-bound IgM activates complement-immune responses remains unclear. We present cryoelectron tomography structures of IgM, C1, and C4b complexes formed on antigen-bearing lipid membranes by normal human serum at 4 °C. The IgM-C1-C4b complexes revealed C4b product release as the temperature-limiting step in complement activation. Both IgM hexamers and pentamers adopted hexagonal, dome-shaped structures with Fab pairs, dimerized by hinge domains, bound to surface antigens that support a platform of Fc regions. C1 binds IgM through widely spread C1q-collagen helices, with C1r proteases pointing outward and C1s bending downward and interacting with surface-attached C4b, which further interacts with the adjacent IgM-Fab and globular C1q-recognition unit. Based on these data, we present mechanistic models for antibody-mediated, C1q-transmitted activation of C1 and for C4b deposition, while further conformational rearrangements are required to form C3 convertases. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4945.map.gz emd_4945.map.gz | 14.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4945-v30.xml emd-4945-v30.xml emd-4945.xml emd-4945.xml | 12.7 KB 12.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_4945_fsc.xml emd_4945_fsc.xml | 6.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_4945.png emd_4945.png | 163.7 KB | ||
| マスクデータ |  emd_4945_msk_1.map emd_4945_msk_1.map | 15.6 MB |  マスクマップ マスクマップ | |
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4945 http://ftp.pdbj.org/pub/emdb/structures/EMD-4945 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4945 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4945 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4945_validation.pdf.gz emd_4945_validation.pdf.gz | 269.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4945_full_validation.pdf.gz emd_4945_full_validation.pdf.gz | 268.7 KB | 表示 | |
| XML形式データ |  emd_4945_validation.xml.gz emd_4945_validation.xml.gz | 8.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4945 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4945 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4945 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4945 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4945.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4945.map.gz / 形式: CCP4 / 大きさ: 15.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Pentameric IgM in complex with complement component C1 and two molecules of C4b | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.3 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_4945_msk_1.map emd_4945_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : pentameric IgM-C1-C4b2
| 全体 | 名称: pentameric IgM-C1-C4b2 |
|---|---|
| 要素 |
|
-超分子 #1: pentameric IgM-C1-C4b2
| 超分子 | 名称: pentameric IgM-C1-C4b2 / タイプ: complex / ID: 1 / 親要素: 0 詳細: Map contains a complex of pentameric IgM, complement component C1 and two molecules of complement component C4b |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 器官: Human serum Homo sapiens (ヒト) / 器官: Human serum |
| 分子量 | 理論値: 2.126 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 構成要素 - 式: PBS / 構成要素 - 名称: PBS |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 277.15 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | 位相板: VOLTA PHASE PLATE / エネルギーフィルター - 名称: GIF Quantum LS / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均露光時間: 2.4 sec. / 平均電子線量: 1.48 e/Å2 詳細: Exposures of 2.4 sec were dose-fractionated into 6 movie frames per tilt angle. The total dose for each tilt series was 80 e-/A^2. Focusing to -300 nm was performed before each image ...詳細: Exposures of 2.4 sec were dose-fractionated into 6 movie frames per tilt angle. The total dose for each tilt series was 80 e-/A^2. Focusing to -300 nm was performed before each image acquisition using a low-dose routine |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 33000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)