+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-4712 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of LSD2/NPAC-linker/nucleosome core particle complex: Class 5 | |||||||||
 マップデータ マップデータ | Unfolded nucleosome | |||||||||
 試料 試料 |
| |||||||||
| 生物種 | ||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.95 Å | |||||||||
 データ登録者 データ登録者 | Marabelli C / Pilotto S / Chittori S / Subramaniam S / Mattevi A | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2019 ジャーナル: Cell Rep / 年: 2019タイトル: A Tail-Based Mechanism Drives Nucleosome Demethylation by the LSD2/NPAC Multimeric Complex. 著者: Chiara Marabelli / Biagina Marrocco / Simona Pilotto / Sagar Chittori / Sarah Picaud / Sara Marchese / Giuseppe Ciossani / Federico Forneris / Panagis Filippakopoulos / Guy Schoehn / Daniela ...著者: Chiara Marabelli / Biagina Marrocco / Simona Pilotto / Sagar Chittori / Sarah Picaud / Sara Marchese / Giuseppe Ciossani / Federico Forneris / Panagis Filippakopoulos / Guy Schoehn / Daniela Rhodes / Sriram Subramaniam / Andrea Mattevi /       要旨: LSD1 and LSD2 are homologous histone demethylases with opposite biological outcomes related to chromatin silencing and transcription elongation, respectively. Unlike LSD1, LSD2 nucleosome-demethylase ...LSD1 and LSD2 are homologous histone demethylases with opposite biological outcomes related to chromatin silencing and transcription elongation, respectively. Unlike LSD1, LSD2 nucleosome-demethylase activity relies on a specific linker peptide from the multidomain protein NPAC. We used single-particle cryoelectron microscopy (cryo-EM), in combination with kinetic and mutational analysis, to analyze the mechanisms underlying the function of the human LSD2/NPAC-linker/nucleosome complex. Weak interactions between LSD2 and DNA enable multiple binding modes for the association of the demethylase to the nucleosome. The demethylase thereby captures mono- and dimethyl Lys4 of the H3 tail to afford histone demethylation. Our studies also establish that the dehydrogenase domain of NPAC serves as a catalytically inert oligomerization module. While LSD1/CoREST forms a nucleosome docking platform at silenced gene promoters, LSD2/NPAC is a multifunctional enzyme complex with flexible linkers, tailored for rapid chromatin modification, in conjunction with the advance of the RNA polymerase on actively transcribed genes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_4712.map.gz emd_4712.map.gz | 62.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-4712-v30.xml emd-4712-v30.xml emd-4712.xml emd-4712.xml | 16.8 KB 16.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_4712.png emd_4712.png | 92.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-4712 http://ftp.pdbj.org/pub/emdb/structures/EMD-4712 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4712 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4712 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_4712_validation.pdf.gz emd_4712_validation.pdf.gz | 193.5 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_4712_full_validation.pdf.gz emd_4712_full_validation.pdf.gz | 192.6 KB | 表示 | |
| XML形式データ |  emd_4712_validation.xml.gz emd_4712_validation.xml.gz | 6.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4712 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4712 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4712 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4712 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_4712.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_4712.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unfolded nucleosome | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
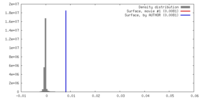
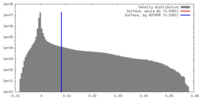
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : partly unfolded nucleosome
| 全体 | 名称: partly unfolded nucleosome |
|---|---|
| 要素 |
|
-超分子 #1: partly unfolded nucleosome
| 超分子 | 名称: partly unfolded nucleosome / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#8 詳細: Xenopus laevis histones recombinantly expressed. Alkylated K4C-C110A H3. 601 Widom DNA sequence. Human LSD2 Human NPAC |
|---|---|
| 由来(天然) | 生物種: |
| 分子量 | 理論値: 178 KDa |
-分子 #1: H3
| 分子 | 名称: H3 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVALFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA |
-分子 #2: H4
| 分子 | 名称: H4 / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG |
-分子 #3: H2A
| 分子 | 名称: H2A / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKQGGKT RAKAKTRSSR AGLQFPVGRV HRLLRKGNYA ERVGAGAPVY LAAVLEYLTA EILELAGNAA RDNKKTRIIP RHLQLAVRN DEELNKLLGR VTIAQGGVLP NIQSVLLPKK TESSKSAKSK |
-分子 #4: H2B
| 分子 | 名称: H2B / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MPDPAKSAPA AKKGSKKAVT KTQKKDGKKR RKSRKESYAI YVYKVLKQVH PDTGISSKAM SIMNSFVND VFERIAGEAS RLAHYNKRST ITSREIQTAV RLLLPGELAK HAVSEGTKAV T KYTSAK |
-分子 #7: H3
| 分子 | 名称: H3 / タイプ: protein_or_peptide / ID: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: ARTKQTARKS TGGKAPRKQL ATKAARKSAP ATGGVKKPHR YRPGTVALRE IRRYQKSTEL LIRKLPFQRL VREIAQDFKT DLRFQSSAV MALQEASEAY LVALFEDTNL CAIHAKRVTI MPKDIQLARR IRGERA |
-分子 #8: H4
| 分子 | 名称: H4 / タイプ: protein_or_peptide / ID: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGRGKGGKGL GKGGAKRHRK VLRDNIQGIT KPAIRRLARR GGVKRISGLI YEETRGVLKV FLENVIRDAV TYTEHAKRKT VTAMDVVYA LKRQGRTLYG FGG |
-分子 #5: DNA (147mer)
| 分子 | 名称: DNA (147mer) / タイプ: dna / ID: 5 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: ATCGGATGTA TATATCTGAC ACGTGCCTGG AGACTAGGGA GTAATCCCCT TGGCGGTTAA AACGCGGGGG ACAGCGCGTA CGTGCGTTT AAGCGGTGCT AGAGCTGTCT ACGACCAATT GAGCGGCCTC GGCACCGGGA TTCTCGAT |
-分子 #6: DNA (147mer)
| 分子 | 名称: DNA (147mer) / タイプ: dna / ID: 6 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: ATCGAGAATC CCGGTGCCGA GGCCGCTCAA TTGGTCGTAG ACAGCTCTAG CACCGCTTAA ACGCACGTAC GCGCTGTCCC CCGCGTTTT AACCGCCAAG GGGATTACTC CCTAGTCTCC AGGCACGTGT CAGATATATA CATCCGAT |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.87 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| 凍結 | 凍結剤: ETHANE | |||||||||
| 詳細 | LSD2/NPAC(214-225)/nucleosome was monodisperse (gel filtration peak isolation and concentration in buffer described. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2078 / 平均露光時間: 8.0 sec. / 平均電子線量: 1.25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 0.00305 µm / 最小 デフォーカス(公称値): 0.0007 µm / 倍率(公称値): 130000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)