登録情報 データベース : EMDB / ID : EMD-4163タイトル CBF3 Core Complex CBF3 core 複合体 : CBF3 Core Complexタンパク質・ペプチド : Centromere DNA-binding protein complex CBF3 subunit Bタンパク質・ペプチド : Suppressor of kinetochore protein 1リガンド : ZINC ION / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
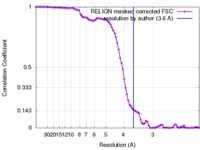
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Saccharomyces cerevisiae (パン酵母)手法 / / 解像度 : 3.6 Å Leber V / Singleton MR 資金援助 Organization Grant number 国 Cancer Research UK FC001155 Medical Research Council (United Kingdom) FC001155 Wellcome Trust FC001155
ジャーナル : EMBO J / 年 : 2018タイトル : Structural basis for assembly of the CBF3 kinetochore complex.著者 : Vera Leber / Andrea Nans / Martin R Singleton / 要旨 : Eukaryotic chromosomes contain a specialised region known as the centromere, which forms the platform for kinetochore assembly and microtubule attachment. The centromere is distinguished by the ... Eukaryotic chromosomes contain a specialised region known as the centromere, which forms the platform for kinetochore assembly and microtubule attachment. The centromere is distinguished by the presence of nucleosomes containing the histone H3 variant, CENP-A. In budding yeast, centromere establishment begins with the recognition of a specific DNA sequence by the CBF3 complex. This in turn facilitates CENP-A nucleosome deposition and kinetochore assembly. Here, we describe a 3.6 Å single-particle cryo-EM reconstruction of the core CBF3 complex, incorporating the sequence-specific DNA-binding protein Cep3 together with regulatory subunits Ctf13 and Skp1. This provides the first structural data on Ctf13, defining it as an F-box protein of the leucine-rich-repeat family, and demonstrates how a novel F-box-mediated interaction between Ctf13 and Skp1 is responsible for initial assembly of the CBF3 complex. 履歴 登録 2017年11月17日 - ヘッダ(付随情報) 公開 2017年12月13日 - マップ公開 2017年12月13日 - 更新 2024年10月9日 - 現状 2024年10月9日 処理サイト : PDBe / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 英国, 3件
英国, 3件  引用
引用 ジャーナル: EMBO J / 年: 2018
ジャーナル: EMBO J / 年: 2018
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_4163.map.gz
emd_4163.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-4163-v30.xml
emd-4163-v30.xml emd-4163.xml
emd-4163.xml EMDBヘッダ
EMDBヘッダ emd_4163_fsc.xml
emd_4163_fsc.xml FSCデータファイル
FSCデータファイル emd_4163.png
emd_4163.png emd-4163.cif.gz
emd-4163.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-4163
http://ftp.pdbj.org/pub/emdb/structures/EMD-4163 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4163
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-4163 emd_4163_validation.pdf.gz
emd_4163_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_4163_full_validation.pdf.gz
emd_4163_full_validation.pdf.gz emd_4163_validation.xml.gz
emd_4163_validation.xml.gz emd_4163_validation.cif.gz
emd_4163_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4163
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4163 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4163
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-4163 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_4163.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_4163.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素




 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 画像解析
画像解析
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)