+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3865 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
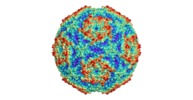
| タイトル | Sacbrood virus of honeybee | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | full particle / icosahedral virus particle / iflavirus / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell membrane / viral capsid / host cell cytoplasm / RNA helicase activity / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / structural molecule activity / proteolysis ...host cell membrane / viral capsid / host cell cytoplasm / RNA helicase activity / viral RNA genome replication / cysteine-type endopeptidase activity / RNA-dependent RNA polymerase activity / DNA-templated transcription / structural molecule activity / proteolysis / RNA binding / ATP binding / membrane / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  Sacbrood virus (ウイルス) Sacbrood virus (ウイルス) | |||||||||
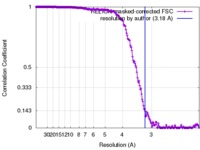
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.18 Å | |||||||||
 データ登録者 データ登録者 | Plevka P / Prochazkova M | |||||||||
| 資金援助 | 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2018 ジャーナル: Proc Natl Acad Sci U S A / 年: 2018タイトル: Virion structure and genome delivery mechanism of sacbrood honeybee virus. 著者: Michaela Procházková / Tibor Füzik / Karel Škubník / Jana Moravcová / Zorica Ubiparip / Antonín Přidal / Pavel Plevka /  要旨: Infection by sacbrood virus (SBV) from the family Iflaviridae is lethal to honey bee larvae but only rarely causes the collapse of honey bee colonies. Despite the negative effect of SBV on honey ...Infection by sacbrood virus (SBV) from the family Iflaviridae is lethal to honey bee larvae but only rarely causes the collapse of honey bee colonies. Despite the negative effect of SBV on honey bees, the structure of its particles and mechanism of its genome delivery are unknown. Here we present the crystal structure of SBV virion and show that it contains 60 copies of a minor capsid protein (MiCP) attached to the virion surface. No similar MiCPs have been previously reported in any of the related viruses from the order Picornavirales. The location of the MiCP coding sequence within the SBV genome indicates that the MiCP evolved from a C-terminal extension of a major capsid protein by the introduction of a cleavage site for a virus protease. The exposure of SBV to acidic pH, which the virus likely encounters during cell entry, induces the formation of pores at threefold and fivefold axes of the capsid that are 7 Å and 12 Å in diameter, respectively. This is in contrast to vertebrate picornaviruses, in which the pores along twofold icosahedral symmetry axes are currently considered the most likely sites for genome release. SBV virions lack VP4 subunits that facilitate the genome delivery of many related dicistroviruses and picornaviruses. MiCP subunits induce liposome disruption in vitro, indicating that they are functional analogs of VP4 subunits and enable the virus genome to escape across the endosome membrane into the cell cytoplasm. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3865.map.gz emd_3865.map.gz | 53.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3865-v30.xml emd-3865-v30.xml emd-3865.xml emd-3865.xml | 18 KB 18 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3865_fsc.xml emd_3865_fsc.xml | 18.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3865.png emd_3865.png | 276.6 KB | ||
| Filedesc metadata |  emd-3865.cif.gz emd-3865.cif.gz | 6.3 KB | ||
| その他 |  emd_3865_additional.map.gz emd_3865_additional.map.gz | 61.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3865 http://ftp.pdbj.org/pub/emdb/structures/EMD-3865 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3865 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3865 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3865_validation.pdf.gz emd_3865_validation.pdf.gz | 298.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3865_full_validation.pdf.gz emd_3865_full_validation.pdf.gz | 297.8 KB | 表示 | |
| XML形式データ |  emd_3865_validation.xml.gz emd_3865_validation.xml.gz | 16.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3865 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3865 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3865 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3865 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6egvMC  3863C  3866C  3867C  3881C  5lsfC  5oypC  6egxC  6eh1C  6eiwC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3865.map.gz / 形式: CCP4 / 大きさ: 620.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3865.map.gz / 形式: CCP4 / 大きさ: 620.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.063 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 3865 additional.map
| ファイル | emd_3865_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Sacbrood virus
| 全体 | 名称:  Sacbrood virus (ウイルス) Sacbrood virus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Sacbrood virus
| 超分子 | 名称: Sacbrood virus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: Petrusov, CZ / NCBI-ID: 89463 / 生物種: Sacbrood virus / ウイルスタイプ: VIRION / ウイルス・単離状態: STRAIN / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  |
| ウイルス殻 | Shell ID: 1 / 名称: capsid / 直径: 271.12 Å / T番号(三角分割数): 3 |
-分子 #1: structural protein VP1
| 分子 | 名称: structural protein VP1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sacbrood virus (ウイルス) Sacbrood virus (ウイルス) |
| 分子量 | 理論値: 27.659998 KDa |
| 配列 | 文字列: MDTGAKEDED ETANFSDGVT AMGFQSLDTQ VSIKDILRRP VLLFNHVELD PDYTGFFIPI MPPSRMMQYK SGDKETSFQR LIGRTPQAA IMNLFRFWRG SLRYTIIIHS TDGHPIYVTH VPHTGNRVYG LMKVNNLHEY TKVPIFGCGL TTEMIIPSVN P SICVEVPF ...文字列: MDTGAKEDED ETANFSDGVT AMGFQSLDTQ VSIKDILRRP VLLFNHVELD PDYTGFFIPI MPPSRMMQYK SGDKETSFQR LIGRTPQAA IMNLFRFWRG SLRYTIIIHS TDGHPIYVTH VPHTGNRVYG LMKVNNLHEY TKVPIFGCGL TTEMIIPSVN P SICVEVPF DTENNWAVTF DEDAQRNYSW RDKGDTVTGH LVVTPVVSVY MSVWVEAGDD FEVSNFYGPP SVKTNDWNYA FS DEH UniProtKB: Genome polyprotein |
-分子 #2: structural protein VP2
| 分子 | 名称: structural protein VP2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sacbrood virus (ウイルス) Sacbrood virus (ウイルス) |
| 分子量 | 理論値: 26.952393 KDa |
| 配列 | 文字列: EVPSKESIQG DATQQSSKEE NTIITRDQQQ TVSENIPSTV GDLVIASSEP TQQFRSLTNR WMPINSIRVT VNGKRNDLLA QYYIPEDFL STHAKCAPNT IPFETYVYGK YELEMKFVAN GNKFQCGKVI ISVKFDSYQA DNINTGFQAA LSRPHIMLDL S TNNEGVLK ...文字列: EVPSKESIQG DATQQSSKEE NTIITRDQQQ TVSENIPSTV GDLVIASSEP TQQFRSLTNR WMPINSIRVT VNGKRNDLLA QYYIPEDFL STHAKCAPNT IPFETYVYGK YELEMKFVAN GNKFQCGKVI ISVKFDSYQA DNINTGFQAA LSRPHIMLDL S TNNEGVLK IPFRYHRAFV RNQTHKTATA GVRPGKFASI YVQVLSPLQT GEGGANDMFI RPFYRYTRAE FAGMSYKVPL T UniProtKB: Structural polyprotein |
-分子 #3: structural protein VP3
| 分子 | 名称: structural protein VP3 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sacbrood virus (ウイルス) Sacbrood virus (ウイルス) |
| 分子量 | 理論値: 30.701373 KDa |
| 配列 | 文字列: DKPKDVSSIT IIPKPRLGFP HGKGKSDAVA MRVNPVALTS FQDVSAYPDE PRTTLDIARI WGLRSTFNWG SGDEHGKELF NTVLDPGLR FYDQDYEGQI TPMEYVTGLY NFWSGPIELR FDFVSNAFHT GTVIISAEYN RSSTNTDECQ SHSTYTKTFH L GEQKSVHF ...文字列: DKPKDVSSIT IIPKPRLGFP HGKGKSDAVA MRVNPVALTS FQDVSAYPDE PRTTLDIARI WGLRSTFNWG SGDEHGKELF NTVLDPGLR FYDQDYEGQI TPMEYVTGLY NFWSGPIELR FDFVSNAFHT GTVIISAEYN RSSTNTDECQ SHSTYTKTFH L GEQKSVHF TVPYIYDTVV RRNTASAYLP VTDYDKVDNV SRAQAMGIRA ESKMRVKVRV VNVLRPVAST TSTIEVLVYM RG GKNYALH GLKQSTYWPS NSVVPIDSFP PDGYDP UniProtKB: Genome polyprotein |
-分子 #4: minor capsid protein MiCP
| 分子 | 名称: minor capsid protein MiCP / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Sacbrood virus (ウイルス) Sacbrood virus (ウイルス) |
| 分子量 | 理論値: 3.107398 KDa |
| 配列 | 文字列: DNPHRFLPAN VSNRWNEYSS AYLPRV UniProtKB: Structural protein Vp1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 15 mg/mL | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 5.8 構成要素:
| |||||||||||||||
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: OTHER / 前処理 - 気圧: 101.325 kPa | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 70 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK II | |||||||||||||||
| 詳細 | full particle treated with pH 5.8 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 詳細 | Preliminar grid screening was performed manually on FEI Tecnai F20 (cryo stage). |
| 撮影 | フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / デジタル化 - 画像ごとのフレーム数: 2-16 / 撮影したグリッド数: 1 / 平均露光時間: 1.0 sec. / 平均電子線量: 21.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 倍率(補正後): 74325 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 47.1 / 当てはまり具合の基準: R-factor |
|---|---|
| 得られたモデル |  PDB-6egv: |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)