+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3401 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of CSN-SCF-N8 complex | |||||||||
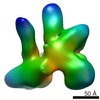
 マップデータ マップデータ | reconstruction of CSN5H138A-N8_SCF/Skp2 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | COP9 signalosome / Cullin-RING ligase / SCF / deneddylation | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / COP9 signalosome assembly / trophectodermal cell proliferation / macrophage migration inhibitory factor binding / positive regulation of protein polyubiquitination / regulation of IRE1-mediated unfolded protein response / Parkin-FBXW7-Cul1 ubiquitin ligase complex / exosomal secretion / deNEDDylase activity / GTPase inhibitor activity ...regulation of DNA damage response, signal transduction by p53 class mediator resulting in transcription of p21 class mediator / COP9 signalosome assembly / trophectodermal cell proliferation / macrophage migration inhibitory factor binding / positive regulation of protein polyubiquitination / regulation of IRE1-mediated unfolded protein response / Parkin-FBXW7-Cul1 ubiquitin ligase complex / exosomal secretion / deNEDDylase activity / GTPase inhibitor activity / F-box domain binding / regulation of protein neddylation / eukaryotic translation initiation factor 3 complex / protein deneddylation / Aberrant regulation of mitotic exit in cancer due to RB1 defects / PcG protein complex / cullin-RING-type E3 NEDD8 transferase / cellular response to chemical stress / NEDD8 transferase activity / mitotic cell cycle phase transition / cullin-RING ubiquitin ligase complex / COP9 signalosome / Cul7-RING ubiquitin ligase complex / ubiquitin-dependent protein catabolic process via the C-end degron rule pathway / activation of NF-kappaB-inducing kinase activity / positive regulation of ubiquitin protein ligase activity / Loss of Function of FBXW7 in Cancer and NOTCH1 Signaling / regulation of proteolysis / maintenance of protein location in nucleus / positive regulation of protein autoubiquitination / protein neddylation / metal-dependent deubiquitinase activity / cyclin-dependent protein serine/threonine kinase activator activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / NEDD8 ligase activity / RHOBTB1 GTPase cycle / Cul5-RING ubiquitin ligase complex / negative regulation of response to oxidative stress / positive regulation of intracellular estrogen receptor signaling pathway / ubiquitin-ubiquitin ligase activity / Cul4A-RING E3 ubiquitin ligase complex / SCF ubiquitin ligase complex / inner cell mass cell proliferation / Cul2-RING ubiquitin ligase complex / negative regulation of type I interferon production / Cul4B-RING E3 ubiquitin ligase complex / ubiquitin ligase complex scaffold activity / SCF-dependent proteasomal ubiquitin-dependent protein catabolic process / Cul3-RING ubiquitin ligase complex / protein deubiquitination / Prolactin receptor signaling / skeletal muscle cell differentiation / TGF-beta receptor signaling activates SMADs / protein monoubiquitination / cullin family protein binding / protein K63-linked ubiquitination / regulation of JNK cascade / ubiquitin-like ligase-substrate adaptor activity / positive regulation of double-strand break repair via homologous recombination / response to light stimulus / cyclin-dependent protein kinase holoenzyme complex / anatomical structure morphogenesis / protein K48-linked ubiquitination / Nuclear events stimulated by ALK signaling in cancer / JNK cascade / positive regulation of TORC1 signaling / regulation of mitotic cell cycle / T cell activation / translation initiation factor activity / Regulation of BACH1 activity / intrinsic apoptotic signaling pathway / MAP3K8 (TPL2)-dependent MAPK1/3 activation / post-translational protein modification / ubiquitin binding / SCF-beta-TrCP mediated degradation of Emi1 / NIK-->noncanonical NF-kB signaling / molecular function activator activity / Vpu mediated degradation of CD4 / Degradation of DVL / Dectin-1 mediated noncanonical NF-kB signaling / Recognition of DNA damage by PCNA-containing replication complex / Degradation of GLI1 by the proteasome / Activation of NF-kappaB in B cells / animal organ morphogenesis / Negative regulation of NOTCH4 signaling / cellular response to amino acid stimulus / Iron uptake and transport / GSK3B and BTRC:CUL1-mediated-degradation of NFE2L2 / Vif-mediated degradation of APOBEC3G / Hedgehog 'on' state / DNA Damage Recognition in GG-NER / Degradation of GLI2 by the proteasome / GLI3 is processed to GLI3R by the proteasome / FBXL7 down-regulates AURKA during mitotic entry and in early mitosis / APC/C:Cdh1 mediated degradation of Cdc20 and other APC/C:Cdh1 targeted proteins in late mitosis/early G1 / RING-type E3 ubiquitin transferase / Degradation of beta-catenin by the destruction complex / protein modification process / negative regulation of canonical Wnt signaling pathway / Oxygen-dependent proline hydroxylation of Hypoxia-inducible Factor Alpha 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.2 Å | |||||||||
 データ登録者 データ登録者 | Mosadeghi R / Reichermeier KM / Winkler M / Schreiber A / Reitsma JM / Zhang Y / Stengel F / Cao J / Kim M / Sweredoski MJ ...Mosadeghi R / Reichermeier KM / Winkler M / Schreiber A / Reitsma JM / Zhang Y / Stengel F / Cao J / Kim M / Sweredoski MJ / Hess S / Leitner A / Aebersold R / Peter M / Deshaies RJ / Enchev RI | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2016 ジャーナル: Elife / 年: 2016タイトル: Structural and kinetic analysis of the COP9-Signalosome activation and the cullin-RING ubiquitin ligase deneddylation cycle. 著者: Ruzbeh Mosadeghi / Kurt M Reichermeier / Martin Winkler / Anne Schreiber / Justin M Reitsma / Yaru Zhang / Florian Stengel / Junyue Cao / Minsoo Kim / Michael J Sweredoski / Sonja Hess / ...著者: Ruzbeh Mosadeghi / Kurt M Reichermeier / Martin Winkler / Anne Schreiber / Justin M Reitsma / Yaru Zhang / Florian Stengel / Junyue Cao / Minsoo Kim / Michael J Sweredoski / Sonja Hess / Alexander Leitner / Ruedi Aebersold / Matthias Peter / Raymond J Deshaies / Radoslav I Enchev /   要旨: The COP9-Signalosome (CSN) regulates cullin-RING ubiquitin ligase (CRL) activity and assembly by cleaving Nedd8 from cullins. Free CSN is autoinhibited, and it remains unclear how it becomes ...The COP9-Signalosome (CSN) regulates cullin-RING ubiquitin ligase (CRL) activity and assembly by cleaving Nedd8 from cullins. Free CSN is autoinhibited, and it remains unclear how it becomes activated. We combine structural and kinetic analyses to identify mechanisms that contribute to CSN activation and Nedd8 deconjugation. Both CSN and neddylated substrate undergo large conformational changes upon binding, with important roles played by the N-terminal domains of Csn2 and Csn4 and the RING domain of Rbx1 in enabling formation of a high affinity, fully active complex. The RING domain is crucial for deneddylation, and works in part through conformational changes involving insert-2 of Csn6. Nedd8 deconjugation and re-engagement of the active site zinc by the autoinhibitory Csn5 glutamate-104 diminish affinity for Cul1/Rbx1 by ~100-fold, resulting in its rapid ejection from the active site. Together, these mechanisms enable a dynamic deneddylation-disassembly cycle that promotes rapid remodeling of the cellular CRL network. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3401.map.gz emd_3401.map.gz | 37.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3401-v30.xml emd-3401-v30.xml emd-3401.xml emd-3401.xml | 29.3 KB 29.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-3401_image.tif EMD-3401_image.tif | 732.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3401 http://ftp.pdbj.org/pub/emdb/structures/EMD-3401 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3401 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3401 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3401_validation.pdf.gz emd_3401_validation.pdf.gz | 213.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3401_full_validation.pdf.gz emd_3401_full_validation.pdf.gz | 212.6 KB | 表示 | |
| XML形式データ |  emd_3401_validation.xml.gz emd_3401_validation.xml.gz | 6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3401 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3401 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3401 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3401 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3401.map.gz / 形式: CCP4 / 大きさ: 39.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3401.map.gz / 形式: CCP4 / 大きさ: 39.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | reconstruction of CSN5H138A-N8_SCF/Skp2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.74 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : CSN5H138A-N8_SCF/Skp2
+超分子 #1000: CSN5H138A-N8_SCF/Skp2
+分子 #1: Csn1
+分子 #2: Csn2
+分子 #3: Csn3
+分子 #4: Csn4
+分子 #5: Csn5
+分子 #6: Csn6
+分子 #7: Csn7b
+分子 #8: Csn8
+分子 #9: Cul1
+分子 #10: Rbx1
+分子 #11: Nedd8
+分子 #12: Skp1
+分子 #13: Skp2
+分子 #14: Cks1
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 15 mM HEPES, pH 7.6, 100 mM NaCl, 0.5 mM DTT |
| グリッド | 詳細: Quantifoil grids (R1.2/1.3 Cu 400 mesh), freshly coated with an extra layer of thin carbon and glow-discharged for 2 min at 50 mA and 0.2 mbar vacuum |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 120 K / 装置: HOMEMADE PLUNGER 手法: the sample was diluted to 0.1 mg/ml and 2 ul were applied to Quantifoil grids (R1.2/1.3 Cu 400 mesh), freshly coated with an extra layer of thin carbon and glow-discharged for 2 min at 50 mA ...手法: the sample was diluted to 0.1 mg/ml and 2 ul were applied to Quantifoil grids (R1.2/1.3 Cu 400 mesh), freshly coated with an extra layer of thin carbon and glow-discharged for 2 min at 50 mA and 0.2 mbar vacuum. The grids were manually blotted to produce a thin sample film and plunge-frozen into liquid ethane |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 90 K / 最高: 110 K / 平均: 100 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected manually at a nominal magnification of 100,000x |
| 日付 | 2015年5月12日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 平均電子線量: 25 e/Å2 詳細: Microgrpahs were the average of six frames recorded by the direct electron detector. All micrographs were initially visually inspected and only those with appropriate ice thickness as well as ...詳細: Microgrpahs were the average of six frames recorded by the direct electron detector. All micrographs were initially visually inspected and only those with appropriate ice thickness as well as Thon rings in their power spectra showing regularity and extending to 6 A or beyond were used for subsequent analysis. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 80460 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 60000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | CTF-estimation and subsequent correction were performed using RELION (Scheres, 2012) and CTFFIND3 (Mindell and Grigorieff, 2003). All micrographs were initially visually inspected and only those with appropriate ice thickness as well as Thon rings in their power spectra showing regularity and extending to 6 A or beyond were used for subsequent analysis. In order to generate 2D references for automated particle selection, ~ 4,000 single particles were manually picked and subjected to 2D classification in RELION. Six well-defined 2D class averages were selected, low-pass filtered to 35 A to prevent reference bias, and used as references. Approximately 150,000 single particles were automatically selected and subjected to reference-free 2D and 3D classification, in order to de-select the particles, which resulted in poorly defined or noisy averages. Approximately half of these single particles resulted in a well-defined 3D class average, which resembled the previously published negative stain EM map of the same complex (Enchev et al., 2012). This dataset was subject to 3D auto-refinement in RELION, using a version low-pass filtered to 50 A as an initial reference. The converged map was further post-processed in RELION, using MTF-correction, FSC-weighting and a soft spherical mask with a 5-pixel fall-off. |
|---|---|
| CTF補正 | 詳細: micrographs |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 7.2 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: relion, CTFFIND3 / 使用した粒子像数: 75000 |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E |
|---|---|
| ソフトウェア | 名称: Chmiera |
| 詳細 | some domains were fitted separately to account for conformational rearrangements, see publication for details |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: overlap |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F / Chain - #6 - Chain ID: H |
|---|---|
| ソフトウェア | 名称: Chmiera |
| 詳細 | Csn7b was modeled using Csn7a as a template on the Phyre2 server; some domains were fitted separately to account for conformational rearrangements, see publication for details |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: overlap |
-原子モデル構築 3
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B |
|---|---|
| ソフトウェア | 名称: Chmiera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 当てはまり具合の基準: overlap |
 ムービー
ムービー コントローラー
コントローラー